Command Palette
Search for a command to run...
CVPR 最佳学生论文!1 千万张图像、跨越 45 万+物种的超大数据集,多模态模型 BioCLIP 实现零样本学习

不同于传统学术领域对期刊发表的重视,计算机界,尤其是机器学习、计算机视觉、人工智能等领域,顶级会议才是王道,无数「热门研究方向」、「创新方法」都将从这里流出。
作为计算机视觉乃至人工智能领域最具学术影响力的三大顶会之一,今年的国际计算机视觉与模式识别会议 (CVPR) 从会议规模到接受论文数量,都刷新了此前记录。

根据 CVPR 官方的最新公告,CVPR 2024 已经成为该会议历史上规模最大、参与人数最多的一届,截止 6 月 19 日,现场参会人数已超过 1.2w 人。

此外,作为领先的计算机视觉盛会,CVPR 每年都会录用当前视觉领域的最新研究,在今年提交的 11,532 份有效论文中,2,719 篇论文被接收,与 CVPR 2023 相比,接收论文的数量提高 20.6%,而录用率却降低 2.2% 。这些数据表明,CVPR 2024 的热度、竞争激烈程度以及获奖论文的含金量均有所提升。

北京时间 6 月 20 日凌晨,CVPR 2024 正式公布了本届会议的最佳论文等奖项。据统计,本次共有 10 篇论文获奖,其中最佳论文 2 篇,最佳学生论文 2 篇,最佳论文提名 2 篇和最佳学生论文提名 4 篇。

其中,「BIoCLIP: A Vision Foundation Model for the Tree of Life」被评为最佳学生论文。对此,麻省理工学院计算机科学与人工智能实验室助理教授 Sara Beery 更是评价其作者和团队为「当之无愧」的获奖者,论文第一作者 Samuel Stevens 第一时间在平台上表示了感谢。
HyperAI 超神经将从数据集、模型架构、模型性能等方面,全面解读「BIoCLIP: A Vision Foundation Model for the Tree of Life」,并为大家汇总整理了一作 Sam Stevens 的其他成果。
论文下载地址:
https://arxiv.org/pdf/2311.18803
创建最大、最多样化的生物图像数据集
目前最大的机器学习生物图像数据集是 iNat21,包含 270 万张图像,覆盖 1 万个物种。尽管,与 ImageNet-1k 等通用领域数据集相比,iNat21 的分类广度有极大提升,但对于生物学而言,1 万个物种仍是凤毛麟角。国际自然保护联盟 (IUCN) 在 2022 年就报告了超过 200 万个已知物种,仅鸟类和爬行动物就分别超过 1 万个物种。
针对生物图像数据集物种类别限制的问题,研究人员构建了一个包含 1,000 万张图像的数据集 TreeOfLife-10M,跨越 450K+ 物种,在数据集规模和物种多样性方面完成了革命性突破。
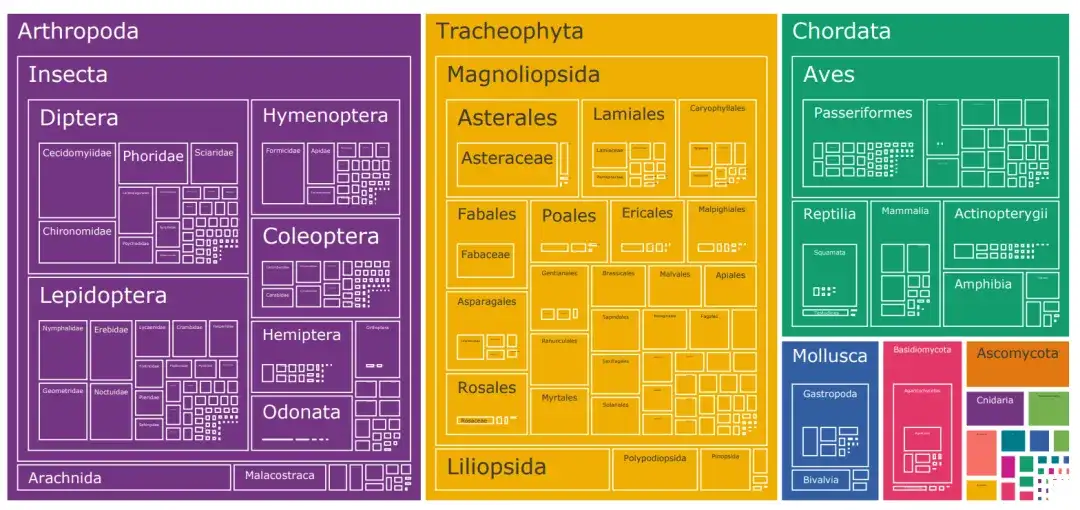
该数据集结合了 iNaturalist 、 BIOSCAN-1M 和生命百科全书 (Encyclopedia of Life, EOL) 中的生物图像。
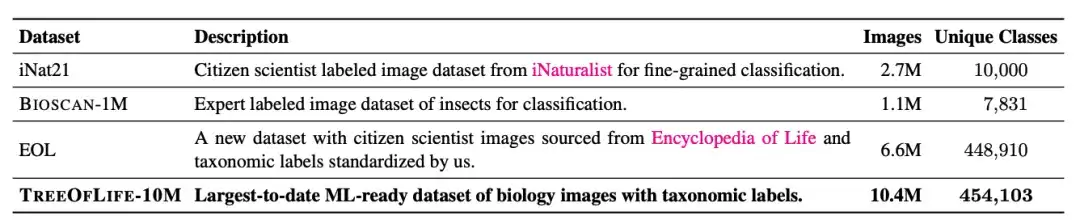
除了 iNat21 所覆盖的 1 万个物种类别外,研究人员从 EOL 下载了 660 万张图像,将 TreeOfLife-10M 扩展到覆盖额外的 44 万种分类群。同时,为了帮助基础模型学习昆虫的极细粒度视觉表征,研究人员还纳入了 BIOSCAN-1M,这是一个包含 100 万张实验室昆虫图像的最新数据集,覆盖 494 个不同的科、 7,831 种物种分类。
TreeOfLife-10M 下载地址:
https://go.hyper.ai/Gliol
多模态模型 BioCLIP:基于 CLIP 提升泛化能力
与通用任务相比,生物学计算机视觉的标注空间 (label space) 更加丰富,不仅分类标注数量庞大,且标注在层级分类系统中相互连接,这无疑为训练高物种覆盖率、强泛化能力的基础模型带来了巨大挑战。
借助生物学数百年研究所积累的经验,研究人员认为,如果基础模型能够成功编码标注空间的结构,那么即便没有见过某个特定物种,模型就可能已经能够识别其所对应的属 (genus) 或科 (family) 并给出相应的表征,而这种层级表征 (hierarchical representation) 将有助于实现新分类群的少样本、甚至零样本学习。
基于此,研究人员选择了 OpenAI 开发的多模态模型架构 CLIP,并利用 CLIP 的多模态对比学习目标在 TREEOFLIFE-10M 上持续进行预训练。
具体而言,CLIP 对视觉编码器和文本编码器这两种单模态嵌入模型进行训练,从而最大化正对 (positive pairs) 之间的特征相似性,最小化负对 (negative pairs) 之间的特征相似性,其中正对来自训练数据,负对是 batch 中所有其他可能的配对。

此外,CLIP 的一个重要优势便是其文本编码器接受自由形式的文本,恰恰能够应对生物学领域多样化的类名格式问题。针对该研究中的文本形式,研究人员主要考虑了:
* 分类名称 (Taxonomic name):标准的七级生物分类从高到低依次为界 (Kingdom) 、门 (Phylum) 、纲 (Class) 、目 (Order) 、科 (Family) 、属 (Genus) 、种 (Species) 。对于每个物种,通过将从根到叶的所有标注连接成一个字符串来「展平 (flatten)」分类系统,这个字符串就是分类名称。
* 学名 (Scientific name):由属和种组成。
* 通用名 (Common name.):分类名称通常是拉丁文,而在通用的图像-文本预训练数据集中不常见。相反,诸如「黑嘴喜鹊 (black-billed magpie)」这样的通用名称更为普遍。需要注意的是,通用名称与分类群之间可能并不是一对一映射关系,一个物种可能有多个通用名称,或者同一个通用名称可能指多个物种。
在实际应用中,可能只有一种类型的标注输入,为了在推理时提高灵活性,研究人员提出了一种混合文本类型训练策略,即在每个训练步骤中,将每个输入图像与从所有可用文本类型中随机抽取的文本配对。实验证明,这种训练策略不仅保持了分类名称的泛化优势,还在推理时提供了更多灵活性。
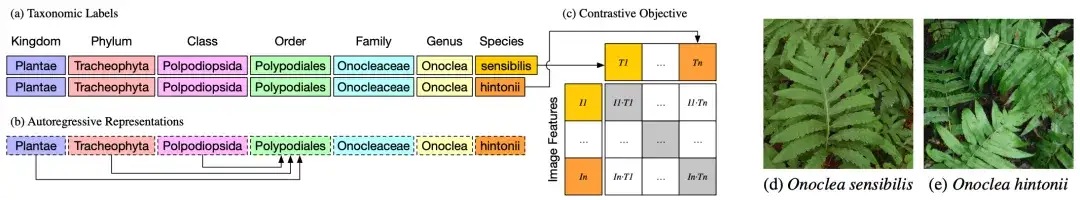
如上图 a 所示,两种不同植物 Onoclea sensibilis (d) 和 Onoclea hintonii (e) 的分类群或分类标签,除物种 (species) 外,完全相同。
上图 b 所示,文本编码器是一个自回归语言模型,能够自然编码 taxonomy 的分层表征 (hierarchical representations),其中的阶次表征 (order representation) Polypodiales 只能依赖于更高的阶次,从 Kingdom 、 Phylum 和 Class tokens 中吸收信息。这些分类标注 (taxonomic labels) 的分层表征被输入到标准的对比预训练目标中,并与图像表征 (d) 和 (e) 相匹配。
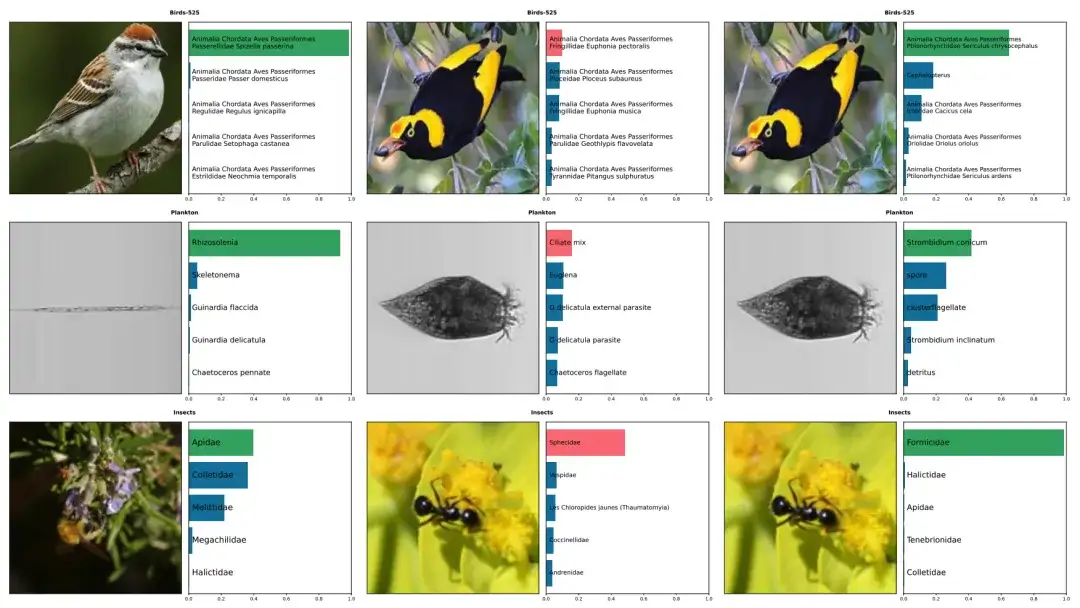
上图为 BioCLIP 和 CLIP 对鸟类 525 、浮游生物、昆虫等五种的预测示例,正确标注为绿色,错误标注为红色。左侧一列为 BioCLIP 的正确预测。中间及右侧是 CLIP 错误标注但 BioCLIP 正确标注的图像。
BioCLIP 在零样本和少样本任务上表现出色
研究人员将 BioCLIP 与通用视觉模型进行了比较。结果显示,BioCLIP 在零样本和少样本任务中均表现出色,并且显著优于 CLIP 和 OpenCLIP,在零样本和少样本任务中的平均绝对改善 (average absolute improvement) 分别为 17% 和 16% 。内在分析进一步表明,BioCLIP 学习到了更细粒度的符合生命树的层次表示,解释了其卓越的泛化能力。
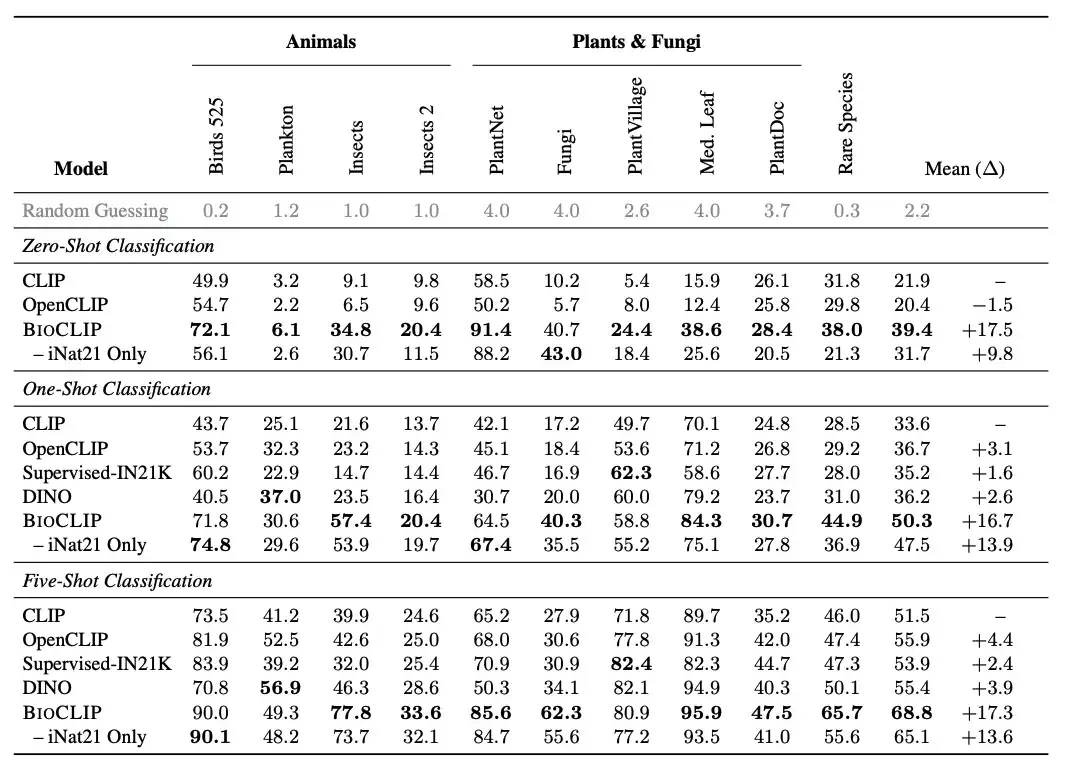
具体而言,研究人员引入了一项新的评估任务「稀有物种 (RARE SPECIES)」,收集了世界自然保护联盟红色名录中的约 25K 个物种,这些物种被分类为近危、易危、濒危、极危或野外灭绝。研究人员在 EOL 数据集中,选取了至少有 30 幅图像的 400 个此类物种,然后将它们从 TreeOfLife-10M 中移除,创建了一个全新的稀有物种测试集,每个物种有 30 幅图像。
对比结果如上图所示,BioCLIP 在零样本分类中明显优于基线 CLIP 模型以及 iNat21 训练的 CLIP 模型,尤其是在未见过的分类上(见 Rare Species 列)。
硕果累累,探索最佳 BioCLlP 背后的科研之道
「BioCLlP: A Vision Foundation Model for the Tree of Life」由美国俄亥俄州立大学、微软研究院、加州大学欧文分校、伦斯勒理工学院共同发布,论文的第一作者 Samuel Stevens 博士和通讯作者 Jiaman Wu 均来自俄亥俄州立大学。
尽管 Samuel Stevens 自谦地在其个人网页上描述自己「不是一个非常严肃对待自己的人」,但从他近几年丰硕的科研成果和不懈努力来看,他显然是一个严谨对待科研事业的人。
据了解,Samuel Stevens 从 2017 年开始,一直从事计算机方向的工作,多模态模型 BioCLlP 是他在 2023 年 12 月发布的一项研究成果,并于 2024 年 2 月被 CVPR 2024 接收。
事实上,类似 BioCLlP 等 computer vision 方向的工作只是他的研究方向之一,他的兴趣很广泛,也曾在 AI for crypto 、 various LLM projects 等领域展开了一系列研究。
例如,他曾参与的「MMMU:A Massive Multi-discipline Multimodal Understanding and Reasoning Benchmark for Expert AGI」,提出了一个新基准 MMMU (大规模多任务语言理解),作为目前业界最有影响力的大模型测评基准之一,MMMU 侧重于利用特定领域(科学、健康与医学、人文等)的知识进行高级感知和推理,要求多模态模型能够执行与专家面临的类似任务。
研究人员用其对 14 个开源 LMM 和专有 GPT-4V (ision) 进行了评估,发现即使是先进的 GPT-4V 也只达到了 56% 的准确率,这表明模型还有很大的改进空间。对此,研究人员表示,希望该基准能激励社区建立下一代多模态基础模型,以实现专家级人工通用智能。
MMMU: https://mmmu-benchmark.github.io
当然,他对科研的热情和开放态度也是他取得成功的关键要素。昨日,BioCLlP 被评为最佳学生论文的消息刚出来,Samuel Stevens 博士第一时间通过社交平台向外界表达了他的观点:「如果您想聊聊动物的计算机视觉、多模态基础模型或 AI for Science,请私信我!」

值得一提的是,Samuel Stevens 博士不仅在科研路上砥砺前行,亦不忘提携后进。他的个人网页上也分享着对初学者的建议:「如果你想开始机器学习和人工智能。或许可以从 Coursera 的机器学习课程和 Andrej Karpathy 的 Neural Networks : Zero to Hero 开始。这两门课程的质量都非常高,与其他免费资源相比,应该会提供很多价值。」
参考资料:
1. https://samuelstevens.me/#news
最后推荐一个活动!
扫码即可报名参与「Meet AI Compiler」技术沙龙第 5 期线下聚会↓









