온라인 튜토리얼 | 컴퓨팅 파워 비용 급락! Apple, 스트림 매칭 기반 단백질 폴딩 모델인 Ml-simplefold 출시

2025년 9월, Apple은 경량 단백질 폴딩 예측 AI 모델인 Ml-simplefold를 출시했습니다. 흐름 매칭 기반 최초의 단백질 폴딩 모델로서, CAMEO22 및 CASP14와 같은 권위 있는 벤치마크에서 검증되었습니다.SimpleFold는 계산 비용을 줄인 후에도 AlphaFold2 및 RoseTTAFold2와 같은 최고 모델과 비슷한 성능을 보여줍니다.동시에, 더 작은 버전인 SimpleFold-100M 역시 경쟁력 있는 성능을 보입니다.
기존의 단백질 접힘 모델은 뛰어난 정확도를 제공하지만, 수많은 도메인별 아키텍처 설계와 수작업으로 개발된 특징에 의존합니다. 이러한 모델에서 흔히 사용되는 삼각형 업데이트 모듈, 명시적인 쌍 표현 메커니즘, 그리고 여러 학습 목표는 학습에 많은 계산량을 요구합니다. 더욱이, 아키텍처와 하드웨어의 확장성이 부족하여 다양한 구조 생성이나 앙상블 예측을 지원하기 어렵습니다.
기존 방법의 공통적인 결함을 목표로 하여,SimpleFold는 단백질 접힘 모델이 복잡한 아키텍처에 의존하는 것을 깨고 순수하게 Transformer에 기반한 일반 생성 프레임워크를 제안합니다.
* Flow Matching 기술을 기반으로 다중 시퀀스 정렬(MSA)과 같은 복잡한 모듈을 건너뛰고 무작위 노이즈에서 단백질의 3차원 구조를 직접 생성하여 계산 비용을 크게 절감합니다.
* 삼각형 업데이트 및 쌍 표현과 같은 특정 모듈을 버리고 표준 변환기만 사용하며 적응형 계층을 통해 구조 인식을 강화하는 일반 아키텍처를 채택합니다.
* 단백질 3차원 구조를 생성할 때 모델의 유연성과 물리적 일관성을 높이기 위해 구조적 제약 조건이 추가되었습니다. 9백만 개의 구조 데이터로 학습된 3B 매개변수 모델은 일반 소비자용 하드웨어에서 원활하게 실행되어 계산량을 크게 줄일 수 있습니다.
"Ml-simplefold: 경량 단백질 접힘 예측 AI 모델"이 HyperAI Hyperneuron 웹사이트(hyper.ai)의 "튜토리얼" 섹션에서 이용 가능합니다. 클릭 한 번으로 실행하여 최신 단백질 생성 도구를 경험해 보세요.
튜토리얼 링크:
데모 실행
- 브라우저에 URL hyper.ai를 입력하세요. 홈페이지에 접속한 후 "튜토리얼" 페이지를 클릭하고 "흐름 매칭 기반 단백질 접힘 모델"을 선택한 후 "이 튜토리얼을 온라인으로 실행"을 클릭하세요.
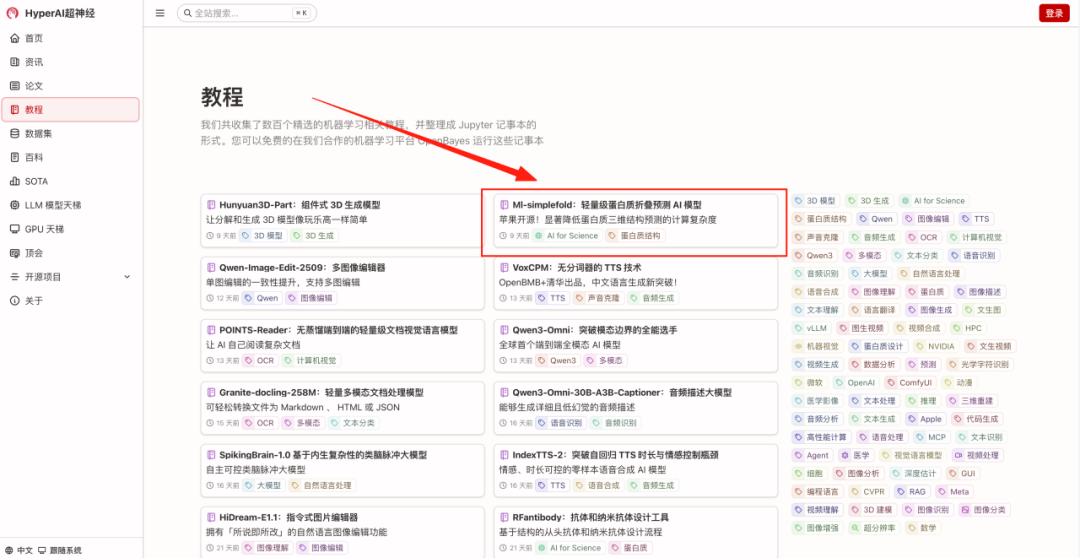
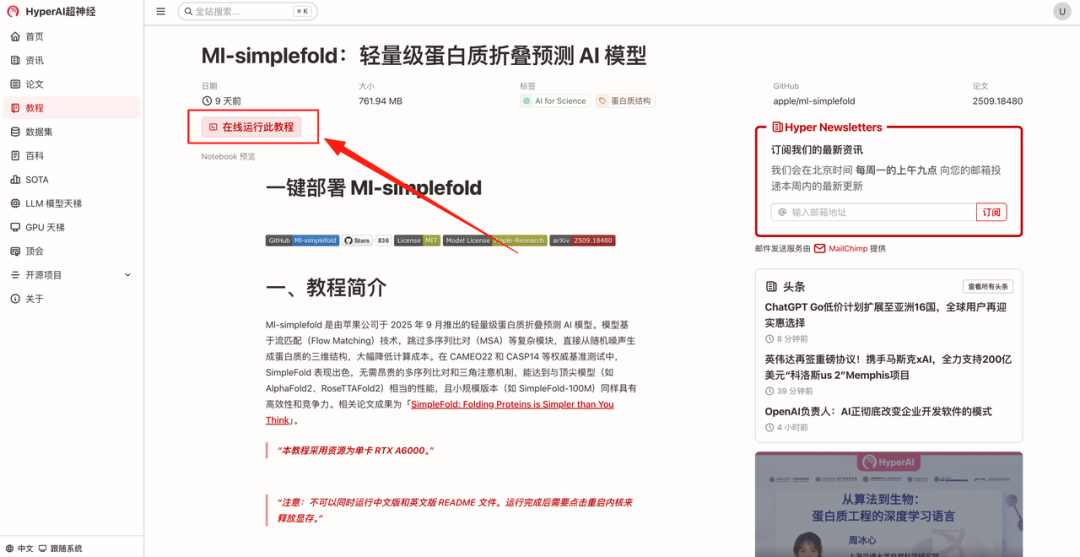
- 페이지가 이동한 후 오른쪽 상단의 "복제"를 클릭하여 튜토리얼을 자신의 컨테이너에 복제하세요.
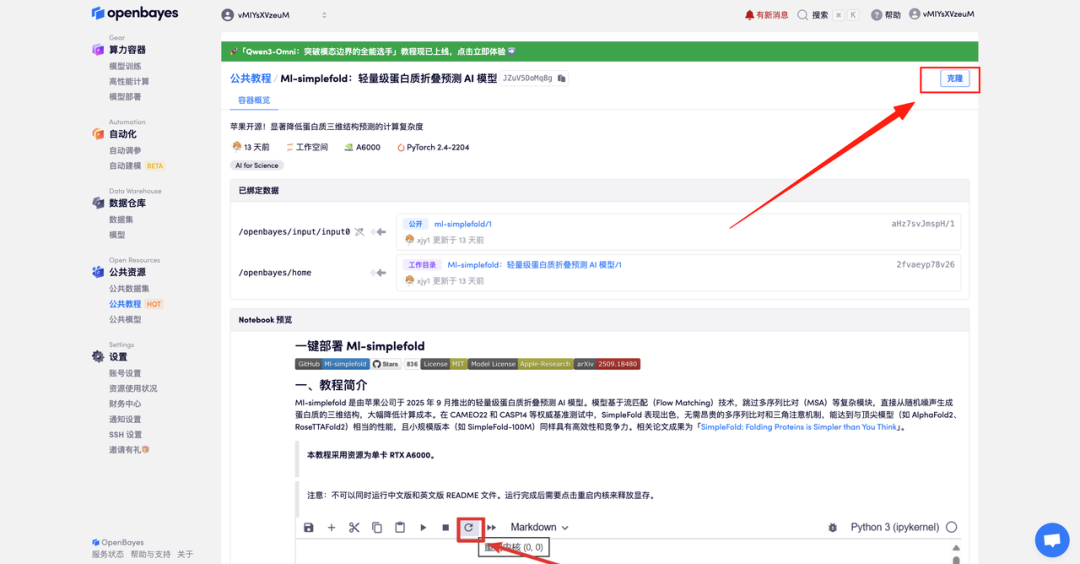
- "NVIDIA RTX A6000 48GB" 및 "PyTorch" 이미지를 선택하고 "계속"을 클릭하세요. OpenBayes 플랫폼은 종량제 또는 일/주/월 요금제 네 가지 결제 옵션을 제공합니다. 신규 사용자는 아래 초대 링크를 통해 등록하시면 RTX 4090 4시간과 CPU 5시간을 무료로 이용하실 수 있습니다!
HyperAI 독점 초대 링크(복사하여 브라우저에서 열기):
https://openbayes.com/console/signup?r=Ada0322_NR0n
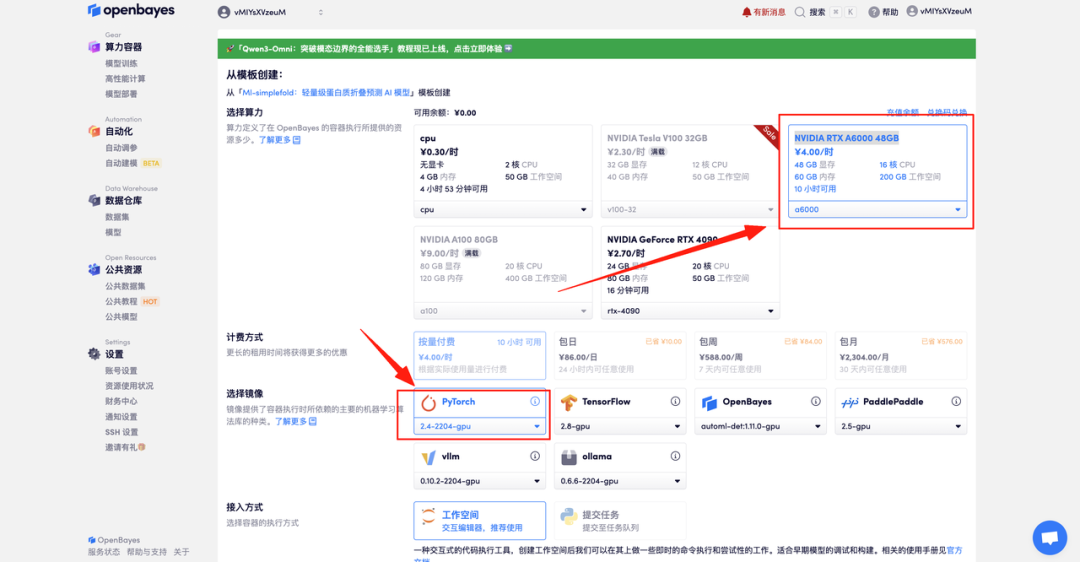
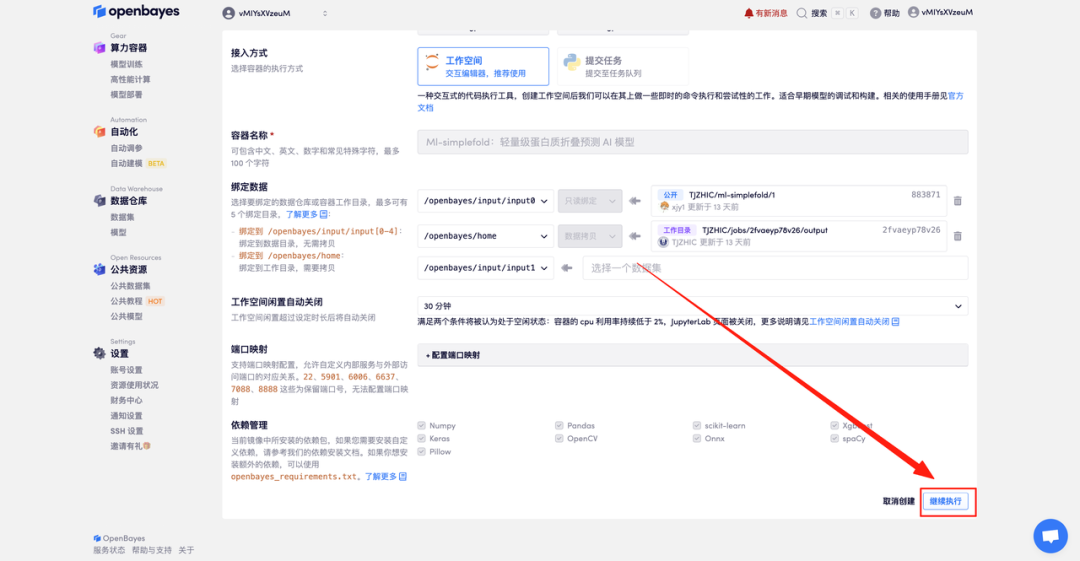
4. 리소스가 할당될 때까지 기다리세요. 첫 번째 복제 프로세스는 약 2분 정도 소요됩니다. 상태가 "실행 중"으로 변경되면 "작업 공간 열기"를 클릭하여 데모 페이지로 이동하세요.
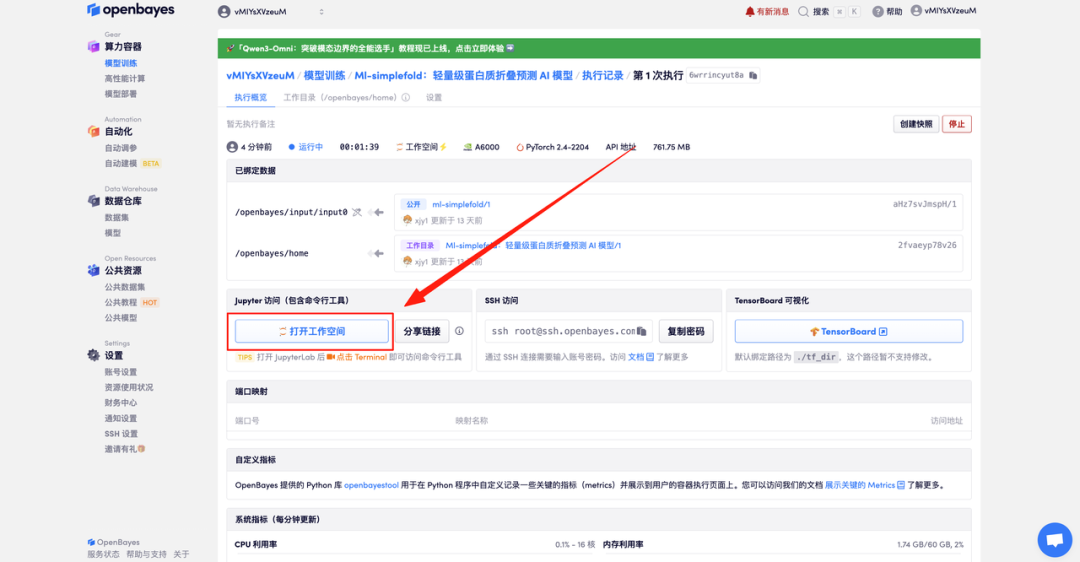
효과 시연
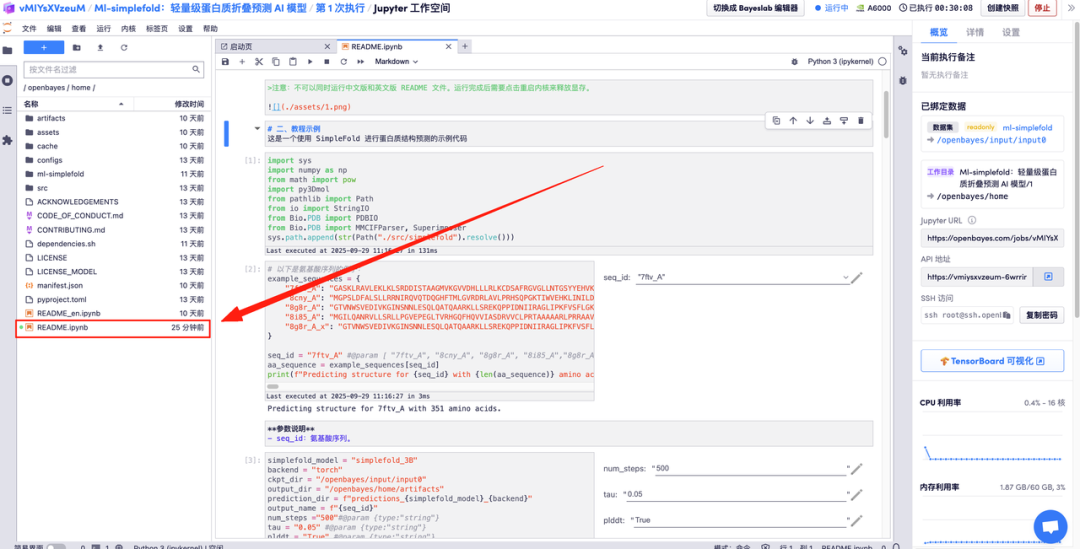
다음은 Ml-simplefold 사용 페이지입니다. "README"를 클릭하면 생성 인터페이스로 이동합니다.
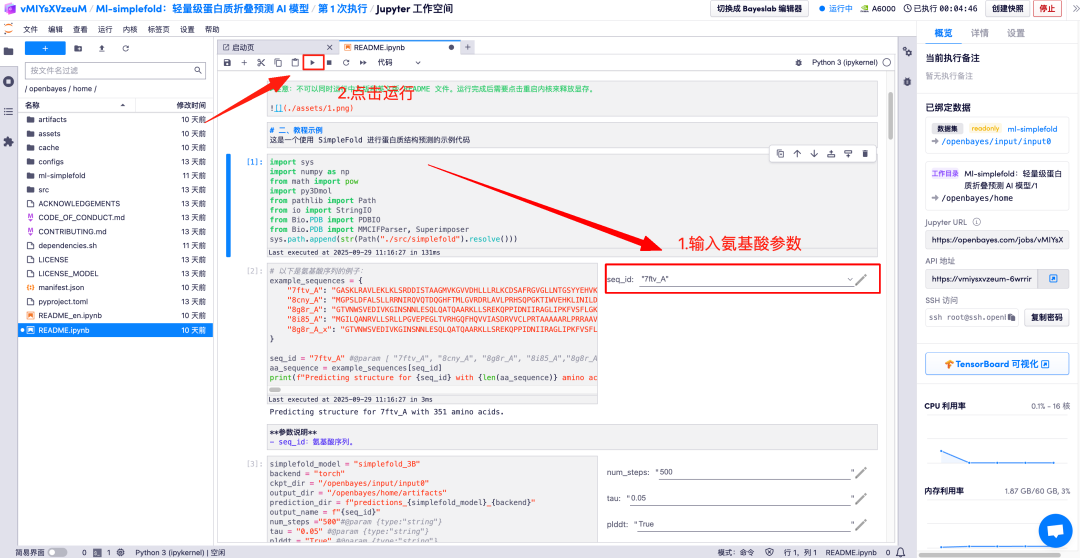
해당 영역에 아미노산 매개변수 설명을 입력하고 실행을 클릭하면 예측된 단백질 파일, 전체 원자 구조 및 3D 시각화 이미지가 GT 구조와 병합됩니다.
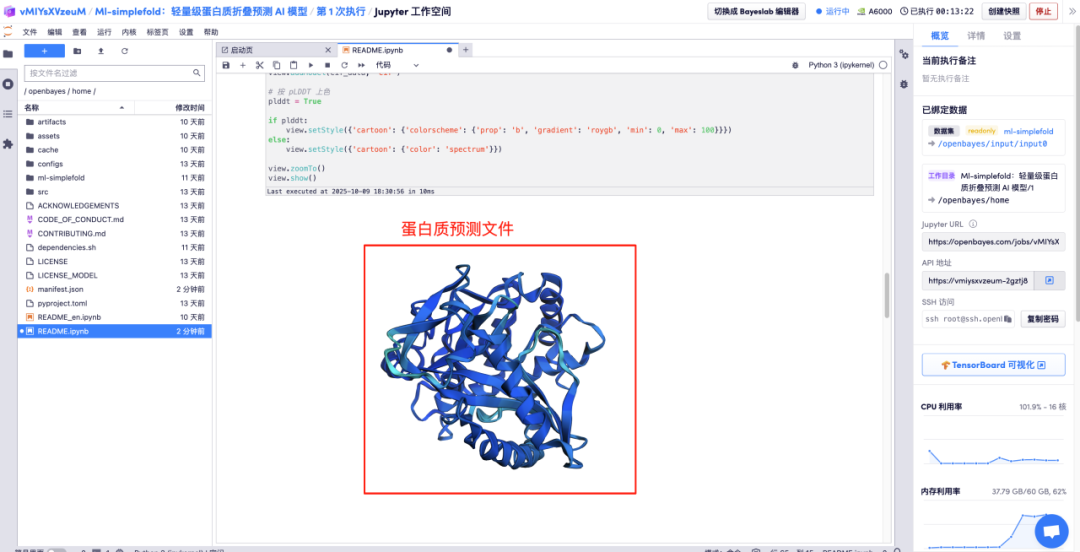
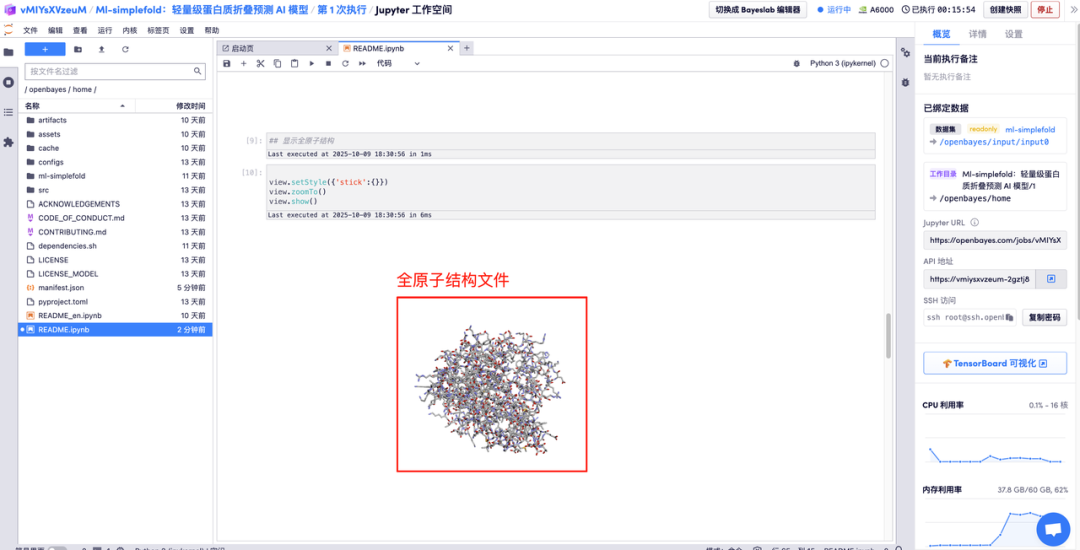
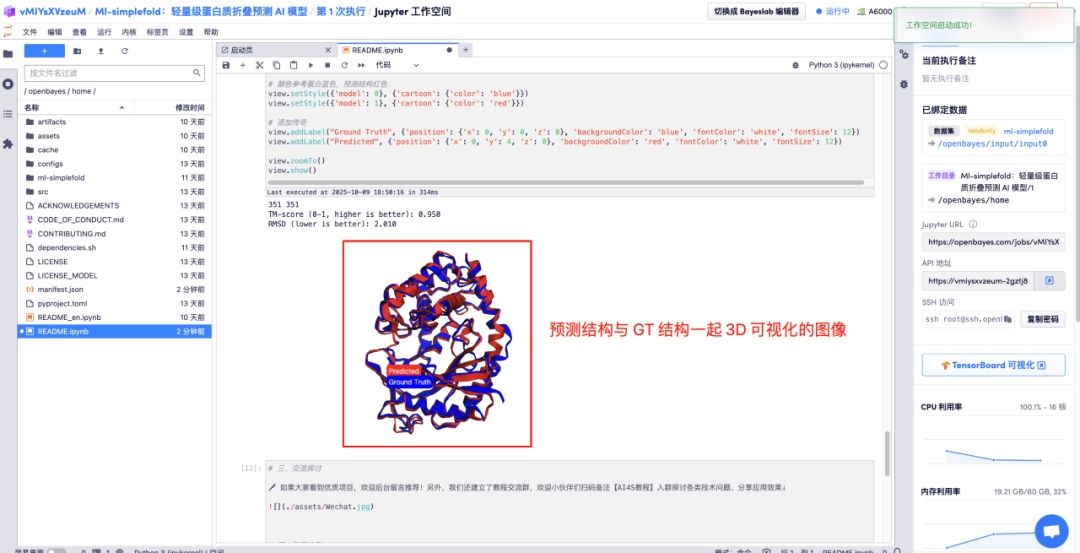
위는 HyperAI가 이번에 추천하는 튜토리얼입니다. 누구나 와서 체험해 보세요!
튜토리얼 링크: