Command Palette
Search for a command to run...
홍콩 중국 대학의 연구진을 비롯한 여러 연구진은 유전자 발현 데이터를 세포 형태 이미지와 연결하여 전사체 유도 확산 모델을 개발해 표현형 약물 개발을 가속화했습니다.
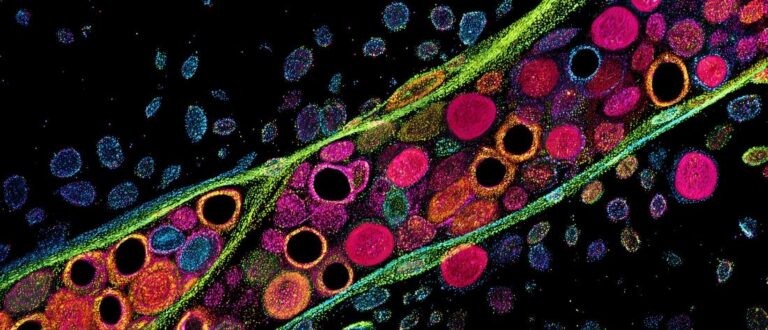
세포 형태학은 단일 세포 생물학의 핵심 연구 분야입니다. 이 연구의 가치는 고처리량 이미지 분석을 통해 유전적 또는 약물 교란에 따른 세포 형태의 역동적인 변화를 체계적으로 분석하는 데 있습니다. 이 연구는 화합물의 작용 기전(MOA) 예측의 정확도를 크게 향상시킬 뿐만 아니라 화합물의 생리활성 평가 정확도도 향상시킵니다. 궁극적으로 후보 화합물 스크리닝 및 작용 기전 검증과 같은 표현형 신약 개발의 핵심 단계에 대한 데이터 지원을 제공하여 R&D 프로세스를 효과적으로 가속화합니다.
그러나 유전적 또는 약물 교란에 따른 세포 형태 변화를 관찰하고 분석하는 것은 간단한 작업이 아닙니다. 스크리닝할 수 있는 화합물의 수는 수백만 개가 넘고, 편집할 수 있는 유전자의 수도 수만 개에 달합니다. 전통적인 실험 방법을 사용하여 각 화합물을 개별적으로 검증하는 것은 매우 비효율적일 뿐만 아니라 많은 시간과 비용을 초래합니다. 세포 형태 예측을 위해 다양한 계산 방법이 제안되고 적용되었지만, 그 정확도와 충실도는 여전히 실제 연구의 요구를 충족하지 못합니다.
구체적으로,기존 방법의 한계는 주로 두 가지 측면에서 드러난다.첫째, IMPA(IMage Perturbation Autoencoder)와 같은 고급 모델의 성능은 알려진 생물학적 지식이나 특정 데이터셋에 크게 의존하여 일반화 능력이 약하고 폭넓은 적용성이 부족합니다. 둘째, 세포 형태 데이터는 배치 효과나 웰 위치 효과와 같은 실험적 간섭 요인의 영향을 받기 쉽고, 높은 수준의 노이즈를 나타내어 실제 세포 형태학적 특성을 효과적으로 포착하기 어렵습니다. 이는 결국 데이터의 안정성과 신뢰성에 직접적인 영향을 미치고 후속 분석 결과의 정확도를 제한합니다.
위의 과제를 해결하기 위해 홍콩 중국 대학, 모하메드 빈 자이드 인공지능 대학 및 기타 기관의 연구자들은 확장 가능한 전사체 유도 확산 모델인 MorphDiff를 제안했습니다.이 모델은 교란에 대한 세포 형태 반응을 고충실도로 시뮬레이션하도록 특별히 설계되었습니다. 잠재 확산 모델(LDM) 아키텍처를 기반으로 하며, L1000 유전자 발현 프로파일을 노이즈 제거 학습을 위한 조건부 입력으로 사용합니다.
연구 결과는 다음 사실을 확인했습니다.MorphDiff의 핵심적인 장점은 "보이지 않는 교란 조건"에서 세포 형태를 정확하게 생성할 수 있는 능력입니다.이 기능은 두 가지 주요 이점을 제공합니다. 첫째, 연구자들이 표현형 교란 스크리닝이라는 방대한 영역을 효율적으로 탐색할 수 있도록 지원하여 대규모 현장 실험에 대한 의존도를 크게 줄이고, 이를 통해 실험 비용을 절감하고 스크리닝 효율성을 향상시킵니다. 둘째, 구조적으로 다양한 약물 분자의 작용 기전을 규명하여 화합물 기전 검증에 핵심적인 기반을 제공합니다. 따라서 MorphDiff는 표현형 약물 개발을 가속화하는 고성능 도구로 활용될 수 있습니다.
해당 연구 결과는 "전사체 유도 확산 모델을 이용한 교란 상황에서의 세포 형태 변화 예측"이라는 제목으로 Nature Communications에 게재되었습니다.
연구 하이라이트:
* 이 연구는 세포 형태를 예측하기 위해 확산 모델을 혁신적으로 적용하여 처음으로 표현형 약물 개발을 위한 새로운 길을 열고 새로운 도구를 제공했습니다.
* 광범위한 벤치마크 테스트를 통해 MorphDiff의 효과성이 입증되었습니다. 특히 MOA 검색에서 MorphDiff는 기준 진실 형태학과 비슷한 정확도를 달성하여 기준 방식보다 각각 16.9%와 8% 더 우수한 성능을 보였습니다.

서류 주소:
https://www.nature.com/articles/s41467-025-63478-z
공식 계정을 팔로우하고 "전사체 유도 확산"에 답글을 달면 전체 PDF를 받을 수 있습니다.
더 많은 AI 프런티어 논문:
https://hyper.ai/papers
데이터 세트: 효과 검증을 위한 대규모 다중 오믹스 데이터 세트
교란 조건에서 세포 형태를 예측하는 MorphDiff 모델의 효과성과 일반화를 체계적으로 검증하려면본 연구에서는 "유전적 교란-약물 교란"의 이중적 차원을 기반으로 다중 세포주와 다중 소스 데이터 세트 시스템을 구축했습니다.실험에 사용된 각 샘플에 대해 두 가지 유형의 데이터, 즉 L1000 유전자 발현 프로파일과 세포 형태 이미지가 수집되어 쌍을 이루었습니다. 전자는 "분자적 특징 입력"으로, 후자는 "표현형 특징 입력"으로 사용되었습니다. 이 방법은 유전자 수준의 교란 신호와 형태적 수준의 표현형 반응 간의 상관관계를 보장하며, 이는 표적 교란에 의해서만 유도됩니다. 이는 세포주 차이 및 실험 배치와 같은 관련 없는 변수의 간섭을 제거합니다.
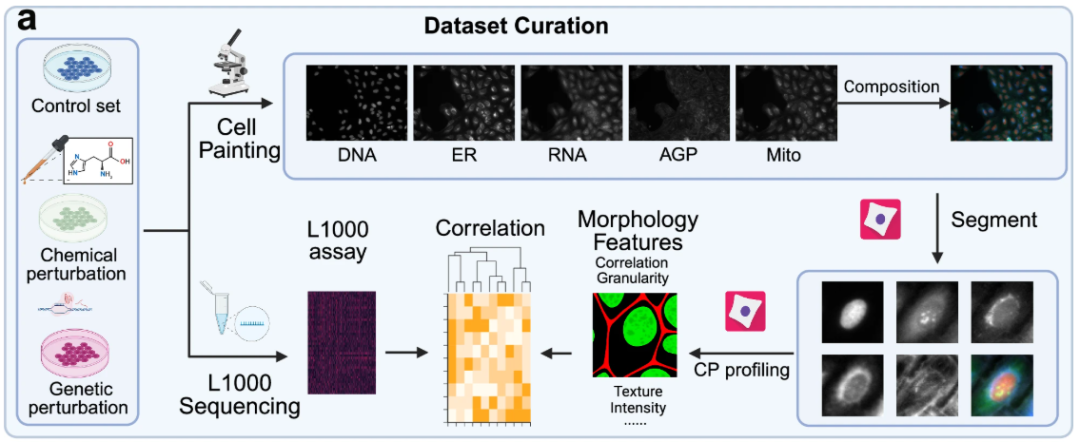
세포 형태 이미지 데이터 세트의 관점에서,본 연구에서는 모델 학습, 평가 및 분석을 위해 세 가지 대규모 세포 형태 이미지 데이터셋을 사용했습니다. 하나는 유전적 교란 데이터셋이고 다른 하나는 약물 교란 데이터셋입니다. U2OS 세포주 기반 JUMP 데이터셋에서 파생된 유전적 교란 데이터셋은 130개의 유전적 교란을 포함합니다. U2OS 세포주 기반 CDRP 데이터셋과 A549 세포주 기반 LINCS 데이터셋에서 파생된 약물 교란 데이터셋은 각각 1,028개의 약물 교란과 61개의 약물 교란을 포함합니다.
세 가지 세포 형태 이미지 데이터셋 모두 전처리 및 분할되었습니다. CellProfiler 4.2.5를 사용하여 대량 세포 플레이트 이미지를 단일 세포 이미지로 분할하여 더욱 세부적인 분석을 수행했습니다.Cell Painting 기술을 통해 얻은 세포 형태 이미지에는 5개의 핵심 채널이 포함되어 있습니다.즉, DNA(핵), RNA(핵소체와 세포질), ER(소포체), AGP(골지체/세포막/액틴 골격) 그리고 Mito(미토콘드리아)입니다.
게다가,이 실험은 또한 "해당하는 형태학적 이미지가 없는" L1000 데이터 세트를 추가로 계획했습니다.이 데이터 세트는 주로 "유전자 발현 데이터만 얻는" 시나리오에서 모델의 적용을 더욱 탐색하는 데 사용되며, 이후의 메커니즘 검증, 약물 스크리닝 등을 위한 기반을 마련하여 "데이터 기반" 가설을 제공할 수 있습니다.
모델 아키텍처 및 방법: 유전자 발현 데이터와 세포 형태 이미지 연결
MorphDiff의 핵심 목표는 전사체 유도 잠복 확산 모델 프레임워크를 통해 L1000 유전자 발현 프로필에서 세포 형태 이미지까지 종단 간 정확한 매핑을 달성하는 것입니다.간단히 말해서, "브리지"와 같은 모델을 설계하고 훈련하는 것입니다. 즉, 특정 "교란"에 해당하는 L1000 유전자 발현 데이터를 입력한 다음, 이 교란 하에서 세포의 형태학적 이미지 또는 교란 형태를 출력하는 것입니다.
MorphDiff 모델의 핵심은 두 가지 주요 모듈로 구성됩니다.아래 그림 b에 표시된 것처럼 형태 변형 자동 인코더(MVAE)와 잠재 확산 모델(LDM)입니다.
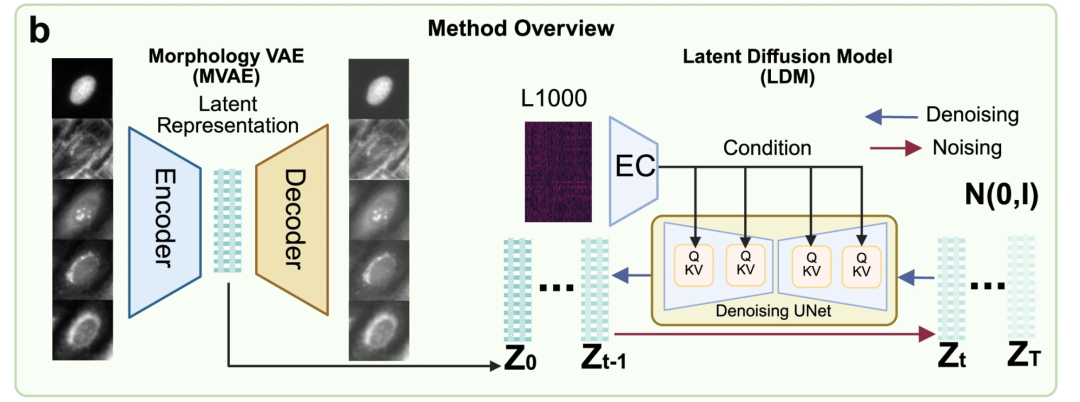
그 중 MVAE는 모델의 "이미지 압축 엔진"으로, 다채널, 고해상도 세포 형태 이미지를 저차원의 해석 가능한 잠재적 표현으로 변환하는 역할을 합니다.이 접근법은 고차원 이미지에서 확산 모델을 직접 학습하는 데 따르는 높은 연산 비용과 불안정한 학습 문제를 해결합니다. MVAE는 구조적으로 인코더와 디코더의 두 부분으로 구성됩니다. 인코더는 복잡한 5채널 형태학적 이미지를 더 단순한 저차원 표현으로 압축하고, 디코더는 이 저차원 표현으로부터 원래 형태학적 이미지를 복원합니다.
LDM은 주로 "유전자 발현"과 "압축된 형태적 특징"을 결합하는 역할을 하며, 모델이 "유전자에서 형태적 특징"까지의 관계를 완성하는 데 도움을 줍니다. LDM은 잡음 제거 과정과 잡음 제거 과정을 포함합니다. 잡음 제거 과정은 압축된 형태적 특징에 가우시안 잡음을 점진적으로 추가하여 완전히 무작위가 되도록 합니다. 잡음 제거 과정을 통해 모델은 L1000 유전자의 알려진 발현을 고려하여 무작위 잡음을 원래 형태적 특징에 점진적으로 복원할 수 있습니다. 이 모델은 U-Net 네트워크 아키텍처를 사용하고 어텐션 메커니즘을 통합하여 주요 유전 정보와 형태적 정보를 더욱 정확하게 연결합니다.
아래 그림 c는 사전 학습된 MorphDiff 모델의 두 가지 적용 사례, 즉 G2I와 I2I를 보여줍니다. G2I는 L1000 유전자 발현을 조건으로 한 랜덤 노이즈 분포로부터 해당 세포 형태 이미지의 노이즈를 제거하여 해당 세포 형태 이미지를 생성합니다. I2I는 특정 섭동 조건에서 L1000 유전자 발현을 조건으로 하여 대조군 세포 형태 이미지를 예측된 섭동된 형태 이미지로 변환하여 "정상 형태에서 섭동된 형태"로 예측하는 기능을 구현합니다.
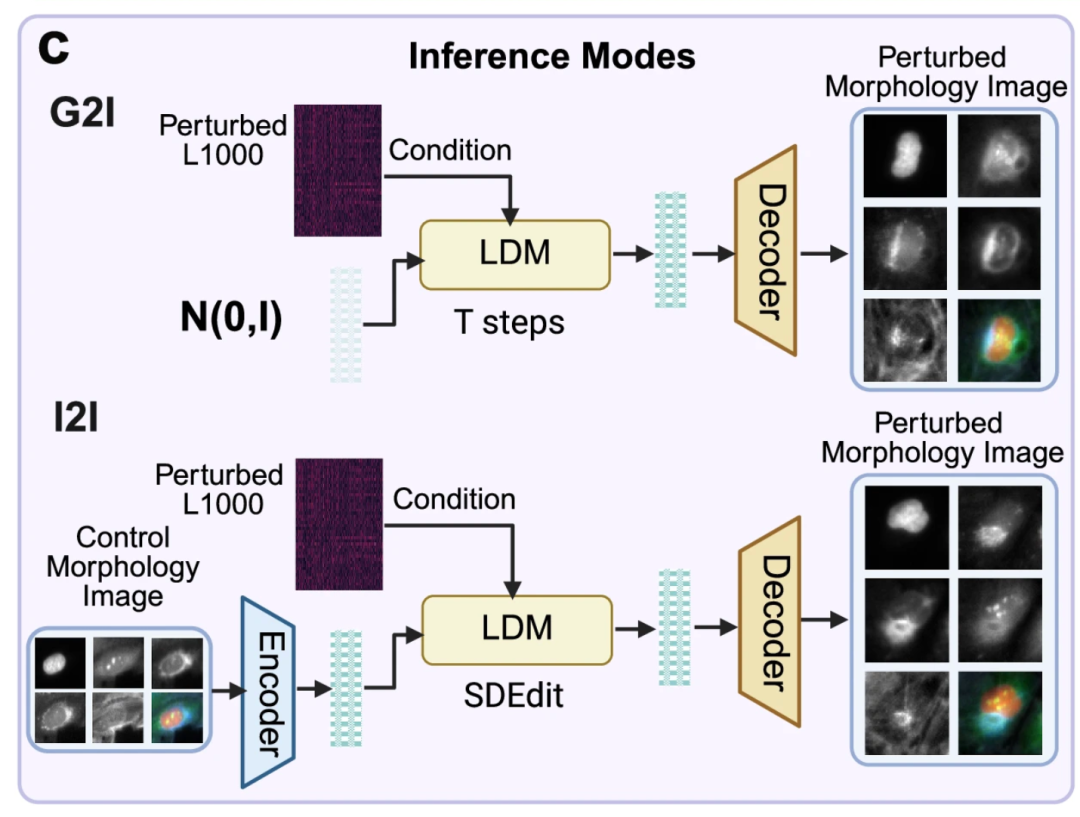
아래 그림 d는 MorphDiff 모델이 실제 응용 분야에서 갖는 가치를 보여줍니다.첫째, 이 모델은 훈련 중에는 발생하지 않았던 "보이지 않는 교란"으로 인한 세포 형태학적 변화를 예측할 수 있습니다.이를 통해 연구자들은 물리적 실험을 수행하지 않고도 컴퓨터로 신약의 교란에 따른 세포 변화 상태를 시뮬레이션할 수 있어 더 많은 가능성을 빠르고 저렴하게 탐색할 수 있습니다.모델 프레임워크는 CellProfiler와 DeepProfiler와 같은 도구를 결합했습니다.이는 약물의 작용기전을 파악하는 데 도움이 되어 표현형 약물의 개발을 촉진합니다.
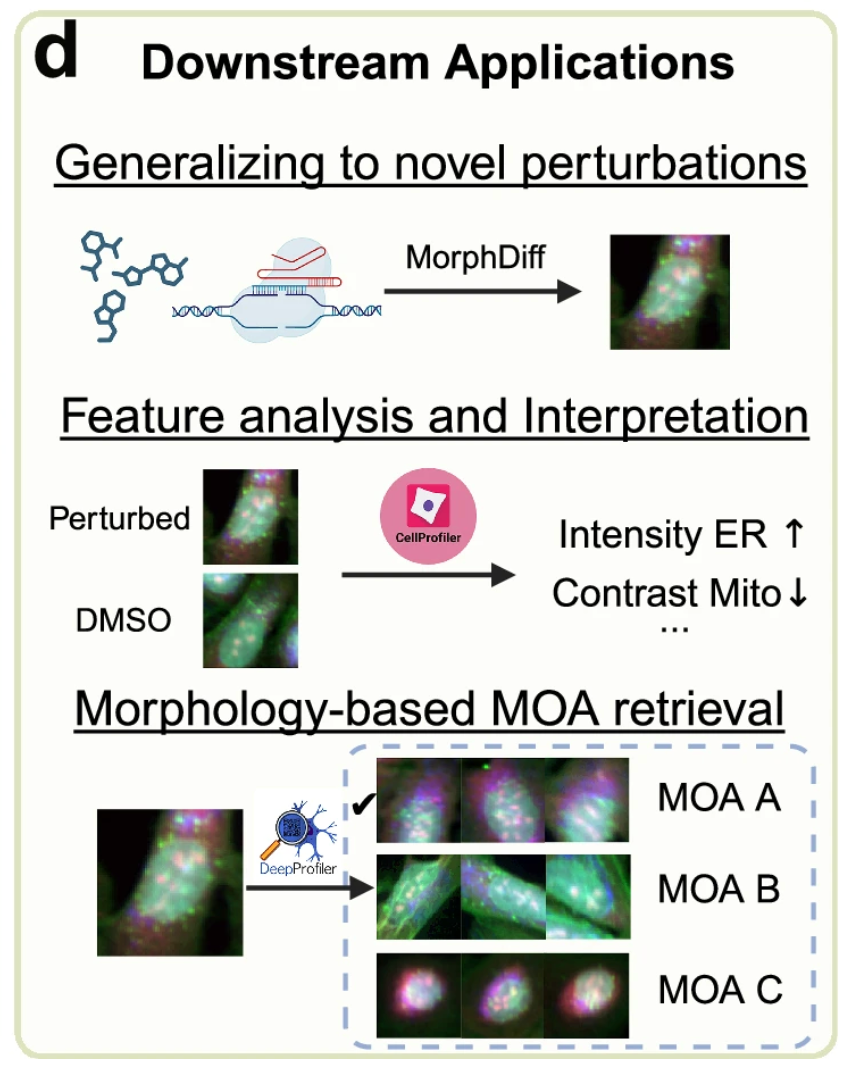
연구자들은 MorphDiff가 현재 유전자 발현으로부터 형태학적 이미지를 생성하고, 교란되지 않은 형태에서 교란된 형태로 변환하는 것을 지원하는 유일한 도구라고 말합니다.
실험 결과: IMPA를 능가하는 성능으로 표현형 약물 개발 가속화
MorphDiff 모델의 효과를 검증하기 위해 연구자들은 다양한 목적에 맞는 일련의 실험을 설계했습니다.고급 도구와의 실험적 비교를 통해 MorphDiff의 효과와 실용성이 체계적으로 검증되었습니다.
첫째, 실험을 통해 유전적 교란 예측이 검증되었습니다.연구진은 JUMP OOD 데이터 세트에 대한 벤치마크 테스트를 수행하고 MorphNet, DMIT(Disentanglement for Multi-mapping Image-to-Image Translation), DRIT++(Disentangled Representation for Image-to-Image Translation), StarGANv1, IMPA, VQGAN(Vector Quantized Generative Adversarial Network), MDTv2(Masked Diffusion Transformers)를 포함한 여러 기준 방법과 비교했습니다.
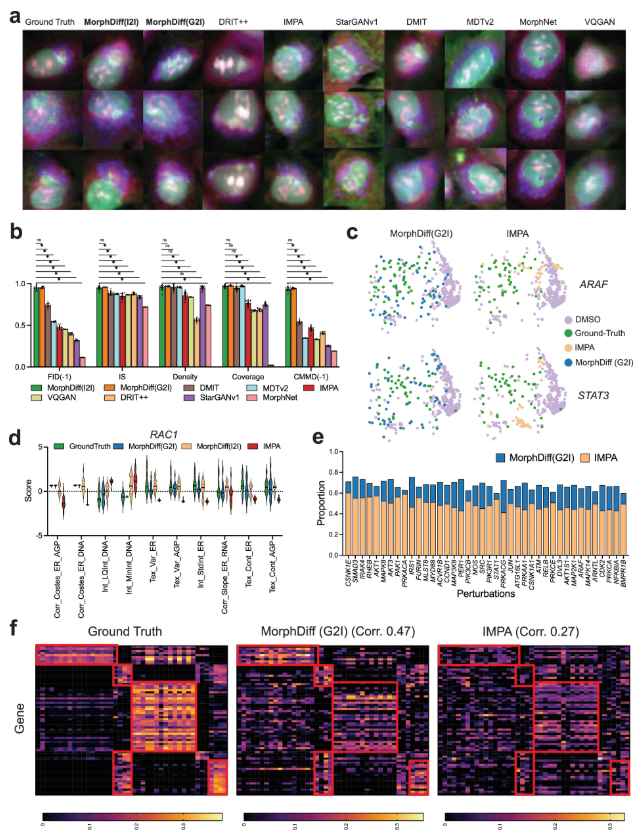
결과는 다음과 같습니다MorphDiff의 두 가지 모드로 생성된 결과는 시각적 품질과 구조적 형태 측면에서 실제 기준선에 더 가깝습니다.FID, IS(Inception Score), CMMD, 밀도, 커버리지를 포함한 정량적 지표 측면에서 두 MorphDiff 모드 모두 일반화, 충실도, 다양성 측면에서 기준선 모델보다 우수한 성능을 보였으며, 더 높은 출력 품질을 달성했습니다. 세포 형태 변화를 예측할 때, MorphDiff(G2I) 출력은 더욱 다양하고 실제 기준선에 더 가깝습니다. 반면, MorphDiff(I2I)로 생성된 특징은 실제 기준선 특징과 더 높은 겹침을 보이는데, 이는 예측 정확도가 실제 섭동 형태와 상당히 일치함을 나타냅니다.
이후, 약물 교란 예측을 검증하기 위한 실험이 수행되었습니다. 연구진은 먼저 CDRP OOD 데이터 세트를 사용하여 모든 방법을 벤치마킹했습니다.MorphDiff는 대부분의 지표에서 다른 기준 방법에 비해 인상적인 성능을 달성하여 더욱 강력하고 안정적인 종합적 생성 기능을 보여줍니다.그런 다음 연구자들은 LINCS 데이터 세트에서 모델에 대한 보다 엄격한 평가를 수행하여 생성된 결과를 CellProfiler 기능과 비교하여 소분자 화합물에 대한 세포 형태의 반응을 추가로 조사했습니다.
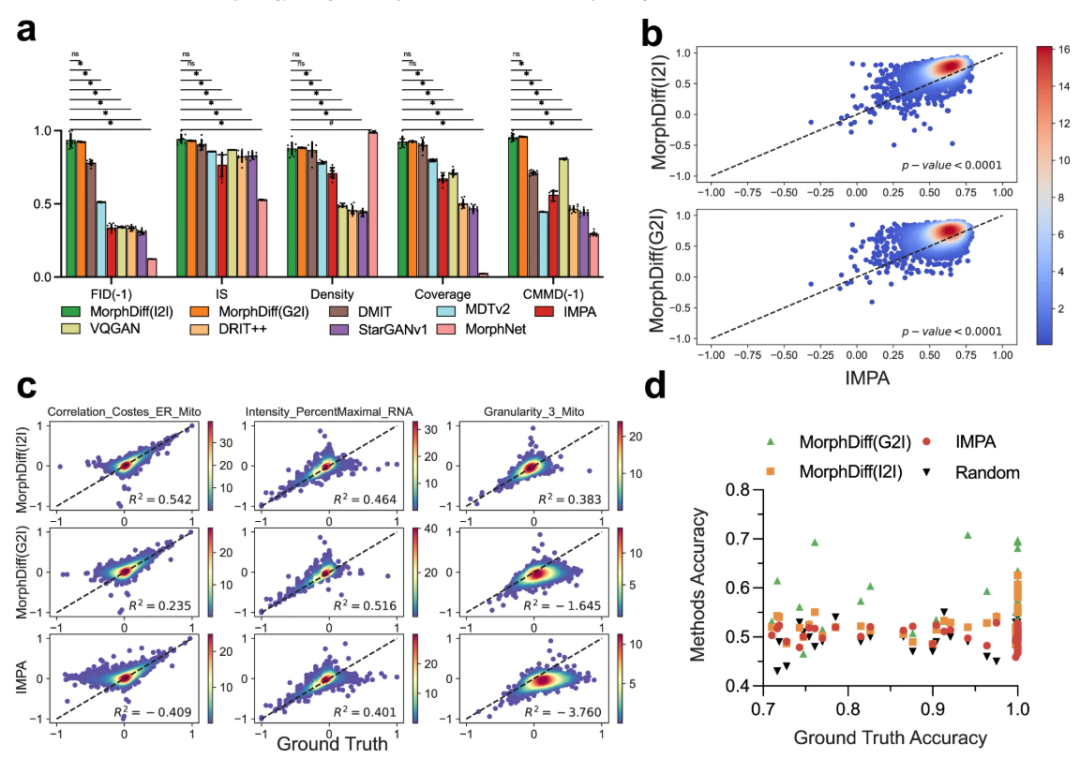
결과는 MorphDiff의 두 모드 모두 기준선보다 상당히 우수한 성능을 보인다는 것을 보여줍니다.G2I 분석에서는 87.61개의 TP3T 샘플이 0.5보다 높은 점수를 받았고, 16.21개는 0.8을 초과했습니다. I2I 분석에서는 891개의 TP3T 샘플이 0.5보다 높은 점수를 받았고, 27.21개는 0.8을 초과했습니다. 반면, IMPA 분석에서는 78.31개의 TP3T 샘플이 0.5보다 높은 점수를 받았지만, 0.8을 초과하는 샘플은 없었습니다. CDRP OOD 데이터셋과 LINCS leave-one-out 데이터셋에 대한 동일한 분석 결과, 두 MorphDiff 모델 모두 p값이 0.0001 미만으로 기준선보다 우수한 성능을 보였으며, 이는 해당 방법의 일반화 가능성을 보여줍니다.
DeepProfiler 임베딩 분석에서MorphDiff(G2I)는 교란에 따른 세포 형태학적 패턴을 가장 안정적이고 정확하게 포착할 수 있습니다.제약 수준에서,G2I 패턴으로 생성된 출력은 I2I 패턴보다 섭동 특이도가 더 높습니다. 이러한 결과는 약물 스크리닝에서 MorphDiff의 잠재력을 더욱 잘 보여줍니다.
마지막으로, 이 실험은 MorphDiff의 약물 개발 역량을 검증했습니다. 연구진은 MorphDiff의 두 가지 응용 모드와 IMPA를 벤치마킹하기 위해 CDRP Target_MOA 데이터셋을 선택했습니다.
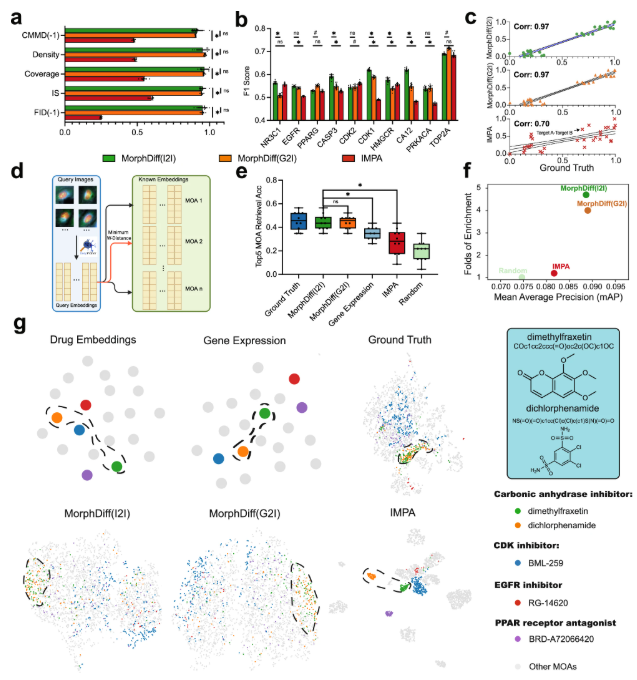
결과는 MorphDiff로 생성된 섭동 형태가 실제 기준선과 매우 일치한다는 것을 보여줍니다.이는 표적 관련 다양성을 포착하고 약물 섭동과 형태 사이의 복잡한 관계를 효과적으로 학습하는 능력을 입증했습니다. 작용 기전(MOA) 검색 작업에서 MorphDiff로 생성된 결과의 평균 정확도는 IMPA 기반 검색보다 16.9%, 유전자 발현 기반 검색보다 8% 높았습니다. 또한, MorphDiff는 세포 형태가 상호 보완적인 정보를 포함하고 있음을 발견하고 작용 기전은 동일하지만 구조가 다른 약물을 식별할 수 있음을 실험을 통해 입증했으며, 이는 표현형 약물 개발을 가속화하는 데 도움이 될 수 있습니다.
실리콘 기반 시뮬레이션이 주류가 되어 하류 약물 개발이 가속화됩니다.
표현형 기반 신약 개발은 표적 기반 신약 개발과 달리, 약물이 전반적인 생물학적 시스템이나 세포 표현형에 미치는 영향을 관찰하여 약물을 탐색하고 개발합니다. 이러한 접근 방식은 새로운 작용 기전과 표적을 발견하고 복잡한 질병을 치료하는 데 상당한 이점을 제공합니다. 수많은 실험실과 연구 기관들이 이 주제를 적극적으로 연구하고 있으며, 계산 기술과 생물의학을 통합하여 표현형 기반 신약 개발의 새로운 장을 열고 있습니다.
논문에서 언급했듯이 IMPA 모델은 독일 뮌헨 공과대학교와 영국 옥스퍼드대학교의 팀이 제안한 심층 생성 모델입니다.스타일 전송 방법을 사용하면 세포 이미지를 "스타일"(섭동/배치 표현)과 "컨텐츠"(세포 표현)로 분해할 수 있으며, 이를 통해 세포의 섭동에 대한 반응을 예측하고 배치 효과를 제거할 수 있습니다."생성 모델링을 사용하여 섭동에 대한 세포 형태학적 반응 예측"이라는 제목의 논문도 Nature Communications에 게재되었습니다.
또한 미시간 대학 팀이 발표한 "MorphNet은 단일 세포 유전자 발현에서 세포 형태를 예측합니다"라는 제목의 기사에서MorphNet이라는 계산적 방법을 제안했는데, 이는 유전자 발현 프로필을 기반으로 세포의 형태적 이미지를 그릴 수 있습니다.이 방법은 짝을 이룬 형태학적 데이터와 분자적 데이터를 사용하여 유전자 발현을 기반으로 핵이나 전체 세포의 형태를 예측하도록 신경망을 훈련시킵니다.
요약하자면, 유전적 또는 약물적 교란 하에서 세포 상태의 변화를 관찰하고 분석함으로써 표현형 약물 개발 및 생물학 연구를 촉진하는 것이 중요한 주제가 되었습니다. MorphDiff는 훈련 데이터 외부의 수많은 새로운 교란에 직면하여 보이지 않는 교란을 예측해야 하는 어려움과 같은 많은 단점을 여전히 가지고 있지만, MorphDiff가 이전 버전들을 기반으로 끊임없이 발전하고 있으며 실용성, 일반화, 사용 편의성 및 확장성 면에서 이전 버전들을 능가하고 있다는 것은 분명합니다.
2023년부터 2024년까지 AI4S 분야의 고품질 논문과 심층 해석 기사를 클릭 한 번으로 받아보세요⬇️
