Command Palette
Search for a command to run...
데이비드 베이커의 팀은 Science에 약물로 치료할 수 없는 표적을 특별히 표적으로 삼아 무질서한 영역 결합 단백질을 설계하는 새로운 접근 방식을 제안했습니다.

대부분의 질병은 비정상적인 단백질 기능과 직접적으로 연관되어 있기 때문에 단백질은 약물 개발에 중요한 역할을 합니다. 신약을 개발할 때 연구자들은 종종 단백질을 핵심 약물 표적으로 삼아, 약물이 부분적으로 안정한 단백질에 결합하여 질병 과정에 개입할 수 있도록 합니다.그러나 구조적, 서열적, 구조적 선호도가 명확하게 정의되지 않은 본질적으로 무질서한 단백질(IDP)을 표적으로 삼아 약물을 투여하는 것은 여전히 어려운 일입니다.
항체 표적화의 기존 방식은 표적 단백질을 인식하고 조절하기 위해 항체가 특정 단백질에 매우 특이적으로 결합하는 능력에 주로 기반합니다. 그러나 이러한 표적화 경로는 많은 실험적 조작을 필요로 할 뿐만 아니라, 무질서한 항원은 주입 후 쉽게 분해되어 효과가 없습니다. 따라서,프로테옴에서 50% 이상을 차지하는 본질적으로 무질서한 영역(IDR)을 가진 단백질은 일반적으로 "치료가 불가능한" 표적으로 간주되며 약물 개발에 사용된 적이 없습니다.
이러한 맥락에서, 2024년 노벨 화학상을 수상한 뛰어난 계산 생물학자이자 워싱턴 대학교 단백질 디자인 연구소 소장인 데이비드 베이커와 그의 팀은 로고스(Logos)라는 단백질 디자인 전략을 제안했습니다.유도적 적합 결합 전략에 기초하여, 39개의 표적 무질서 아미노산 서열에 적응할 수 있는 결합 단백질이 설계되었습니다.이 연구는 특수 확장 반복 단백질 백본을 생성한 후 RFdiffusion 모델을 사용하여 일반화했습니다. 이 백본은 반복 펩타이드 서열을 위해 특별히 설계된 포켓을 포함하고 있어, 설계된 바인더-타겟 펩타이드 템플릿이 무질서한 단백질 영역을 보편적으로 인식할 수 있도록 합니다. 이는 더 많은 단백질이 신약 개발의 표적이 될 수 있음을 의미하며, 이는 암과 알츠하이머병 연구를 가속화할 수 있는 잠재력을 가지고 있습니다.
관련 연구 결과는 "본질적으로 무질서한 영역 결합 단백질의 설계"라는 제목으로 Science에 게재되었습니다.
연구 하이라이트:
*모든 표적 서열에 대한 결합 적응 형태 유도를 달성하기 위해 일반적인 인식에 적합한 템플릿 구조 라이브러리를 구축합니다.
* 광범위한 다양성과 치료적 잠재력을 지닌 18개의 합성 펩타이드 서열과 21개의 자연적 무질서 영역(IDR)에 대한 결합 단백질을 설계하여 암 관련 세포외 수용체의 무질서 영역을 표적으로 삼고 세포 내에서 단백질 국소화를 촉진할 수 있습니다.

서류 주소:
https://www.science.org/doi/10.1126/science.adr8063
공식 계정을 팔로우하고 "자연 무질서 단백질"에 답글을 달면 전체 PDF를 받을 수 있습니다.
더 많은 AI 프런티어 논문:
템플릿 라이브러리 생성: 범용 펩타이드 식별
본 연구는 IDR 결합 문제에 대한 해결책으로 물리적 설계 방법과 딥러닝 설계 방법을 결합합니다. 펩타이드 단위와 이질적 표적 서열의 비호환성으로 인해 제약을 받습니다.이 연구는 다양한 반복 단백질 구조로 시작하여 확산 모델을 사용하여 다양한 반복 단위의 아미노산 결합 포켓을 재구성하고 이를 다양한 아미노산과 구조적 템플릿으로 구분했습니다.이를 통해 시퀀스를 더 폭넓게 인식할 수 있습니다.
자연적으로 무질서한 단백질에서 펩타이드를 확인하기 위해, 이 연구는 먼저 백본 템플릿 라이브러리를 구축했습니다. 템플릿 라이브러리는 두 가지 특징을 가지고 있습니다.
*각 템플릿 구조는 늘어난 펩타이드 사슬 형태를 "감쌀" 수 있어야 하며 수소 결합 및 단단한 패킹과 같은 상호작용을 위한 많은 기회를 제공해야 하며, 이를 통해 표적 서열에 대한 매우 구체적인 인식을 달성해야 합니다.
* 템플릿 구조는 광범위하여 모든 표적 서열과 일치할 수 있으므로 적어도 하나의 템플릿이 이를 정의되고 적합한 결합 형태로 유도할 수 있습니다.
백본 템플릿 라이브러리를 생성하는 과정은 백본 생성, 단백질 활성 포켓 특수화, 단백질 활성 포켓 조립의 세 단계로 나뉩니다.
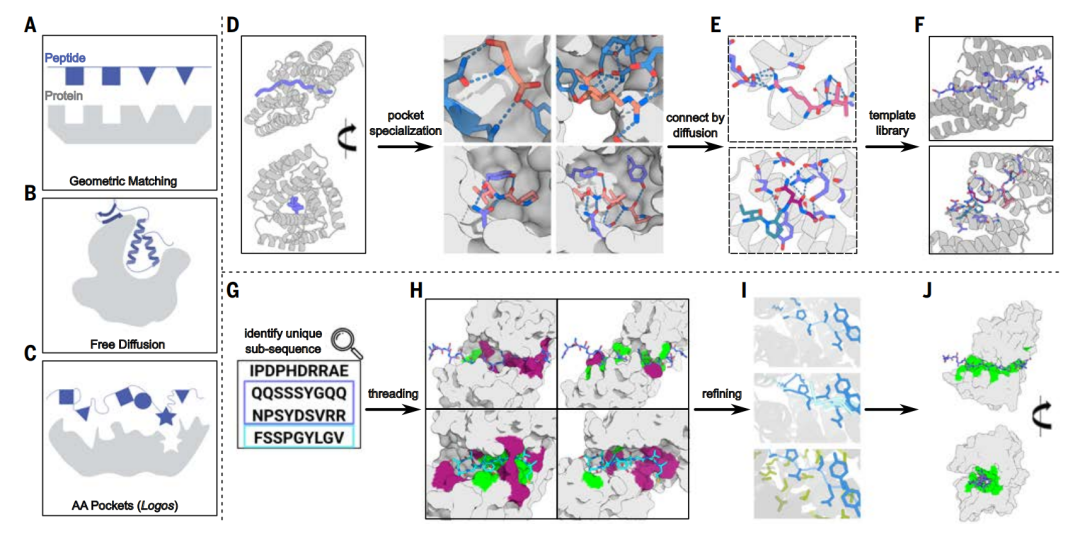
스캐폴드 생성
연구진은 백본 생성 단계에서 폴리프롤린 II 구조에 국한하지 않고 여러 확장된 구조를 표적으로 삼기로 했습니다. 폴리프롤린 II 구조는 주로 프로린이 풍부한 펩타이드에서 발생하기 때문입니다.
늘어난 구조에서는 아미노산의 측쇄가 번갈아 반대 방향을 향하고 있는데, 이는 2잔기 반복의 특성과 일치합니다.연구진은 로제타 설계 방법을 사용하여 일련의 디펩타이드 반복 서열을 설계했습니다.LK, RT, YD, PV 및 GA(모두 아미노산의 단일 문자 약어)를 포함하여, 이들은 서로 다른 늘어난 구조로 이러한 펩타이드 세그먼트와 얽히고 결합하도록 설계되어 각 반복 단위가 디펩타이드 단위와 상호 작용합니다.
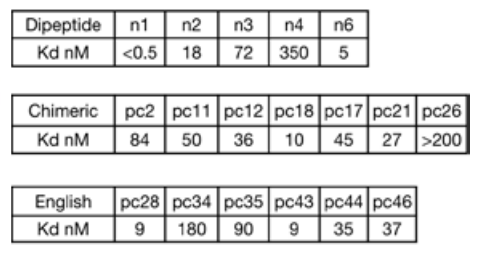
이후, 연구진은 형광 편광 실험을 통해 이렇게 설계된 4-반복 단위 결합 단백질의 특성을 분석했습니다. 그 결과, LK 및 PV 반복 펩타이드에 대해서는 나노몰 단위의 결합력을 보였지만, 극성이 더 강한 RT 및 YD에 대해서는 결합력이 약했고, 유연성이 높은 GA에서는 결합 신호가 전혀 검출되지 않았습니다.
포켓 스페셜리제이션
단백질 활성 포켓을 지정하는 단계에서 연구자들은 확산 모델링을 사용하여 포켓을 미세 조정하여 특정 표적 펩타이드 서열과 더 정확하게 일치하도록 했습니다.
템플릿 매칭 효율을 향상시키기 위해 연구진은 설계된 결합 포켓을 개선하여 상호작용하는 반복 단위의 수를 4개에서 5개로 늘리는 동시에 표적 서열과의 매칭도 향상시켰습니다. 이러한 접근 방식은 표적 구조 간의 친화도 또한 향상시켰습니다. 반복 단백질과 펩타이드 골격 사이의 각 측쇄에 있는 분기 수소 결합을 둘러싼 4~9개의 아미노산은 고정된 상태로 유지되었고, 설계된 결합 단백질 간의 소수성 상호작용은 다양화되었습니다.
이 전략의 장점은 수소 결합의 기하학적 구성 요건이 더 엄격하다는 것입니다. 이에 비해 비극성 소수성 적층은 공간적 자유도가 더 높습니다. 따라서 설계 시 수소 결합을 처음부터 반복적으로 샘플링하는 것보다 템플릿 방식으로 수소 결합을 직접 유지하는 것이 더 효율적입니다.
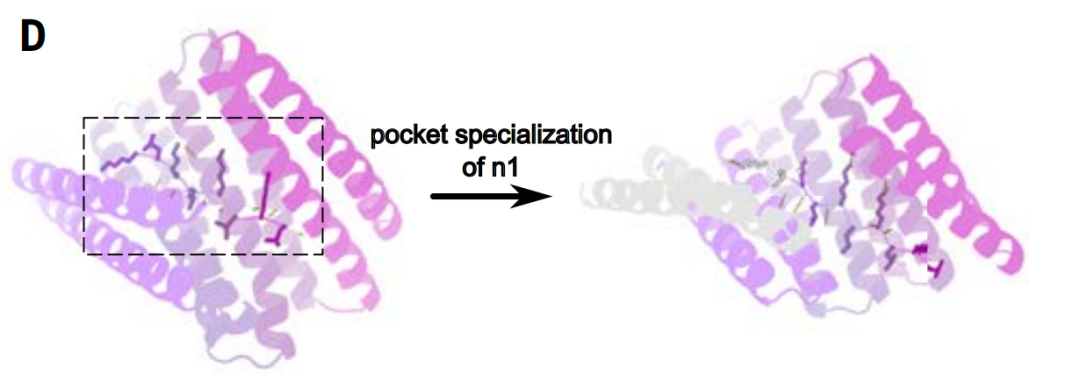
새로 확장된 다섯 번째 반복 구조는 밝은 회색으로 표시됩니다.
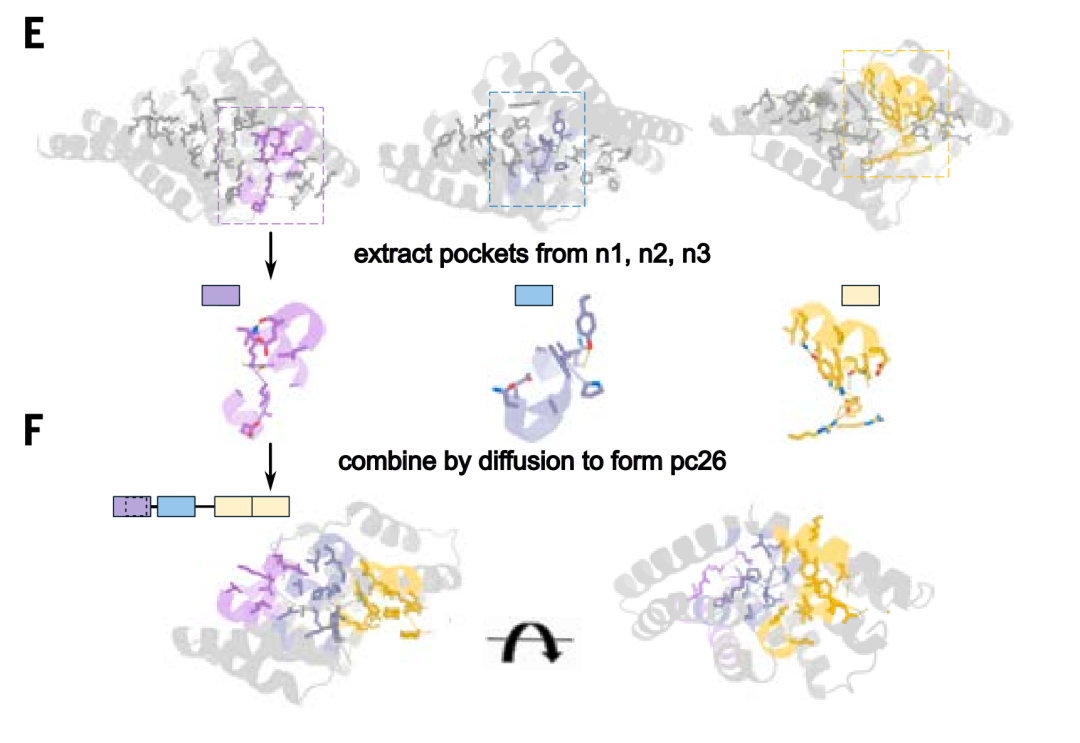
포켓 어셈블리
포켓 조립 단계 동안,연구진은 RF 확산 모델을 사용하여 포켓 사이의 인터페이스를 생성하여 전체적으로 견고한 구조를 만들고 결합 포켓을 새로운 백본으로 조립하기 위한 템플릿을 생성했습니다.템플릿의 다양한 포켓은 서로 다른 순서와 기하학적 구조에 따라 배열되어 일련의 확장된 형태로 펩타이드 표적과 상호 작용하며, 반복되지 않는 서열을 보다 일반적으로 인식할 수 있습니다.
키메라 펩타이드 표적과 상호작용하는 키메라 단백질 모델을 생성한 후, 본 연구는 매개 변수적으로 결합 포켓의 위치를 파악하고 고주파 확산법을 통해 연결했습니다. 이 접근법을 사용하여 7개의 키메라 표적에 대해 70개의 설계 제안서를 생성했습니다. 분할 루시퍼라제 존재비 실험과 생물층 간섭계 실험을 통한 특성 분석 결과, 표적당 평균 10개의 설계만 시험했음에도 불구하고 7개 표적 중 6개가 두 자릿수 나노몰 수준의 결합을 달성했습니다.
더 광범위한 시퀀스를 포괄하도록 템플릿 라이브러리의 크기를 확장하기 위해 이 연구에서는 포켓 조립 기술을 사용하여 극성 잔류물을 인식하는 포켓을 포함하는 36개의 키메라 백본을 구성하고, 설계된 결합 단백질과 해당 펩타이드 백본으로 구성된 1,000개의 템플릿을 생성했습니다. 여기서 펩타이드 구조의 아미노산은 결합 단백질의 설계된 포켓과 일치할 수 있습니다.
IDR 결합 단백질 설계 및 최적화
연구진은 템플릿 라이브러리를 구축한 후, 자연적으로 무질서한 영역을 템플릿 라이브러리에 삽입하고 이를 이용하여 반복되지 않는 합성 서열과 자연적으로 구조화되지 않은 모든 표적에 결합할 수 있는 결합 단백질을 생성했습니다. 이 단계는 스레딩 매칭과 구조적 정밀화의 두 부분으로 나뉩니다.
스레드 매칭: 가장 호환되는 시퀀스 조각-템플릿 쌍 결정
스레딩 매칭에서는 대상 시퀀스를 각 템플릿의 백본에 스레딩하여 템플릿과 가장 호환되는 시퀀스 조각을 식별합니다.
일반적으로 IDP 또는 IDR은 표적 펩타이드로 사용할 수 있는 펩타이드가 매우 많습니다. IDR에서 가장 큰 표적화 잠재력을 가진 펩타이드를 찾으려면,이 연구에서는 먼저 서열 복잡성이 낮은 펩타이드와 프로테옴에서 여러 개의 밀접한 일치를 보이는 펩타이드를 제거하여 이러한 표적의 결합제와의 교차 반응을 방지했습니다.템플릿 라이브러리의 타겟 백본에 남아 있는 아미노산의 고유한 서열 조각을 매핑하여 로컬 백본 리샘플링 후,이 연구에서는 딥러닝 기반 단백질 서열 설계 도구인 ProteinMPNN을 사용하여 결합 단백질의 서열을 최적화하고, 설계된 결합 단백질과 표적 서열 간의 적합성, AF2 예측값과 모델 간의 일관성을 기준으로 이를 평가했습니다.
AF2 지표가 최적이 아닌 경우, RFdiffusion을 사용하여 특정 표적에 대한 백본을 맞춤화했습니다. 그런 다음 스레드 매칭을 사용하여 치료적으로 관련 있는 IDP, IDR 및 IDP 단편에 대한 바인더를 생성하여 표적당 평균 28개의 디자인을 생성했습니다.
구조 최적화: 결합 단백질과 표적 펩타이드 간의 일치성 향상
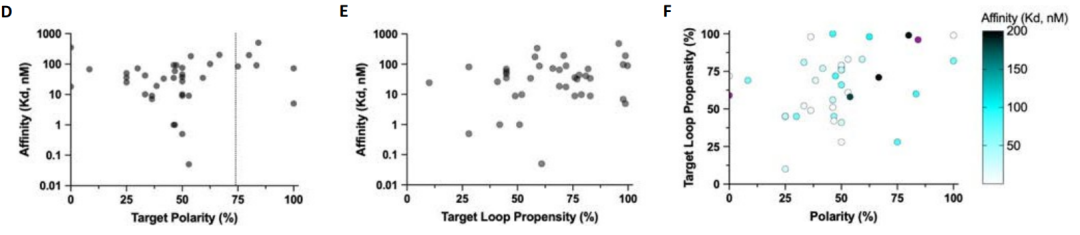
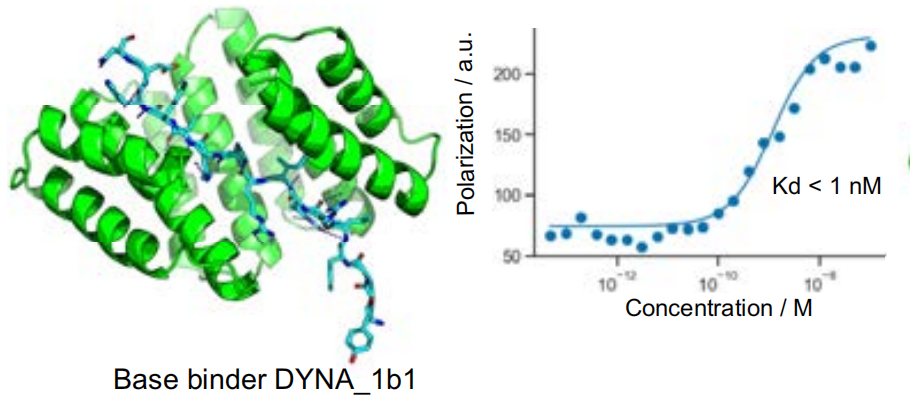
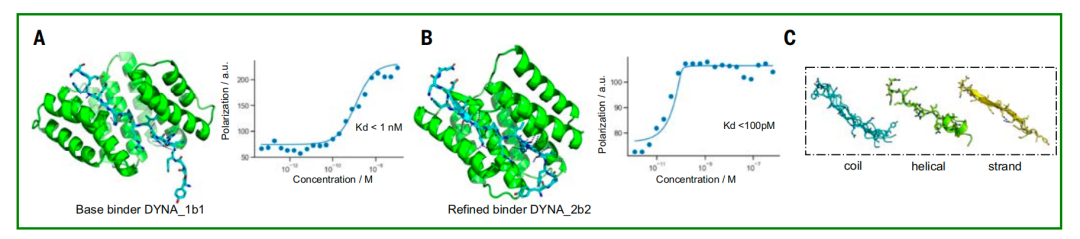
또한, 설계된 결합 단백질과 표적 펩타이드 사이의 적합성을 높이기 위해 최적의 매치가 최적화되었습니다.이 연구에서는 DYNA_1b1 결합 단백질과 디노르핀의 해리 상수를 선택하여 테스트하고, 합성 표적의 적중률을 가장 높이기 위해 고주파 확산을 최적화했습니다.결과는 48개 디자인 중 45개가 스크리닝 테스트에서 강한 친화성을 보였고, 6개 디자인의 해리 상수만이 약한 친화성을 보였다는 것을 보여주었습니다.
디노르핀 구조와 결합 단백질 간의 직교성 검증
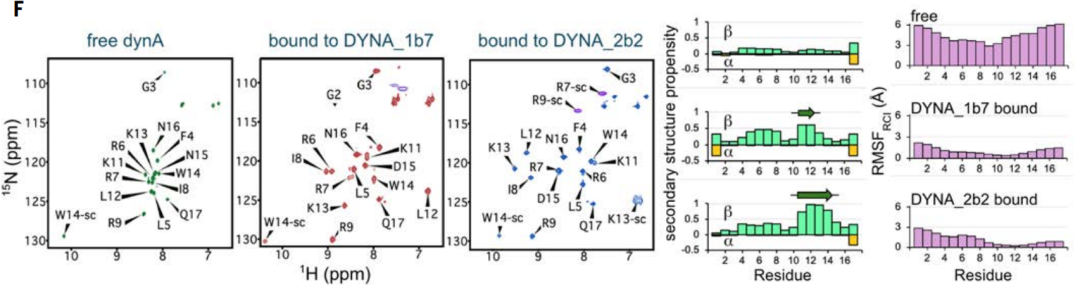
결합 중 다이노르핀 구조의 변화를 확인하기 위해, 동위원소 표지된 다이노르핀 A의 핵자기공명(NMR) 스펙트럼을 용액에서 검출했는데, 이때 다이노르핀 A는 결합되지 않았을 때, DYNA_1b1에 결합되었을 때, 그리고 더 높은 친화도로 DYNA_2b2에 결합되었을 때였습니다.
NMR 결과로부터, 유리된 디노르핀 A는 본질적으로 무질서하지만, 설계된 골격에 포함된 영역은 결합 시 질서가 잡힌다는 것이 밝혀졌습니다.두 결합된 복합체 모두에 대해 NMR 데이터는 확장된 결합 상태 형태를 나타냈는데, 이는 설계된 모델과 일치하며, 무질서한 단백질과 펩타이드를 비자연적인 형태로 유도하는 데 있어 디노르핀 작용의 효과를 확인시켜 주었습니다.
Logos의 최적화 가능성을 탐색하기 위해 연구진은 결합 상수(Kd)가 약 1nM인 다이노르핀 결합제 DYNA_1b1을 선택했습니다. 연구진은 RFdiffusion을 사용하여 상위권 디자인을 최적화했습니다. 48개 디자인 중 45개는 BLI 스크리닝을 통해 5nM 농도에서 강력한 결합을 보였으며, 6개는 BLI 측정 결과 Kd 값이 ≤100pM이었습니다. 최적화된 디자인 중 두 개(DYNA_2b1과 DYNA_2b2)에 대한 형광 편광 측정 결과, 아래 그림 B와 같이 각각 60pM과 100pM 미만의 Kd 값을 나타냈습니다.
참고사항: 디노르핀은 만성 통증과 관련된 카파-오피오이드 수용체(KOR) 펩타이드 리간드입니다.
다이노르핀 A의 최초 설계 및 최적화된 설계에서, 이 펩타이드는 위 그림 C에서 볼 수 있듯이 랜덤 코일, 부분 β-가닥 구조, 부분 α-나선 구조를 포함한 다양한 형태를 나타냈습니다. 다이노르핀 A와 B는 62%의 서열 유사성을 공유하지만, 각각의 결합 단백질은 서로 교차하지 않고 각각의 표적에만 결합합니다. 동시에, 다이노르핀 A에 결합된 설계된 단백질 DYNA_1b7의 공결정 구조는 특히 핵심 결합 계면에서 계산 설계 모델과 매우 일치합니다(위 그림 DE). NMR 데이터 또한 다음 사실을 추가로 확인했습니다.원래 무질서했던 디노르핀 A의 골격은 설계된 단백질과 결합한 후 질서를 되찾아 유도적 적합 메커니즘의 효과를 다시 한번 확인했습니다(위의 그림 F).
결합 단백질의 기능 및 직교성 확인
본 연구에서는 WASH 복합체와 PER 복합체의 면역침전 모델을 사용했습니다. WASH 복합체는 WASH, FAM21, CCDC53, SWIP, 그리고 WASHC2를 포함합니다. 검사 결과, FAM21_1b1은 세포 용해물에서 WASH 복합체 전체를 추출하는 것으로 나타났습니다.
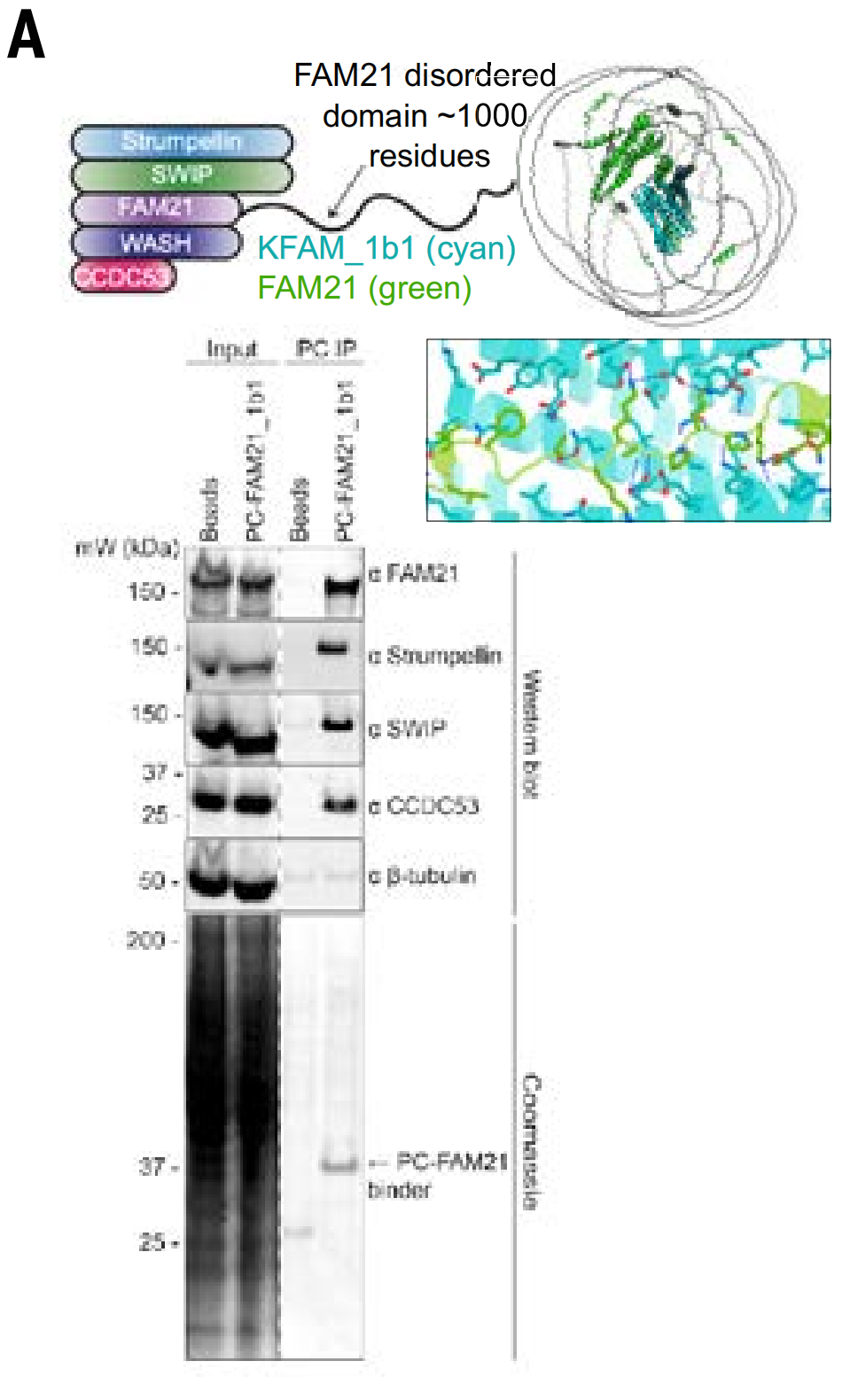
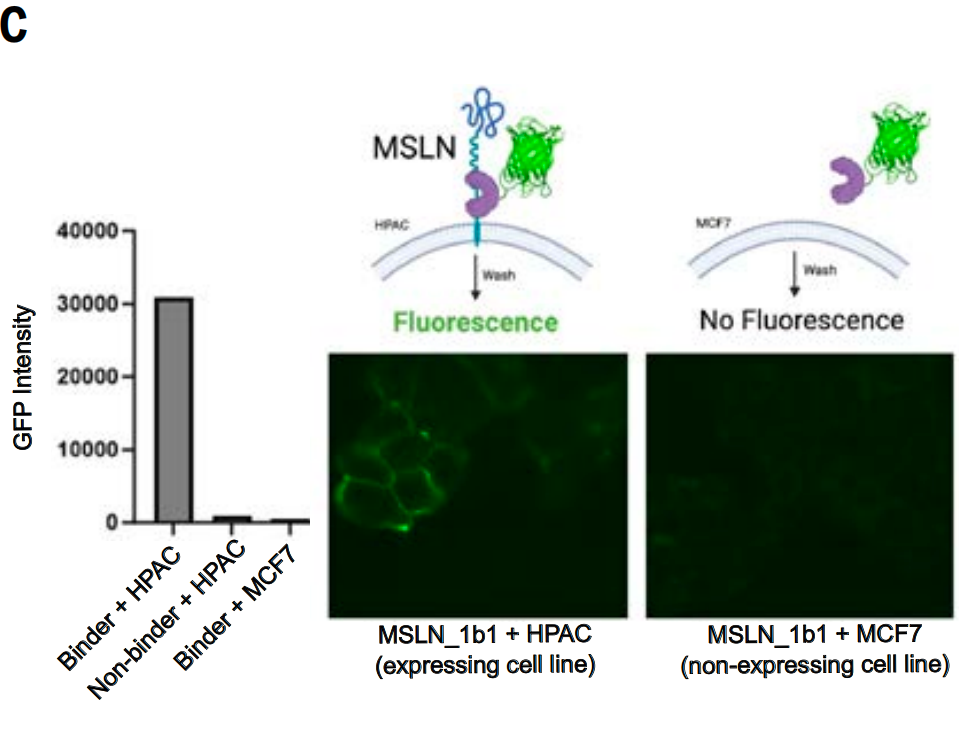
또한, 그들은 MSLN 막근접 영역을 위해 설계된 결합 단백질(MSLN_1b1)이 이 표적을 발현하는 세포에 특이적으로 결합할 수 있는지도 연구했습니다(이 영역에서 프로테아제 분해가 일어나기 때문에 더 먼 세포외 도메인 영역은 표적으로서 적합하지 않기 때문입니다).
참고사항: 메소텔린(MSLN)은 많은 암에서 상향조절되는 세포 표면 당단백질로, 종양 표적 치료에서 많은 주목을 받고 있습니다.
연구진은 녹색 형광 단백질(GFP)을 MSLN_1b1에 융합시키고 MSLN을 발현하는 세포(인간 췌장선암 세포주 HPAC)와 MSLN을 발현하지 않는 세포주(미시간 암 재단 유방암 세포주 MCF7)와 함께 배양했으며, MSLN에 결합하지 않는 GFP 융합 단백질을 대조군으로 포함시켰습니다.
형광 현미경 검사 결과, GFP-MSLN_1b1 융합 단백질이 HPAC 세포의 세포 접합부에서 응집되는 것을 확인했는데, 이는 MSLN의 국소화 특성과 일치합니다. 이러한 현상은 MCF7 대조군 세포에서는 관찰되지 않았습니다. 또한, 대조군 결합 단백질은 아래 그림 C에서 볼 수 있듯이 HPAC 세포에서 결합 신호를 보이지 않았습니다. 따라서 MSLN_1b1은 세포 표면의 MSLN을 특이적으로 인식하고 결합합니다.
AI 기반, 단백질 타겟팅의 새로운 전망 열어
현재 AI는 단백질 표적 연구에 점점 더 적극적으로 참여하고 있으며, 연구를 "다중 기술 병렬화"라는 새로운 단계로 이끌고 있습니다. 데이비드 베이커 팀 외에도 펜실베이니아 대학교의 조지 M. 버슬렘과 오피르 샬렘 팀 또한 표적 단백질 연구에서 혁신적인 돌파구를 마련했습니다. 이 팀은 "단백질 편집" 기술을 제안하여, 분리된 인테인 시스템을 이용하여 살아있는 포유류 세포에서 단백질 합성 후 아미노산 서열을 직접 변형하는 데 성공했습니다. 비표준 아미노산과 화학적 태그(비오틴, 형광단백질)를 내인성 단백질에 정밀하게 통합한 것은 이번이 처음입니다. 이 연구 결과는 "세포 내 단백질 편집을 통해 내인성 단백질에 비정형 잔류물을 통합할 수 있다"라는 제목으로 사이언스지에 게재되었습니다.
서류 주소:
https://www.science.org/doi/10.1126/science.adr5499
또한, 중국과학원 유전학 및 발달생물학 연구소의 가오 차이샤(Gao Caixia)와 화중농업대학교(Huazhong Agricultural University)의 리궈톈(Li Guotian)이 이끄는 중국 및 국제 연구팀은 데이비드 베이커(David Baker) 팀의 단백질 설계 연구를 바탕으로 역접힘(inverse folding) 모델에 기반한 범용 단백질 공학 방법인 AiCE를 개발했습니다. 이 AI 기반 단백질 설계 전략을 활용하여 탈아미노효소와 뉴클레아제를 포함한 8가지 단백질 클래스를 성공적으로 최적화하고 새로운 염기 편집기를 개발했습니다. "구조적 및 진화적 제약을 통합한 역접힘 모델을 이용한 단백질 진화 진전(Advancing protein evolution with inverse folding models integrating structural and evolutionary constraints)"이라는 제목의 이 연구 논문은 Cell에 게재되었습니다.
서류 주소:
https://www.cell.com/cell/abstract/S0092-8674(25)00680-4
생세포 편집부터 신경 보호 요법, 당화 혁신부터 AI 기반 다중 사슬 설계까지, 생물 의학 분야에서 AI가 지속적으로 발전함에 따라 전 세계 연구진은 자연적으로 무질서한 단백질의 근본적인 생물 의학적 과제를 해결하기 위해 전례 없는 다양한 접근법을 활용하고 있습니다. 연구팀은 자연적으로 무질서한 영역을 표적으로 삼는 연구를 통해 암이나 알츠하이머병과 같은 질병에 대한 새로운 치료법을 개발할 것입니다.
참조 링크: