Command Palette
Search for a command to run...
Nature 저널에 게재된 연구에 따르면, 유전자 시퀀싱과 머신 러닝을 기반으로 한 폐수 역학 평가는 최대 4주 일찍 바이러스를 감지할 수 있습니다.

지난 몇 년 동안 전 세계 공중 보건 안보는 심각한 어려움에 직면해 왔습니다. 특히 COVID-19 팬데믹 발발 이후 더욱 그렇습니다. 이 바이러스의 병원체인 중증급성호흡기증후군코로나바이러스2(SARS-CoV-2)는 여러 우세한 변이체의 출현과 함께 계속해서 진화해 왔습니다. 이러한 변이체들은 감염 및 면역 반응 회피 능력이 다양하여 전염병 예방 및 통제의 어려움을 가중시키고 의료 시스템의 부담을 가중시킵니다.
공중보건 검사와 SARS-CoV-2 게놈 시퀀싱은 순환 변종을 포괄적으로 감지하는 중요한 수단입니다.그러나 이러한 유형의 임상 모니터링은 종종 많은 양의 실험실 자원에 크게 의존하며 개인이 검사에 적극적으로 참여해야 합니다.SARS-CoV-2 변이의 출현과 확산을 완벽하게 추적하는 것은 어렵습니다. 특히 의료 자원이 상대적으로 부족하거나 검사 의지가 낮은 지역에서는 임상 모니터링이 탐지 편향에 더 취약하여 예방 및 통제의 사각지대를 초래합니다.
보완적 접근 방식으로, 폐수 기반 역학(WBE)은 지역 사회 감염을 평가하기 위해 1940년대에 처음 제안된 이래로 질병 발생 경고에서 중요한 역할을 해왔습니다. WBE는 주로 폐수에서 인체가 배출하는 바이러스의 흔적을 분석하여 바이러스 구성과 역동적인 변화를 탐지하고 추적합니다.임상 모니터링과 비교했을 때, WBE는 개별적인 적극적인 검사에 의존하지 않고도 해당 지역의 집단 감염 상황을 객관적이고 편견 없이 반영할 수 있으며, 조기 경보를 달성하고 비용 효율성이 상당히 높습니다.
그러나 현재의 주류 폐수 모니터링 방법(선형 회귀에 기반한 Freyja 및 COJAC 등)은 여전히 한계가 있습니다.탐지는 알려진 변형(예: GISAID 또는 UshER 데이터베이스의 참조 시퀀스)의 돌연변이 패턴을 기반으로 해야 합니다.임상 문헌에 특성화되거나 포함되지 않은 새로운 변종이 나타나는 경우, 이를 정확하게 식별하는 것이 어려운 경우가 많으며, 이로 인해 WBE의 탐지 효율성이 어느 정도 제한됩니다.
이를 해결하기 위해 네바다대학교 라스베이거스 캠퍼스의 연구팀은 ICA-Var(변형의 독립 성분 분석)이라는 다변량 분석 방법을 제안했습니다.이 방법은 비지도 학습 프로세스 설계를 기반으로 하며 독립 성분 분석(ICA)을 사용하여 폐수 데이터에서 공변량 및 시간에 따라 진화하는 돌연변이 패턴을 추출합니다.더 빠르고 정확한 변종 감지가 가능합니다.
연구팀은 이 방법을 사용하여 2021년 말부터 2023년 사이에 델타 변종, 오미크론 변종, 재조합 XBB 변종을 정확하게 검출했습니다. 이 방법은 전염병 예방 및 통제에 대한 조기 경보를 위한 폐수 모니터링의 효과를 재확인할 뿐만 아니라 임상 모니터링이 없는 상황에서 바이러스 돌연변이와 확산을 종합적으로 추적할 수 있는 새로운 도구를 제공합니다.
관련 연구는 "게놈 시퀀싱과 머신 러닝을 통한 폐수에서 발견되는 SARS-CoV-2 변종의 조기 감지"라는 제목으로 Nature Communications에 게재되었습니다.
연구 하이라이트:
* 이 방법은 도시와 농촌 지역에서 바이러스 돌연변이의 시공간적 역학을 밝히고, 도시에서 농촌 지역으로의 바이러스 전파 법칙을 확인하며, 의료 접근성이 낮거나 임상 시퀀싱 데이터가 부족한 지역에 효과적이고 저렴한 변종 탐지 패러다임을 제공합니다.
* ICA-Var의 다변량 분석법은 현행 금본위 도구인 Freyja와 비교했을 때 유의미한 장점을 가지고 있으며, Delta, Omicron 및 최신 EG.5, HV.1, BA.2.86 변이의 검출 시간이 평균 1~4주 앞당겨짐

서류 주소:
https://www.nature.com/articles/s41467-025-61280-5
장기 다중 지점 데이터 수집
본 연구에서는 실험에 사용된 폐수 시료를 2021년 8월부터 2023년 11월까지 수집했습니다.네바다주 남부의 도시와 농촌 지역에서 3,659개의 폐수 샘플이 수집되었습니다.폐수 샘플을 채취한 후, 현장의 얼음 위에 놓고 처리할 때까지 냉장 보관하며, 보관 시간은 36시간을 넘지 않습니다.
핵산 추출 과정 중에,연구팀은 먼저 규제 요건에 따라 Promega Wizard Enviro Total Nucleic Kit(Cat. No. A2991)를 사용하여 폐수 샘플에서 핵산을 분리했습니다. 그런 다음 Promega 프로토콜을 수정하여 폐수를 프로테아제 용액으로 용해하고 Macherey-Nagel NucleoMag Beads(Cat. No. 744970)를 사용하여 유리 핵산을 결합했습니다. 10ng 이상의 RNA의 경우, 연구팀은 New England BioLabs LunaScript RT SuperMix Kit를 사용하여 첫 번째 가닥 cDNA를 합성했습니다.
시퀀싱 라이브러리 구축 및 시퀀싱,연구팀은 Paragon Genomics의 CleanPlex SARS-CoV-2 FLEX 패널을 사용하여 앰플리콘 시퀀싱 라이브러리를 구축한 다음, 300사이클 플로우 셀을 사용하여 Illumina NextSeq 500 또는 NextSeq 1000 플랫폼에서 시퀀싱했습니다.
시퀀싱 데이터 처리 측면에서,연구팀은 먼저 cutadapt 소프트웨어(버전 4.2)를 사용하여 시퀀싱 리드 쌍에서 Illumina 어댑터 시퀀스를 제거했습니다. 그런 다음 bwa mem 소프트웨어(버전 0.7.17-r1188)를 사용하여 시퀀싱 리드 쌍을 SARS-CoV-2 참조 유전체(NC_045512.2)에 매핑했습니다. 그런 다음 fgbio TrimPrimers 도구(버전 2.1.0, 하드 트리밍 모드)를 사용하여 정렬된 리드에서 Paragon Genomics CleanPlex SARS-CoV-2 FLEX 앰플리콘 프라이머 시퀀스를 제거했습니다. 마지막으로 iVar variants 소프트웨어(버전 1.4.1)를 사용하여 변이를 검출했고(2020년 원본 참조 유전체와 비교한 대립유전자 빈도 차이 기반), samtools 소프트웨어(버전 1.16.1)를 사용하여 유전체 커버리지와 리드 깊이를 계산했습니다.
중복된 샘플과 양성/음성 대조군을 제거한 후,나머지 2,684개 샘플은 품질 관리(QC) 분석에 사용되었습니다.엄격한 품질 관리 후, SARS-CoV-2 게놈의 80% 이상을 포함하고 시퀀싱 깊이가 50배인 폐수 샘플만 후속 분석을 위해 보관했습니다.
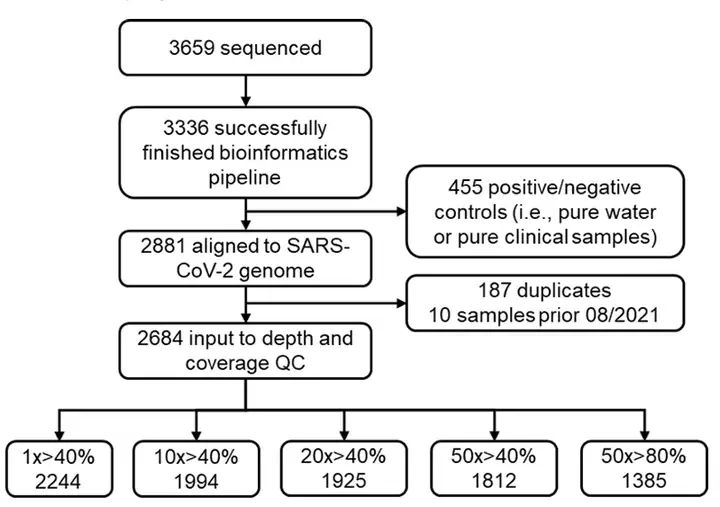
결정적인,이 연구에서는 고품질 샘플 1,385개를 사용했습니다.후속 분석을 위해 SARS-CoV-2 변종의 59,422개 돌연변이 부위를 다룹니다.
연구팀은 ICA-Var 방법의 효과성을 검증하기 위해 임상 데이터를 통제 및 참조 기준으로 사용하고, GISAID 데이터베이스에서 다운로드한 네바다주의 8,810건의 고커버리지 임상 SARS-CoV-2 시퀀스 데이터를 분석했습니다. 이 데이터는 2021년 9월부터 2023년 11월까지의 기간을 포함합니다.
ICA를 핵심으로 이중 회귀 분석법을 도입하여 COVID-19 검출을 위한 새로운 도구를 개발합니다.
ICA-Var의 핵심 프로세스는 다음과 같습니다.독립 성분 분석을 통해 폐수 샘플의 돌연변이 빈도를 처리하고 독립적인 공변이 돌연변이 패턴을 추출합니다.이러한 패턴은 아래 그림에서 볼 수 있듯이 이중 회귀를 통해 원본 샘플과 연관되어 바이러스 변종을 추적합니다.
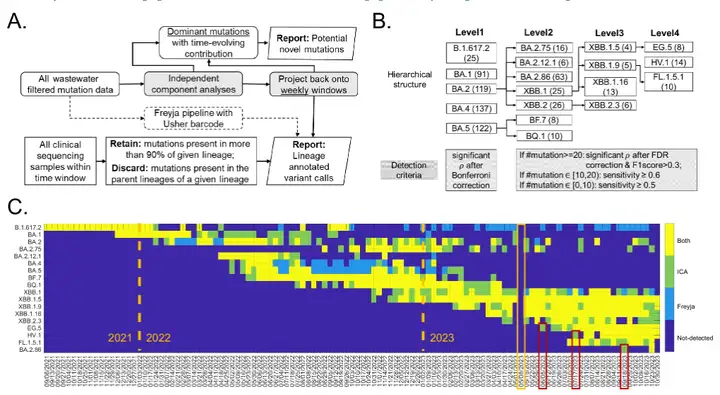
* 그림의 A는 독립성분분석 과정입니다.두 가지 매트릭스는 다음과 같습니다. 주간 SARS-CoV-2 계통 감지(아래쪽 행) 및 잠재적 신규 돌연변이(위쪽 행)
* 그림 B는 관심 있는 18개 변종의 계층 구조를 보여줍니다.각 변종의 주요 돌연변이 부위(즉, 계통 정의 부위)는 다음에서 가져왔습니다. http://covspectrum.org 주요 돌연변이의 수가 괄호 안에 표시되어 있고, 음영 처리된 상자는 제안된 워크플로에서 테스트할 기준을 나타내는 요약된 임상 데이터입니다.
*그림 C는 ICA-Var 방법과 최신 도구인 Freyja를 비교한 것입니다.새롭게 발견된 변종 EG.5, HV.1, BA.2.86의 경우, 빨간색 상자는 ICA-Var 검출 시점이 더 이른 것을 나타내고, 노란색 상자는 기술적 문제로 인해 폐수 샘플링이 수행되지 않은 주를 나타냅니다.
구체적으로, 폐수 샘플의 SARS-CoV-2 게놈 신호는 여러 변이체의 혼합으로 인한 결과이며 샘플 분해, 시퀀싱 오류 등의 영향을 받기 때문에 기존 방법으로는 단일 변이체의 특성을 직접 분석하기 어렵습니다.ICA-Var의 핵심 아이디어는 독립 성분 분석을 사용하는 것입니다.——이 블라인드 소스 분리 기술은 혼합 돌연변이 신호가 여러 "독립적인 소스"의 선형 조합이라고 가정하고 수학적 모델링을 사용하여 이러한 독립적인 패턴을 혼합된 데이터에서 분리합니다.
연구팀은 먼저 데이터를 사전 처리했습니다.폐수 샘플에서 추출한 SARS-CoV-2 유전체 시퀀싱 데이터에 대한 품질 관리를 수행하고, 저품질 리드와 노이즈가 있는 돌연변이를 필터링하여 행은 샘플을, 열은 돌연변이 부위를, 값은 샘플 내 각 부위의 돌연변이 빈도를 나타내는 "돌연변이 빈도 행렬"을 구축했습니다. 이후, 돌연변이 빈도 행렬에 대해 독립 성분 분석을 수행하여 혼합 신호를 독립적인 성분으로 분해했습니다. 각 성분은 "공변이 돌연변이 패턴", 즉 특정 변이의 특징적인 돌연변이 조합을 나타내며, 시간이 지남에 따라 샘플 전체에서 동시에 나타나거나 사라집니다.
여기,이 연구에서는 최소 설명 길이(MDL) 기준을 사용하여 독립 구성요소의 수를 결정하고 fastICA 알고리즘을 사용하여 독립 구성요소 분해를 수행했습니다.결과의 신뢰성을 보장하기 위해, 그들은 서로 다른 초기값을 사용하여 ICA 분석을 50번 반복하고, ICASSO 소프트웨어의 도움을 받아 각 실행에서 얻은 구성 요소를 클러스터링하고 시각화했으며, 마지막으로 밀집된 클러스터에 해당하는 신뢰할 수 있는 추정치만 소스 매트릭스로 유지했습니다.
이후, 주간변동상황을 더욱 정확히 파악하기 위해,연구팀은 이중 회귀 방법을 사용하여 독립 성분 분석에서 얻은 소스 행렬을 원래 샘플로 다시 투영했습니다.각 샘플의 각 독립 요소의 "기여도", 즉 샘플 내 변이체의 상대적 풍부도를 계산하여 출현 시간, 전염병 추세, 도시와 농촌의 분포 차이 등 시간과 공간에 따른 다양한 변이체의 역동적인 변화를 정량화합니다.
연구팀은 일반 선형 모형(GLM)에서 전체 표본 소스 행렬을 소스 회귀 변수 집합으로 사용하여 전체 표본 소스 행렬과 관련된 각 주별 샘플의 신호 분해 패턴을 구했습니다. 그런 다음, 각 주별 샘플의 신호 분해 패턴을 두 번째 GLM의 회귀 변수로 사용하여 전체 표본 소스 행렬과 여전히 관련된 주별 소스 행렬을 구했습니다. 이 과정을 통해 이중 공간을 구성하는 추정치 쌍이 생성되었고, 이를 통해 각 주별 샘플에서 원래 전체 표본 독립 성분 분석 소스 행렬에 대한 최적의 근사치를 제공했습니다.
마침내,연구팀은 분리된 독립 구성요소를 임상 시퀀싱 데이터의 알려진 변형과 비교하고 주석을 달았습니다.이를 통해 해당 변이 균주를 성공적으로 판별하거나 일치하지 않는 공변이 돌연변이 패턴을 걸러내어 새로운 변이 균주의 가능성을 경고할 수 있습니다.
ICA-Var 방법은 "사전 정의된 참조 변형 바코드"에 의존하는 기존 방법의 단점을 극복합니다.돌연변이의 공변이 패턴을 포착함으로써 기존 방법보다 더 일찍, 더 정확하게 새로운 변종을 식별할 수 있습니다.이 방법은 이중 회귀 분석과 결합하여 도시와 농촌 지역 전파의 차이와 돌연변이 부위의 시간적 진화를 보여줍니다. 요약하자면, ICA-Var는 COVID-19 검출에 있어 더욱 민감하고 포괄적이며 비용 효율적인 도구를 제공합니다.
탐지 효율은 현재의 금본위 도구인 Freyja를 능가하며 새로운 변종을 예측할 수 있는 잠재력을 가지고 있습니다.
연구팀은 ICA-Var의 성능을 검증하고 평가하기 위해, 폐수 내 SARS-CoV-2 계통의 상대적 풍부도를 추정하는 도구인 현행 표준 도구인 Freyja와 비교했습니다. Freyja는 계통을 정의하는 돌연변이로 구성된 "바코드" 라이브러리를 사용하여 알려진 모든 SARS-CoV-2 계통을 고유하게 식별하고, 심층 가중 최소절대편차 회귀법을 사용하여 계통 풍부도를 계산합니다.실험 결과, ICA-Var 다변량 분석 방법이 더 큰 장점을 가지고 있음이 확인되었습니다.
아래 그림과 같이, 모델 방법 및 아키텍처 섹션에서는 ICA-Var가 새로운 변종 EG.5, HV.1, BA.2.86을 어떻게 감지할 수 있는지 간략히 설명했으며, 이 섹션에서는 주요 내용을 확장합니다.
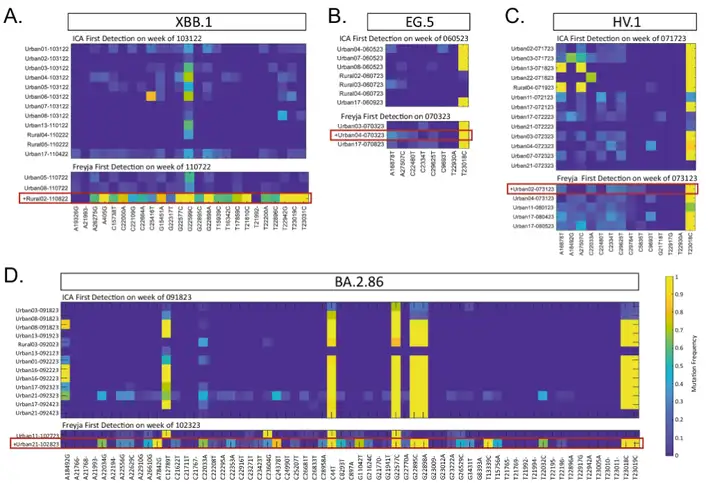
구체적으로 2022년에는ICA-Var는 Freyja보다 1주일 이상 일찍 BA.2, BA.4, BA.5, BF.7, BQ.1, XBB.1, XBB.1.5와 같은 변종을 감지하는 것으로 나타났습니다.EG.5 검출에서 ICA-Var는 6월 5일 주에 이 변이를 검출했지만, Freyja는 7월 3일이 되어서야 EG.5의 신호를 확인했습니다. 그날 폐수 샘플의 풍부도가 23.08%에 도달했고, EG.5의 8개 우성 돌연변이 부위 중 5개가 이미 검출되었습니다. 마찬가지로, XBB.1, HV.1, BA.2.86과 같은 변이의 경우에도ICA-Var도 Freyja보다 몇 주 일찍 발견되었습니다.
이는 ICA-Var가 "신뢰할 수 있지만 우성 돌연변이 부위의 유병률이 낮은" 여러 샘플의 정보를 통합했기 때문입니다.통계적 검정력을 향상시키고 조기 검출을 가능하게 합니다. 즉, 단일 샘플에서 우성 돌연변이의 높은 비율에 의존하지 않고, 여러 샘플에서 약한 신호를 수집하는 것만으로도 검출력을 향상시킬 수 있습니다. 이와 대조적으로, Freyja는 검출을 완료하기 위해 우성 돌연변이 부위를 명확하게 보여주는 최소 하나의 개별 샘플이 필요합니다. 이는 또한 단일 샘플에서 충분히 강한 돌연변이 신호에 더 의존하며, 약하거나 분산된 신호에는 덜 민감함을 의미합니다.
이 실험은 도시와 농촌 표본에서 변이의 역동적인 추세를 더욱 자세히 조사했습니다. 연구팀은 2022년 초부터 네바다 남부 농촌 지역의 폐수 샘플을 시퀀싱하고 분석하여, 도시와 농촌의 포괄적인 역학적 비교를 수행했습니다. 이 연구에서는 도시와 농촌 샘플을 매주 별도로 분석했습니다.
연구 결과에 따르면, 우려되는 18가지 변이 중 ICA-Var와 Freyja 모두 농촌 지역 샘플보다 도시 폐수 샘플에서 16가지 SARS-CoV-2 변이를 먼저 검출했습니다. 이는 바이러스 변이가 일반적으로 도시에서 먼저 발견된 후 농촌 지역으로 확산됨을 시사합니다. 아래 그림과 같습니다.
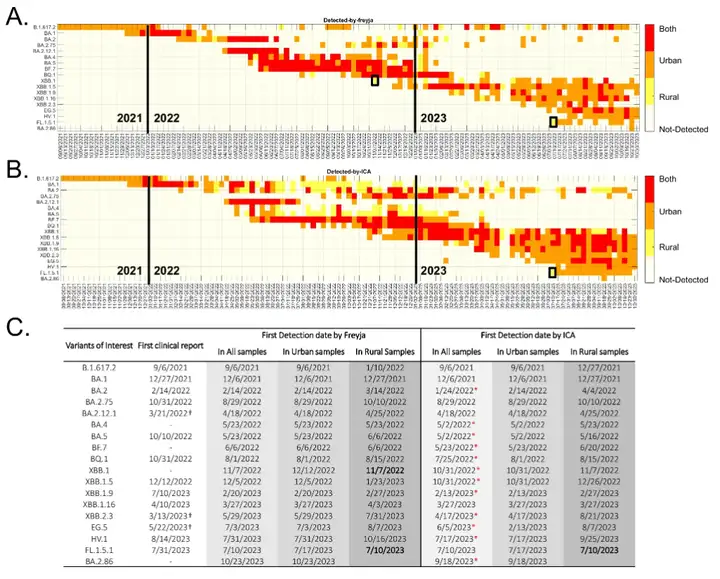
예외는 Freyja가 처음에 농촌 폐수 샘플에서 XBB.1을 감지한 반면, ICA-Var는 일주일 전에 도시 폐수 샘플에서 이 변종을 발견했다는 점입니다. 두 도구 모두 농촌 폐수 샘플에서 FL1.5.1을 발견했지만, 이 변종의 우성 돌연변이의 대체 대립 유전자의 빈도와 유병률은 같은 기간 동안 도시 폐수 샘플에서 훨씬 낮았습니다.
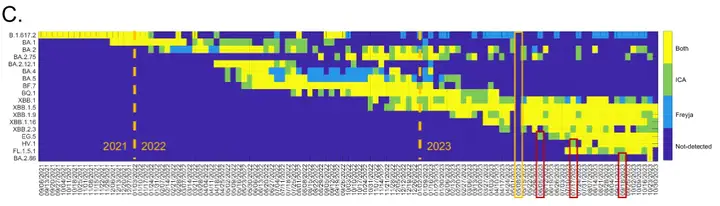
이 연구는 돌연변이 부위의 시간적 진화적 경향도 밝혀냈습니다. 연구팀은 2021년 8월부터 2023년 11월까지 시간적 진화적 기여도가 유의미한 177개 돌연변이 부위를 B.1.617.2, BA.1, XBB.1 변이의 우세 돌연변이 부위와 비교했습니다(아래 그림 참조).
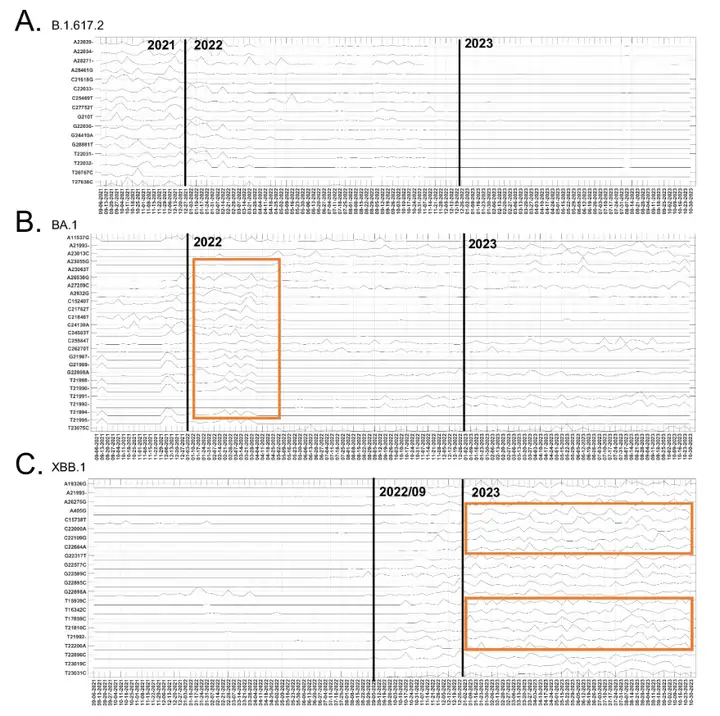
델타 변이체(B.1.617.2)의 25개 주요 돌연변이 부위 중 16개는 2021년 말에 기여도에 상당한 변동을 보인 후 2022년에 점진적으로 감소했습니다. 오미크론 하위 유형 BA.1의 관련 돌연변이의 기여도는 2021년 말에 상당히 증가하여 2022년 초에 정점을 찍었습니다. 일부 BA.1 돌연변이 부위의 기여도는 2023년에도 계속 변동했으며 XBB.1과 같은 다른 오미크론 하위 계통에서도 발견되었습니다. XBB.1 변이체의 25개 주요 돌연변이 중 22개는 상당한 시간적 동적 기여를 보였으며 2022년 9월 이후에 상당한 영향을 미쳤습니다. 여러 돌연변이 부위가 유사한 변동 패턴을 보였으며, 이는 공변이를 나타내며 XBB.1의 재조합 특성을 반영합니다.
이러한 분석은 ICA-Var에 의해 확인된 돌연변이 부위의 시간적 진화적 기여가 Delta, Omicron 및 XBB.1 변종의 임상적 소견과 일치함을 보여주었으며, ICA-Var 결과의 신뢰성을 더욱 잘 보여주고 새로운 변종의 출현으로 이어질 수 있는 새로운 돌연변이 패턴을 식별할 수 있는 잠재력을 보여줍니다.
본 실험은 이에 대한 상세한 검증을 수행했습니다. 연구팀은 15개 주요 변이체의 우세한 돌연변이 부위와 교차 비교하여 113개의 잠재적인 새로운 돌연변이 부위를 선별했습니다. 그런 다음 계층적 클러스터링 알고리즘을 사용하여 이러한 돌연변이 부위를 6개의 특징적인 클러스터로 분류했습니다. 아래 그림과 같습니다.

이러한 특징적인 클러스터 중 4개(클러스터 2~5)의 돌연변이 부위가 2023년 말에 나타난 변이체와 겹칩니다. 클러스터 1과 클러스터 6은 알려진 돌연변이 부위와 겹치는 돌연변이가 없습니다. 그중 클러스터 1의 돌연변이 부위는 2023년 8월 이후 명확한 공변이 패턴을 보였습니다. GISAID의 임상 시퀀싱 데이터 결과, 8개의 돌연변이 부위가 검증되었으며 임상 검체에서 보고 빈도가 낮은 것으로 나타났습니다. 따라서,이러한 돌연변이로 인해 새로운 코로나바이러스 변종이 출현할 가능성이 있으며, 이는 임상 검사를 통해 추가로 검증될 필요가 있습니다.면밀한 모니터링이 필요합니다.
머신 러닝을 기반으로 폐수 모니터링은 고품질 바이러스 예방 및 제어를 추진하기 위해 계속 발전하고 있습니다.
서두에서 언급했듯이, WBE는 새로운 방법이 아닙니다. 1940년대 초, 환경 바이러스학자들은 폐수에서 세포 배양 실험을 통해 폴리오바이러스를 얻는 것의 가치를 인식했습니다. 그 이후로 WBE는 지속적으로 개선되어 질병 발생을 조기에 경고하는 효과적인 도구로 자리 잡았습니다.COVID-19 팬데믹이 발발한 이후, WBE는 다시 한번 전염병 예방 및 통제에 긍정적인 역할을 수행했습니다.
예를 들어, 2023년 말, 스웨덴 연구팀은 하수 및 COVID-19 사례의 유전체 검사를 통합하여 새로운 SARS-CoV-2 BA.2.86 변이의 출현을 성공적으로 검출했다는 보고가 있었습니다. 또한, WBE를 신종 코로나바이러스 변이 검출에 더욱 효과적이고 적극적으로 활용하기 위해, 많은 연구소에서 WBE를 위한 더욱 비용 효율적인 도구를 제공하는 관련 모델을 개발하거나 개선해 왔습니다.
예를 들어, 칭화대학교, 허베이과학기술대학교, 톈진 생태환경모니터링센터의 연구진은 "폐수에서 SRAS-CoV-2 RNA 농축 및 검출 방법 검증"이라는 제목의 연구를 공동 발표했습니다. 이 연구는 초미세여과와 공유 친화성 수지 분리의 두 가지 농축 기법을 역전사 정량 PCR(RT-qPCR)과 역전사 디지털 PCR(RT-dPCR)의 두 가지 검출 방법과 비교하여 폐수 바이러스 모니터링 성능을 평가했습니다.
마침내,연구 결과에 따르면 역전사 디지털 PCR(RT-dPCR) 방법이 폐수에서 낮은 농도의 SARS-CoV-2 RNA를 검출하는 데 더 나은 선택인 것으로 나타났습니다.검출률이 더 높고 PCR 억제제에 대한 내성이 더 좋습니다.
* 서류 주소:
https://link.springer.com/article/10.1007/s10311-025-01843-6
또한, 캐나다 앨버타 대학교 병리학 및 진단검사의학과 싱팡 리 교수가 이끄는 연구팀은 "폐수 내 SARS-CoV-2 변이체의 정량화 및 감별 분석(Quantification and Differentiation of SARS-CoV-2 Variants in Wastewater for Surveillance)"이라는 제목의 연구를 발표했습니다. 연구팀은 이전에 임상 검체를 위해 개발된 감마(ABG) 및 델타 다중 RT-qPCR 검출법을 기반으로 오미크론 아변이체를 표적으로 삼고 그 변이체의 고유한 돌연변이를 활용했습니다.오미크론 변종의 5가지 주요 하위 계통을 구별할 수 있는 오미크론 삼중 RT-qPCR 분석법이 개발되었습니다.이 연구는 1년 동안 폐수 샘플에서 모든 오미크론 하위 변종을 탐지하고 식별하기 위해 단일 튜브 RT-qPCR 삼중 분석법을 사용한 최초의 연구입니다.
* 서류 주소:
https://pubs.acs.org/doi/10.1021/envhealth.3c00089
간단히 말해, 오늘날 세계는 심각한 공중 보건 및 안전 문제에 직면해 있으며, 인구 모니터링의 매우 효과적인 수단으로서 폐수 모니터링은 대체 불가능한 역할을 수행하고 있습니다. 기술의 지속적인 발전과 함께, 폐수 모니터링은 알려진 돌연변이 패턴을 기반으로 하는 조기 표적 탐지부터 전장 유전체 시퀀싱 및 미지의 병원체 식별의 획기적인 발전에 이르기까지 계속해서 발전할 것입니다. 지속적으로 민감도와 적용 범위가 향상됨에 따라, 폐수 모니터링은 전염병 경보, 추적 및 정책 수립에 더욱 정확하고 중요한 데이터를 제공하여 공중 보건 및 안전 방어선에 필수적인 보완책이 될 것입니다.
참고문헌:
1.https://www.nature.com/articles/s41467-025-61280-5
2.https://mp.weixin.qq.com/s/ZzzZt-uNNc5DsD-ib3Ww8g