Command Palette
Search for a command to run...
Stanford 등은 단백질 주쇄와 측쇄 정보를 동시에 처리하여 메시지 전달 신경망을 기반으로 한 전체 원자 구조 모델링을 달성했습니다.
단백질 측쇄 형태는 단백질 아미노산 잔기의 측쇄가 3차원 공간에서 특정하게 공간적으로 배열되는 것을 의미합니다. 단백질 측쇄 형태를 연구하면 단백질 구조와 기능 간의 관계를 이해하는 데 도움이 되며, 단백질 공학, 약물 설계 등 다양한 분야에서 큰 응용 가치를 지닙니다. 그러나 현재의 딥러닝 기반 단백질 서열 설계 방법은 주로 고정된 주쇄 단백질 서열 설계에 초점을 맞추고 있으며, 대부분의 경우 서열 생성 과정에서 단백질 측쇄 형태를 모델링할 수 없습니다.주요 측사슬 상호작용은 주쇄 구조와 알려진 아미노산 서열 태그에만 근거하여 추론되며, 단백질에서 단백질 측사슬 형태의 역할은 무시됩니다.
이러한 차이를 메우기 위해 캘리포니아 팔로 알토에 있는 스탠포드 대학과 Arc Institute의 팀이우리는 각 아미노산 잔기의 서열 동일성과 측쇄 구조를 명확하게 모델링할 수 있는 새로운 단백질 서열 설계 방법인 FAMPNN(Full-Atom MPNN)을 제안했습니다.이 모델은 그래프 신경망(GNN) 기반 메시지 전달 아키텍처를 사용하며, 개선된 MPNN(메시지 전달 신경망) 및 GVP(기하학적 벡터 퍼셉트론) 모듈을 결합하여 모든 원자 인코딩을 구현하고, 단백질 주쇄와 측쇄 정보를 동시에 처리할 수 있습니다. 연구에 따르면 FAMPNN은 모든 원자 구조를 명시적으로 모델링함으로써 단백질 서열 설계의 질과 실험 예측의 정확도를 크게 향상시킬 수 있습니다.
"FAMPNN을 이용한 전체 원자 단백질 서열 설계를 위한 측쇄 조절 및 모델링"이라는 제목의 연구 결과가 ICML 2025에 선정되었습니다.
연구 하이라이트:
* 우리는 잔류물의 이산적 서열 동일성과 연속적 측쇄 구조 모두의 라벨별 분포를 모델링하기 위해 교차 엔트로피와 확산 손실 목적을 결합하는 방법을 소개합니다.
* 우리는 공동 분포에서 샘플을 생성하기 위한 가벼운 반복 샘플링 방법을 구현하고 모든 원자 인코딩을 위해 개선된 MPNN 및 GVP 계층을 사용합니다.
* 연구에 따르면 FAMPNN은 모든 원자 구조를 명시적으로 모델링하여 시퀀스 설계 및 실험적 단백질 적합도 예측의 정확도를 효과적으로 향상시킬 수 있는 것으로 나타났습니다.

서류 주소:
더 많은 AI 프런티어 논문:
https://go.hyper.ai/owxf6
데이터 세트: 다양한 데이터 세트는 모델 학습 및 평가를 최적화합니다.
연구팀은 모델의 효과성과 신뢰성을 보장하기 위해 복잡한 다중 데이터 세트를 사용하여 훈련과 평가를 진행했습니다.
본 연구는 주로 CATH 4.2의 S40 데이터 세트를 사용했습니다.데이터 세트는 단백질 데이터 뱅크(PDB)에서 추출한 큐레이트된 도메인 세트이며, 상동성이 40%를 초과하는 중복 도메인을 제거하고 8:1:1의 비율로 훈련, 검증, 테스트 세트로 나눕니다.
PDB 데이터 세트는 전체 PDB 데이터베이스를 기반으로 구축되었으며 2021년 9월 30일 기준으로 발표된 구조를 포함합니다. 연구진은 단백질 사슬 수준에서 40%의 서열 상동성에 따라 단백질을 클러스터링하고 다중 사슬 단백질 예시를 우선 순위로 지정하여 다중 사슬 단백질을 설계하는 방법을 학습하는 모델을 훈련했습니다.
CASP13, 14, 15 데이터 세트는 주로 측쇄 패킹에서 모델의 성능을 평가하는 데 사용됩니다.연구팀은 MMseqs2 제안 검색을 사용하여 CASP13, 14, 15 데이터세트에서 40%보다 큰 상동성을 가진 모든 시퀀스를 훈련 및 검증 데이터세트에서 제거한 다음, 예측된 측사슬과 실제 측사슬 간의 평균 제곱근 평균 편차(RMSD)를 통해 측사슬 패킹 성능을 측정했습니다.
SKEMPlv2 데이터 세트는 단백질-단백질 결합 친화도에 대한 모델의 예측 능력을 평가하는 데 사용되었습니다.이 데이터 세트는 수백 가지 단백질-단백질 상호작용에서 수천 개의 서열 변형에 대한 실험적으로 측정된 결합 친화도를 정리한 것이며, 처리 후 최종 데이터 세트는 6,649개의 데이터 포인트입니다.
S669, Megascale 및 FireProtDB 데이터 세트는 단백질 안정성에 대한 모델의 제로샷 예측 능력을 평가하는 데 사용되었습니다.이 데이터 세트에는 다양한 천연 단백질(△△G)의 안정성 변화에 대한 실험 측정값이 포함되어 있으며, 이는 안정성 예측 변수에 널리 사용되는 벤치마크 데이터 세트입니다. Megascale 데이터 세트는 중복 제거된 버전의 데이터 세트입니다. 연구팀은 훈련 세트, 검증 세트, 테스트 세트를 단일 데이터 세트로 병합하여 최종적으로 298개의 서로 다른 단백질을 포함하는 272,712개의 실험 데이터 포인트를 포함하는 데이터 세트를 얻었습니다. FireProtDB 데이터 세트에는 100개의 고유 단백질의 3,438개 단일 돌연변이에 대한 자유 에너지 변화가 포함되어 있으며, 그중 3,420개의 예시가 처리 후 최종적으로 사용되었습니다. S669 데이터 세트에는 94개 단백질의 669개 단일 돌연변이에 대한 실험 측정값이 포함되어 있으며, 비표준 아미노산이 존재하기 때문에 4개의 변이체가 데이터 세트에서 제외되었습니다.
CR9114, CR6261 및 G6 데이터 세트는 항체-항원 결합 친화도를 예측하는 모델의 성능을 평가하는 데 사용되었습니다.그중 CR9114 데이터 세트는 16가지 아미노산 치환의 모든 가능한 조합을 포함하고 있습니다. CR6261 데이터 세트는 11가지 아미노산 치환의 모든 가능한 조합을 포함하고 있으며, 각각 총 65,536개와 2,048개의 서열을 가지고 있습니다. G6 데이터 세트는 VEGF-A에 결합하는 총 4,275개의 데이터 포인트를 가지고 있습니다.
단백질 서열과 측쇄 구조를 동시에 이해하는 지능형 도구
본 연구의 핵심 목표는 모델이 단백질 서열과 측쇄 형태를 동시에 학습할 수 있도록 하는 것입니다. 이를 위해 연구팀은 마스크드 랭귀지 모델링(MAM)을 사용하여 서열 일관성을 기반으로 FAMPNN을 학습시켰습니다.훈련은 범주형 교차 엔트로피 손실(시퀀스 예측용)과 확산 손실(측쇄 형태 예측용)을 결합하는 종단 간 방식으로 수행됩니다.이를 통해 모델은 부분적으로 알려진 서열과 측사슬 좌표를 기반으로 마스크된 서열과 해당 측사슬 형태를 동시에 복원할 수 있습니다.
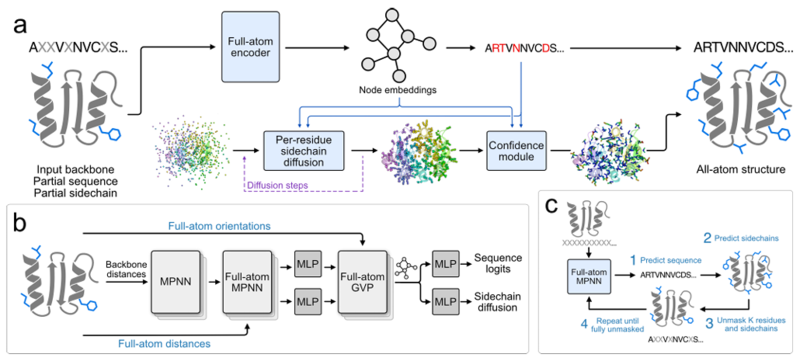
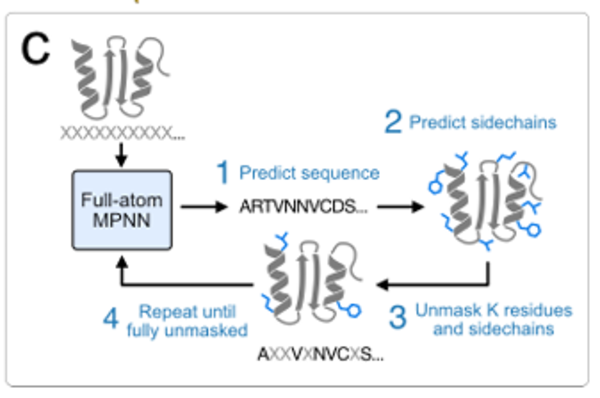
샘플링 측면에서는 MaskGIT과 유사한 반복 샘플링 전략이 도입되었습니다. 이 전략은 시퀀스와 사이드 체인이 완전히 마스크된 상태에서 시작하여, 완전한 시퀀스와 사이드 체인 구조를 얻을 때까지 일부 시퀀스와 사이드 체인 토큰의 마스크를 점진적으로 예측하고 제거하는 방식입니다. 아래 그림과 같습니다.
구체적인 설계에서 측사슬 좌표는 atom37 형식으로 표현됩니다.각 잔기는 4개의 주쇄 원자(N, Cα, C, O)와 33개의 측쇄 원자의 3D 좌표를 포함하여 37개의 원자를 갖는 37 x 3 고정 크기 행렬입니다. 특정 원자 유형이 존재하지 않는 측쇄의 경우, 고스트 원자(잔기의 Cα 위치로 설정됨)를 사용하여 이를 표현합니다. 이 방법은 아미노산에 따라 측쇄 원자의 수가 달라지는 문제를 해결합니다.
특징 추출의 핵심으로, 올-아톰 인코더는 인코딩을 위해 하이브리드 MPNN-GVP 아키텍처 그래프 신경망을 사용합니다.이 아키텍처는 세 가지 주요 구성 요소, 즉 불변 주 사슬 인코더, 불변 전체 원자 인코더, 그리고 등가 전체 원자 인코더로 구성됩니다. 처음 두 구성 요소는 ProteinMPNN 아키텍처를 기반으로 합니다. 불변 주 사슬 인코더는 주 사슬 구조만 인코딩하는 MPNN 인코더와 동일합니다. 불변 전체 원자 인코더는 주 사슬 인코더인 MPNN 인코더와 동일한 MPNN 디코더를 대체하지만, 특성 분석이 모든 원자로 확장됩니다. 마지막 구성 요소는 개선된 GVP(Global Value Proposition)를 사용하여 모델이 이전에 인코딩된 스칼라 값 원자 간 거리뿐만 아니라 벡터 값 원자 간 방향에 대해서도 추론할 수 있도록 합니다.
연구팀은 사이드 체인 좌표 생성 측면에서 토큰별 유클리드 확산 방법을 채택했습니다.이 방법의 핵심은 유클리드 확산 모델(EDM)을 사용하여 측쇄 원자 좌표의 연속적인 값을 생성하는 문제를 해결하는 것입니다. 목표는 주쇄 구조 및 주변 아미노산의 공간 배열과 일치하는 측쇄 구조를 생성하는 것입니다. 학습 과정에서는 먼저 실제 측쇄 좌표에 무작위 노이즈를 추가한 후, 모델이 노이즈를 제거하고 노이즈 수준과 알려진 정보를 기반으로 실제 좌표를 복원합니다. 추론 과정에서는 무작위 노이즈 좌표에서 시작하여 점진적으로 노이즈를 제거하고 실제 좌표에 가까운 측쇄 좌표를 생성합니다.
동시에 단백질의 전체 회전 및 번역이 측쇄 생성에 미치는 영향을 피하기 위해, 훈련 중에 측쇄 원자 좌표는 주쇄 원자를 기준으로 한 로컬 좌표계로 변환되고, 생성 후에는 다시 글로벌 좌표계로 변환됩니다.확산 모델의 입력에는 전체 원자 인코더에서 추출한 특징, 예측된 시퀀스 동일성 및 현재 노이즈 레벨이 포함됩니다.생성된 측사슬 좌표는 아래 그림에서 볼 수 있듯이 조인트 손실 함수에서도 모델 학습을 안내하는 데 사용됩니다.
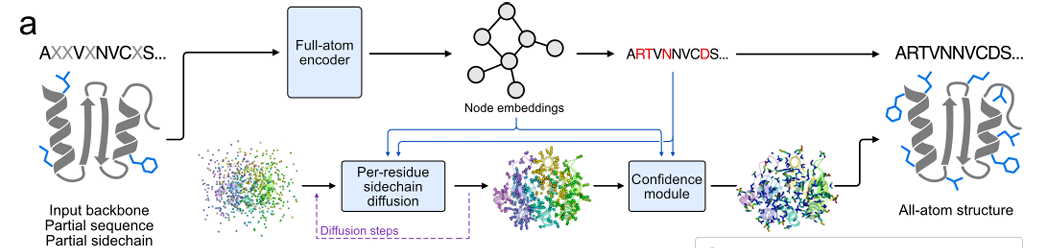
연구팀은 모델 예측 오류를 줄이고 정확도를 향상시키기 위해 사이드체인 패킹 오류(Predicted Sidechain Error, pSCE)를 예측하는 신뢰도 모듈도 설계했습니다. 구체적으로,이 모듈은 측쇄 원자의 실제 오차(생성된 좌표와 실제 좌표 사이의 거리)를 33개 간격으로 나눕니다.이 모델은 범주형 교차 엔트로피 손실(categorical cross entropy loss)을 사용하여 학습되므로, 생성 과정의 정보를 기반으로 각 원자 오류의 간격을 예측하고, 간격 확률의 기대값을 통해 최종 오류 추정값 pSCE를 얻을 수 있습니다. 이 모듈의 입력에는 전체 원자 인코더의 특성, 생성된 시퀀스 및 측쇄 좌표, 그리고 확산 과정의 노이즈 레벨이 포함됩니다. 출력 pSCE는 측쇄 패킹의 정확도를 효과적으로 반영할 수 있으며, 이는 고품질 설계 결과 선별 및 모델 해석성 향상에 도움이 되어 측쇄 구조 생성의 품질 평가 링크를 개선하는 데 도움이 됩니다.
실험 결과: 메인 체인만을 기반으로 한 모델보다 성능이 훨씬 우수합니다.
연구팀은 모델의 성능을 검증하고 정확한 평가를 위해 먼저 시퀀스 복구 및 자기 일관성 평가 실험을 수행하고, FAMPNN을 다른 방법들과 비교 평가했습니다. 구체적인 비교 대상은 아래 그림과 같습니다.
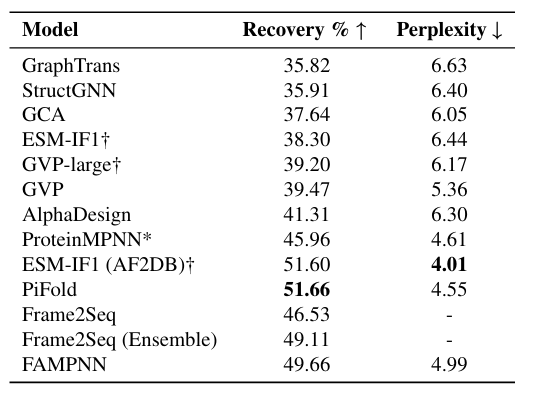
실험 결과FAMPNN은 단일 단계 시퀀스 복구 정확도 측면에서 현재 최첨단 방법을 능가하여 49.66%에 도달했습니다.비교해 보면 ProteinMPNN은 TP3T가 45.961에 불과하고 GVP는 TP3T가 39.471에 불과합니다.
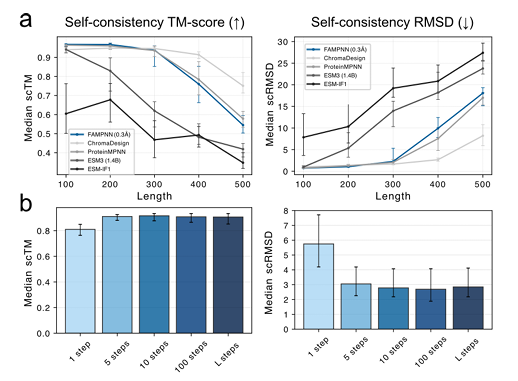
RFdiffusion을 기반으로 생성된 새로운 메인 체인에 대한 자기 일관성 평가에서,FAMPNN(0.3Å)은 scTM(구조적 유사성) 및 scRMSD(제곱평균제곱근 편차) 측면에서 ProteinMPNN과 비슷합니다.10단계 반복 샘플링은 높은 수준의 자기 일관성을 달성할 수 있으며, 이는 완전 자기회귀 방법보다 효율적입니다. 다음 그림에서 볼 수 있듯이:
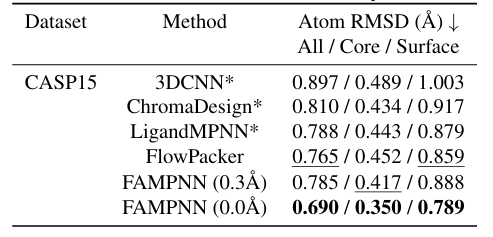
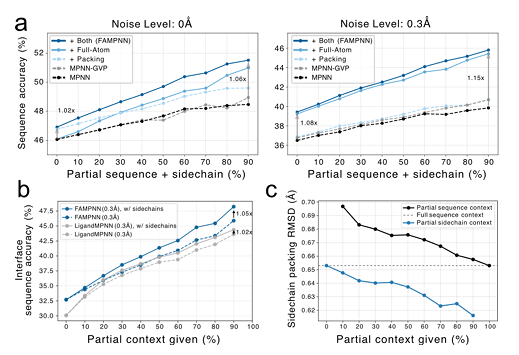
연구진은 사이드체인 패킹 측면에서 제안된 모델을 CASP13, 14, 15 데이터 세트의 다른 방법과 비교했습니다.실험 결과, CASP15 테스트 세트의 결정 구조 평가에서 FAMPNN(0.0Å)의 원자 RMSD(전체/핵심/표면)는 0.690/0.350/0.789Å로 다른 방법보다 우수합니다.그리고 각 원자의 오차 및 각 잔류물의 오차와 강한 상관관계를 보이며, 스피어만 상관계수는 각각 0.843과 0.780입니다. 아래 그림과 같습니다.
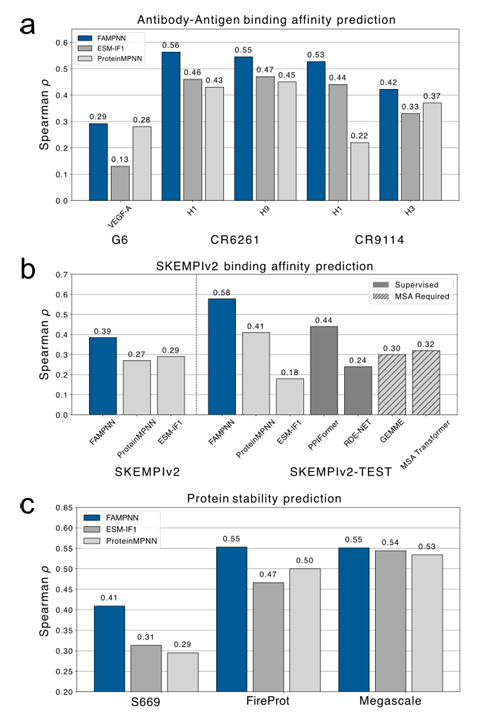
모든 원자 조건에서 단백질 적합도 평가에서,SKEMPlv2 데이터셋에서 FAMPNN은 비지도 학습 모델보다 유의미하게 우수한 성능을 보였고, 테스트 서브셋에서는 지도 학습 모델보다 더 뛰어난 성능을 보였으며, 영표본 예측에서 강력한 일반화 능력을 보여주었습니다. S669, Megascale, FireProtDB의 세 가지 안정성 데이터셋에서 FAMPNN은 ProteinMPNN과 ESM-IF보다 약간 더 나은 성능을 보였습니다. 항체-항원 결합 친화도 예측에서는 FAMPNN이 가장 진보된 비지도 학습 모델인 ProteinMPNN과 ESM-IF1보다 항상 우수한 성능을 보였습니다. 이는 단백질 안정성 및 단백질-단백질 상호작용 향상에 있어 FAMPNN의 유용성을 입증합니다. 다음 그림에서 볼 수 있듯이,
모든 원자 모델링이 시퀀스 설계 성능을 개선할 수 있는지 평가하는 실험에서,연구 결과, 측사슬 패킹 타겟과 전체 원자 조건 설정을 추가하면 시퀀스 정확도가 향상될 수 있는 것으로 나타났습니다.더욱이, FAMPNN과 기준 모델 모두 더 많은 구조 정보가 주입됨에 따라 성능이 향상될 것입니다. 단백질-단백질 계면에서 측쇄 상호작용 모델링은 더욱 중요하며, 서열과 함께 부분적인 측쇄 맥락을 제공하면 부분적인 서열 맥락만 제공하는 것보다 정확도를 크게 향상시킬 수 있습니다.
또한, LigandMPNN과 비교하여 FAMPNN은 측쇄 컨텍스트를 더욱 효과적으로 활용하고, 다양한 개수의 부분 서열 또는 측쇄 형태 컨텍스트를 기반으로 측쇄 패킹을 수행할 수 있습니다. 컨텍스트가 많을수록 패킹 정확도가 높아집니다. 아래 그림 c에서 볼 수 있듯이,
요약하자면, 위의 실험은 FAMPNN이 백본에만 기반한 모델과 비교했을 때 단백질 적합도 예측에 상당한 이점이 있음을 보여줍니다.
인공지능의 주도로 사이드체인 모델링 분야에서 학계가 번창하고 있습니다.
서두에서 언급했듯이, 측쇄 형태는 단백질 기능에 매우 중요합니다. 그러나 단백질의 주쇄가 결정된 후에도 여전히 다양한 측쇄 형태가 존재할 수 있기 때문에, 측쇄 형태 모델링과 연구는 반드시 극복해야 할 어려운 과제입니다. 이 연구 외에도 전 세계 여러 학술 연구 기관들이 최첨단 딥러닝 기술과 생물학적 지식을 활용하여 측쇄 모델링 연구를 추진하고 있습니다.
중국 복단대학교의 한 팀은 OPUS-Rota5라는 2단계 측사슬 모델링 방법을 제안했습니다.이 방법은 개선된 3D-Unet을 사용하여 지역 환경 특징을 포착합니다.각 잔류물에 대한 리간드 정보를 포함하고 RotaFormer 모듈을 사용하여 다양한 유형의 특징을 집계합니다. CAMEO 및 CASP15를 포함한 테스트 세트에 대한 평가 결과, OPUS-Rota5는 다른 주요 측쇄 모델링 방법보다 성능이 훨씬 뛰어납니다. 관련 연구는 ScienceDirect에 "OPUS-Rota5: 3D-Unet 및 RotaFormer를 이용한 매우 정확한 단백질 측쇄 모델링 방법"이라는 제목으로 게재되었습니다.
서류 주소:
https://www.sciencedirect.com/science/article/pii/S0969212624001266
베이징 대학의 한 팀은 GeoPacker라는 또 다른 방법을 제안했습니다.이 방법은 기하학적 딥 러닝과 ResNet을 결합하여 단백질 측사슬을 모델링합니다. GeoPacker는 회전 및 병진 불변 방식으로 원자 상호작용을 명시적으로 표현하여 상대적 위치 정보를 추출합니다. 측쇄 구조 예측 정확도 측면에서 GeoPacker는 에너지 함수 기반 최첨단 방법론보다 성능이 뛰어나며, 딥러닝 기반 방법인 DLPacker와 OPUS-Rota4보다 각각 약 10배와 700배 더 빠르게 실행되며, 예측 정확도도 비슷합니다. 관련 연구는 "GeoPacker: 단백질 측쇄 모델링을 위한 새로운 딥러닝 프레임워크"라는 제목으로 발표되었습니다.
서류 주소:
https://onlinelibrary.wiley.com/doi/epdf/10.1002/pro.4484
동시에 토론토 대학의 팀은 FlowPacker라는 모델을 제안했습니다.그 목적은 단백질의 알려진 아미노산 서열과 주쇄 구조를 바탕으로 측쇄의 구체적인 모양을 정확하게 예측하는 것입니다.기존의 고급 기법들과 비교했을 때, FlowPacker는 대부분의 지표에서 더 나은 성능을 보이고 더 빠르게 실행됩니다. 예를 들어, 각도 예측 오차, 예측 각도와 실제 값의 유사성, 원자 위치 편차 등에서 장점을 보입니다. 관련 연구는 "FlowPacker: 비틀림 유동 매칭을 이용한 단백질 측쇄 패킹"이라는 제목으로 발표되었습니다.
서류 주소:
일반적으로 측쇄 구조의 해독은 생명과학 분야 발전에 매우 중요합니다. 인공지능 기술의 지속적인 발전은 구조생물학과 계산생물학의 급속한 발전을 촉진했을 뿐만 아니라, 국내외 연구기관들이 눈부신 학문적 성과를 달성하는 데 기여했습니다. 이러한 성과가 실험실 수준에서 응용 분야로 확장되면, 생명과학 분야에 새로운 전기를 마련하고 생명과학 및 의학을 새로운 장으로 이끌 것으로 기대됩니다.