Command Palette
Search for a command to run...
MIT와 하버드는 단백질 언어 모델과 이미지 인페인팅 모델을 통합하여 단일 세포 단백질 국소화를 달성하기 위해 PUPS를 공동으로 제안했습니다.
단백질의 세포 내 국소화는 세포 구조에서 단백질의 특정 위치를 말합니다.이는 단백질이 생물학적 기능을 수행하는 데 필수적입니다. 간단한 예를 들자면, 세포를 거대한 기업체로 상상해 보세요. 세포핵, 미토콘드리아, 세포막 등이 대통령실, 발전부, 문지기 등 서로 다른 부서에 대응하고 있다면, 해당 단백질이 올바른 '부서'에 들어가야만 정상적으로 작동할 수 있습니다. 그렇지 않으면 암이나 알츠하이머병과 같은 특정 질병을 유발하게 됩니다. 따라서 단백질의 정확한 세포 내 위치를 알아내는 것은 생명과학의 핵심 과제 중 하나라고 할 수 있습니다.
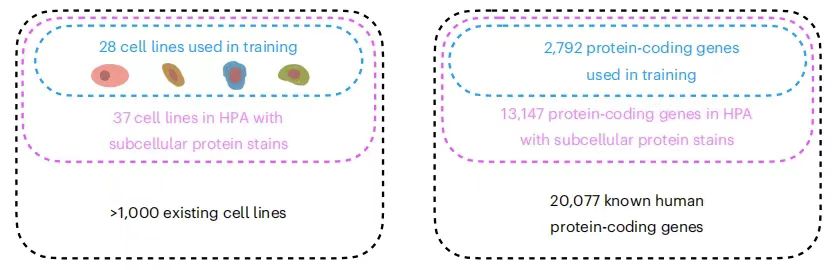
다양한 세포주에서 수천 개에 달하는 단백질의 공간적 국소화가 분석되었지만, 지금까지 측정된 단백질-세포주 조합의 수는 빙산의 일각에 불과합니다. 예를 들어, 현재 사용 가능한 가장 큰 세포 내 위치 데이터 세트는 다음과 같습니다.인간 단백질 지도(HPA)는 13,147개 유전자(알려진 인간 단백질 코딩 유전자의 65%에 해당)에 의해 인코딩된 단백질의 세포 내 위치를 제공합니다.그러나 전체 데이터 세트에는 37개의 세포주가 포함되어 있었고, 각 단백질은 그중 최대 3개에서만 측정되었습니다. 동시에 주류 실험 방법으로는 같은 세포 내에 존재하는 모든 단백질의 수를 동시에 검출하는 것이 어려워 복잡한 단백질 네트워크에 대한 포괄적인 분석을 심각하게 방해하고 실험의 복잡성과 오류 위험을 증가시킵니다.
또한, 단백질 국소화는 고정적이지 않으며 그 가변성은 세포주 간뿐만 아니라 동일 세포주 내의 개별 세포 간에도 발생합니다. 기존 데이터 맵에 기록된 단백질과 세포주 쌍은 특정 조건에서의 결과만을 반영합니다. 그러므로,기존의 결과조차 직접 적용하기 어렵고, 환경 변화에 따른 단백질 국소화에 대한 추가 탐색이 필요합니다.
단백질 세포 내 국소화 기술 방법의 한계와 생물학적 시스템의 복잡성 사이의 모순을 해결하기 위해 머신 러닝이 유망할 것으로 기대됩니다. 단백질 서열 기반 모델과 세포 이미지 기반 모델 등 오늘날 구축되어 성공적으로 적용된 모델은 어떤 측면에서는 좋은 성과를 보였지만, 단점도 매우 두드러진다. 전자는 세포 유형의 특정한 국소적 차이를 무시하고, 후자는 알려지지 않은 단백질에 대한 연구를 촉진하는 일반화 능력이 부족하다.
이를 고려하여,매사추세츠 공과대학과 하버드 대학의 연구팀은 단백질 서열과 세포 이미지를 결합하여 알려지지 않은 단백질의 세포 내 국소화에 대한 예측 프레임워크를 제안했으며, 이를 보이지 않는 단백질의 세포 내 국소화 예측(PUPS)이라고 명명했습니다. PUPS는 단백질 언어 모델과 이미지 페인팅 모델을 혁신적으로 결합하여 단백질 국소화를 예측함으로써 알려지지 않은 단백질 예측의 일반화 기능과 세포 변이성을 포착하는 세포 유형별 예측 기능을 병합할 수 있습니다. 실험 결과, 이 프레임워크는 훈련 데이터 세트 외부의 새로운 실험에서 단백질의 국소화를 정확하게 예측할 수 있고, 우수한 일반화 능력과 높은 정확도를 가지고 있으며, 뛰어난 응용 잠재력을 가지고 있음이 밝혀졌습니다.
"단일 세포에서 단백질의 세포 내 국소화 예측"이라는 제목의 연구 결과는 Nature Methods에 게재되었습니다.
연구 하이라이트:
* 제안된 연구는 단백질 서열과 세포 이미지를 사용하여 단백질 국소화를 예측함으로써 단백질 언어 모델과 이미지 렌더링 모델을 혁신적으로 결합하여 기존 계산 모델의 단점을 보완합니다.
* PUPS는 알려지지 않은 단백질 및 세포주에 일반화할 수 있으므로 세포주 간 및 세포주 내 개별 세포 간 단백질 국소화의 가변성을 평가하고 가변적 국소화를 갖는 단백질과 관련된 생물학적 과정을 식별할 수 있습니다.
* 훈련 데이터세트 외부의 새로운 실험에서도 PUPS는 뛰어난 응용 잠재력과 의학적 가치를 지닌 매우 정확한 예측 능력을 입증했습니다.

서류 주소:
데이터 세트: 가능한 가장 포괄적인 데이터로 신뢰할 수 있는 모델 구축
PUPS의 훈련 데이터 세트는 Human Protein Atlas(HPA)에서 가져왔습니다.연구팀은 가능한 한 많은 단백질 데이터를 수집하고 실험 분석의 포괄성을 보장하기 위해 16번째 버전의 HPA 데이터를 22번째 버전으로 집계했습니다. 다음 그림과 같이:
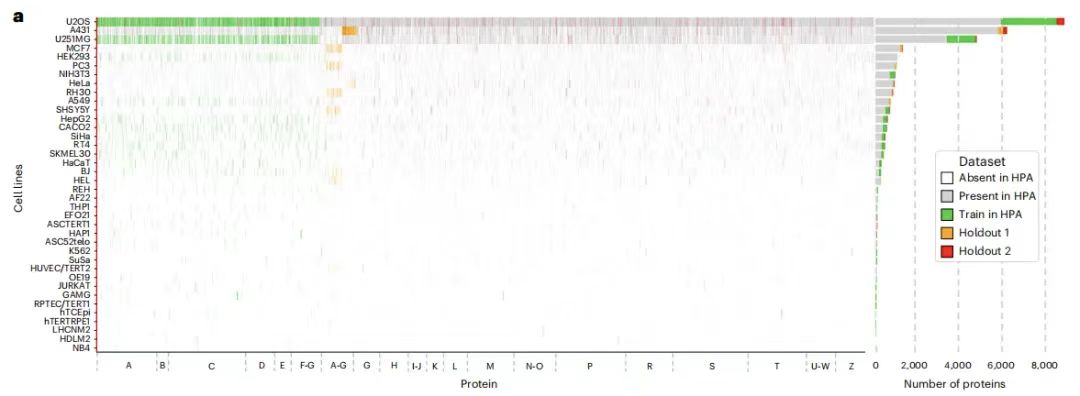
구체적으로, 훈련 데이터 세트에는 HPA의 37개 세포주에 있는 2,801개 유전자에 해당하는 총 8,086개의 단백질 변형을 포함하는 340,553개 세포 집단이 포함되어 있으며, 세포 이름이 AG로 시작합니다. 또한, 훈련 데이터 세트에는 IHO1, IMPAD1, INKA1, ISPD, ITPRID1, KIAA1211L, KIAA1324, LRATD1, SCYL3, TSPAN6을 포함한 10개의 추가 유전자가 포함되어 있습니다.
홀드아웃 데이터 세트는 두 부분으로 나뉩니다.한 부분은 예약된 데이터 세트 1입니다.여기에는 3,312개의 유전자(훈련 세트에 있는 2,801개 포함)에 해당하는 9,472개의 단백질 변형이 있는 36,552개의 세포가 포함되어 있으며, 유전자 이름도 AG로 시작하지만 다른 세포주에서 유래되었으며 훈련 세트와 겹치지 않습니다. 한편, 보류된 데이터 세트 1은 다시 두 부분으로 분할되어 각각 11,050개와 25,502개의 셀을 포함하는 평가 세트와 테스트 세트로 사용되었습니다.보관된 데이터 세트 2에는 515개의 유전자에 해당하는 24,007개의 세포가 포함되어 있습니다.이름은 AZ를 포함한 모든 알파벳으로 시작합니다. 총 556개의 단백질 변형이 있는데, 이는 훈련 세트와 예약된 데이터 세트1에 나타나지 않는 새로운 유전자 패밀리에서 유래되었으며 모델의 일반화 능력을 테스트하는 데 사용할 수 있습니다.
BJ 세포주 이미지가 훈련 세트와 홀드아웃 데이터 세트 1에 모두 보존되었다는 점에 유의하세요.
실험에 앞서 연구팀은 HPA에서 이미지를 사전 처리했는데, 이는 다음의 5단계로 구성되었습니다.
* 1단계,각 이미지는 4배로 다운샘플링되었으며, 계산량을 줄이고 고주파 노이즈를 제거하기 위해 최종 해상도는 픽셀당 0.32μm로 낮아졌습니다.
* 2단계,가우시안 블러(σ=5)와 오츠 임계값을 결합하여 세포핵의 대략적인 영역을 복잡한 배경에서 분리했습니다.
* 3단계, remove_small_holes 함수를 사용하여 300픽셀보다 작은 면적의 구멍을 제거한 다음, 이미지를 이진화하고 100픽셀보다 작은 노이즈 영역을 제거합니다.
* 4단계,각 세포핵의 중심을 계산하고, 중심을 단일 세포의 ROI로 하여 128×128 픽셀 영역을 잘라냈습니다.
* 5단계,강도 정규화와 노이즈 필터링을 통해 표준화된 데이터 분배가 달성되고 채널 간 간섭이 감소합니다.
모델 아키텍처: 단백질 서열과 이미지 표현을 결합하여 단백질 세포 내 위치를 예측합니다.
PUPS 모델은 주로 두 부분으로 구성됩니다.하나는 단백질의 아미노산 서열로부터 서열 표현을 학습하는 데 사용됩니다. 다른 하나는 표적 세포의 상징적 염색으로부터 이미지 표현을 학습하는 데 사용됩니다.그런 다음 단백질 서열 표현과 이미지 표현을 결합하여 표적 세포에서 단백질의 세포 내 위치를 예측합니다. 전자는 모델을 알려지지 않은 단백질 예측으로 일반화할 수 있게 하고, 후자는 모델이 단일 세포 수준의 변동성을 포착하고 세포 유형별 국소화 예측을 달성할 수 있게 합니다. 다음 그림과 같이:
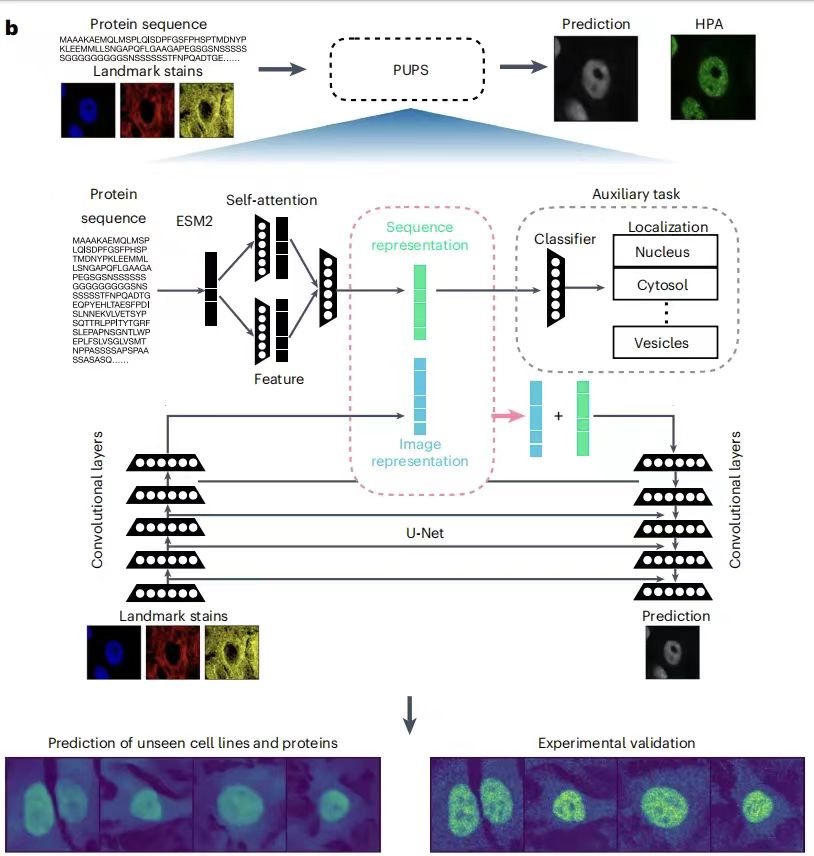
간단히 말해서,PUPS는 사전 학습된 ESM-2(Evolutionary Scale Modeling) 단백질 언어 모델을 사용하여 단백질 서열 특징을 추출하고, 합성 신경망을 사용하여 세포의 상징적 염색 이미지 특징을 학습합니다. 마지막으로, 두 가지 정보 부분을 결합하여 표적 세포에서 단백질의 위치를 예측합니다.모델의 모든 부분이 동시에 학습되므로 사전 작업의 분류 손실과 HPA에서 예측된 단백질 이미지와 실험적으로 측정된 단백질 이미지 간의 차이를 줄이는 데 도움이 됩니다. 모든 매개변수는 학습률이 1e-4인 Adam 최적화 도구를 사용하여 최적화됩니다.
단백질 언어 모델
PUPS는 언어 모델, 셀프 어텐션 계층, 보조 사전 학습 작업을 사용하여 시퀀스 표현을 학습한 다음 학습된 시퀀스 표현을 기반으로 단백질 국소화를 분류합니다.
구체적으로 연구팀은 사전 훈련된 ESM-2 모델에 N 말단 2,000개 아미노산 서열을 입력하여 특정 단백질 변이체의 초기 표현을 얻었고, 이를 통해 각 아미노산 잔기에 대해 1,280차원 벡터를 생성했으며, 잔기가 2,000개 미만인 변이체의 경우 패딩을 0으로 처리했습니다. 이러한 서열 길이 제한은 최대 수만 개의 잔여물에 이르는 서열 길이를 가진 소수의 단백질에 대한 편향된 예측을 피하기 위한 것입니다. 다음 그림과 같이:
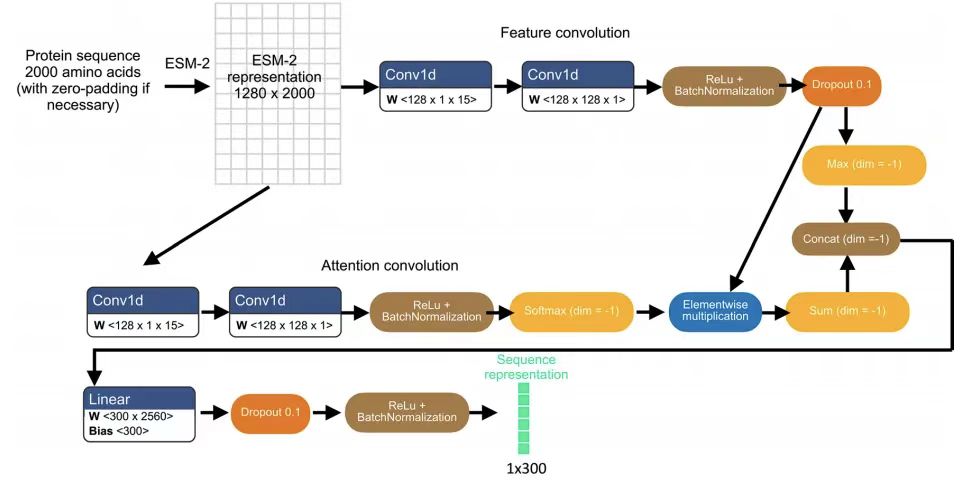
단백질 위치 예측을 위해 ESM-2 특성화를 적용하려면이후 연구팀은 분리 가능한 합성곱의 가벼운 주의 계층을 채택했습니다.ESM-2 표현에 적용하면 최종적으로 300차원 시퀀스 표현이 얻어진다. 이 단백질 서열 표현은 국소화 라벨을 예측하는 보조 사전 작업과 이미지 표현과 함께 단백질 이미지 예측에 모두 사용됩니다. 사전 작업에서는 단백질 서열 표현을 완전히 연결된 신경망 계층에 입력하여 29개의 세포 내 구획 국소화 라벨 간의 확률 분포를 나타내는 29차원 벡터를 입력한 다음, 시그모이드 활성화를 사용한 이진 교차 엔트로피 손실을 사용하여 사전 작업 출력을 HPA 주석이 달린 단백질 구획과 비교합니다.
이미지 렌더링 모델
각 세포의 이미지 입력에는 세포핵, 미세소관, 소포체 염색이라는 세 가지 상징적인 염색 이미지 채널이 포함되어 있습니다.크기는 3 x 128 x 128이고 중심은 핵 중심에 있습니다.
이미지 인코딩은 5개의 분리 가능한 합성곱 계층을 통해 달성됩니다.최종 치수 16 x 16 x 512. 각 합성곱 계층 뒤에는 leakyRelu 활성화 계층, 배치 정규화 계층, 2D 최대 풀링 계층이 이어집니다. 단백질 서열 표현은 세포 이미지 표현의 모든 공간 차원에 연결된 다음, 각 입력 채널에 대해 다른 가중치를 학습하는 U-Net 이미지 디코더에 입력됩니다. 또한, 이 모델의 공간 차원 가중치 메커니즘을 통해 이미지 표현의 각 공간 차원을 다른 가중치를 가진 시퀀스 표현과 결합할 수 있습니다.
디코더는 5개의 분리 가능한 합성곱 계층으로 구성됩니다.해당 세포에 대한 단백질 이미지 예측인 1 x 128 x 128 이미지 출력을 생성합니다. 그런 다음 이미지 분할 U-Net과 유사한 건너뛰기 연결이 랜드마크 염색의 이미지 표현을 생성하는 인코딩 계층과 동일한 깊이에서 단백질 이미지 예측을 생성하는 디코딩 계층 사이에 추가됩니다. 이 연구에서는 평균 제곱 오차 손실 함수를 사용하여 예측된 단백질 이미지와 실험적으로 측정된 단백질 이미지 간의 차이를 최소화하도록 모델을 훈련했습니다.
실험 결과: 단일 세포 수준에서 단백질의 정확한 세포 내 위치 파악 달성
연구팀은 모델의 실현 가능성과 효과성을 검증하기 위해 여러 가지 검증 실험을 제안했습니다. PUPS는 여러 작업에서 좋은 성능을 보였으며, 다중 모델 융합에서의 장점을 강조했습니다.
세포주 간 단백질 국소화의 변동성 예측
세포주 간 단백질 국소화 변동성을 정량화하는 PUPS의 성능을 평가하려면연구팀은 단백질 핵 비율을 계산하여 국소화 변동성을 정량화한 결과, 예측값이 실제 데이터와 높은 상관관계를 갖는 것을 발견했습니다.홀드아웃 1의 피어슨 상관계수는 0.794이고, 홀드아웃 2의 피어슨 상관계수는 0.878입니다. 다음 그림과 같이:
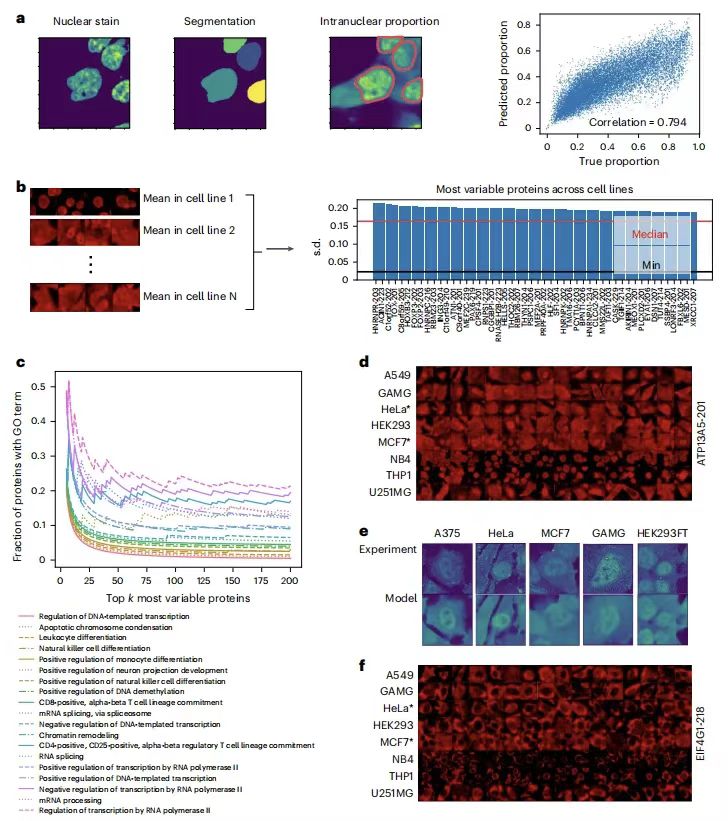
이후 추가 분석 결과, 세포주 간에 가장 큰 국소화 변화를 보인 단백질은 전사, 세포 분화, 크로마틴 조절과 같은 생물학적 과정과 관련이 있는 것으로 나타났습니다. ATP13A5의 실험적 검증을 통해 모델 예측의 정확성이 확인되었습니다. 또한,이 모델은 서명 염색을 통해 세포 형태의 차이를 포착하고 세포주 라벨 없이 단백질 국소화의 세포주 특이성을 추론할 수 있습니다.단백질 기능에 대한 세포 특이적 조절을 연구하는 새로운 방법을 제공합니다.
단일 세포 간 단백질 국소화 차이 예측
연구팀은 동일 세포주 내 단일 세포 간 단백질 국소화의 변동성을 PUPS가 예측할 수 있는 능력을 평가하기 위해 각 세포주 내 모든 단일 세포의 단백질 핵 비율의 분산을 계산했습니다.결과는 각 단백질-세포주 쌍에 대한 단일 세포 변동성 예측 순위가 실제 데이터와 매우 일치한다는 것을 보여주었습니다.예를 들어, Holdout 2에서 처음 500개의 고변이 쌍의 중복률은 60%를 초과했으며, 예측된 핵내 비율 분포는 실제 결과와 일치하여 예측 오류의 영향을 제거했습니다.
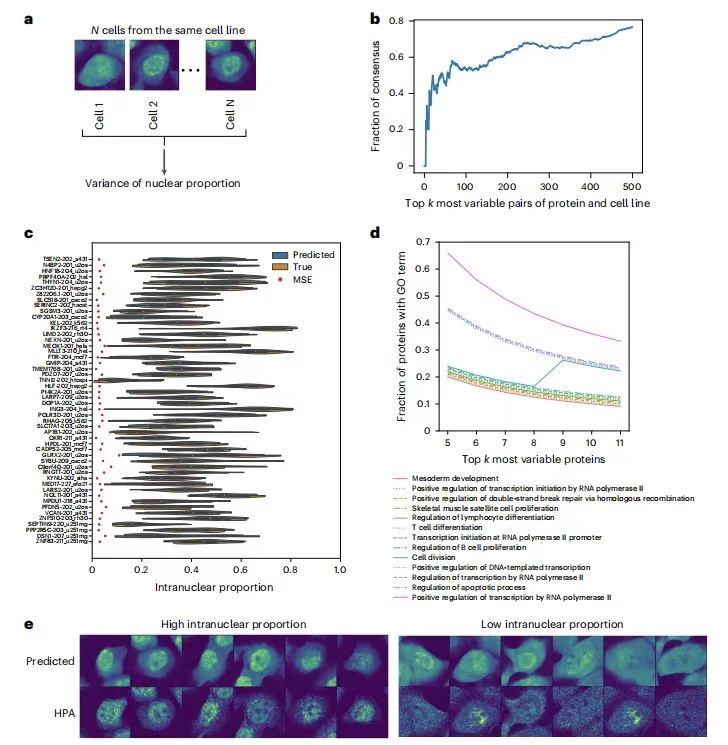
또한, 유전자 온톨로지(GO) 분석은 가변성이 높은 단백질이 세포 분열, 전사, 이중 가닥 절단 복구, 세포 사멸과 같은 과정과 관련이 있다는 것을 보여주었습니다. 또한,이 모델은 세포 표시 염색 이미지를 통해 형태적 특징을 포착하여 단일 세포 변동성이 무작위적일 뿐만 아니라 세포 형태적 특징과도 관련이 있음을 나타냅니다.단일 세포 이질성의 메커니즘을 설명하는 데 새로운 관점을 제공합니다.
훈련 데이터 외부의 새로운 실험에서 PUPS 검증
연구팀은 새로운 실험 환경에서 PUPS의 유비퀴틴화 능력이 단백질 국소화를 예측하는지 확인하기 위해 5개 세포주에서 검증을 위해 9개 단백질을 선택했습니다. 다음 그림과 같이:
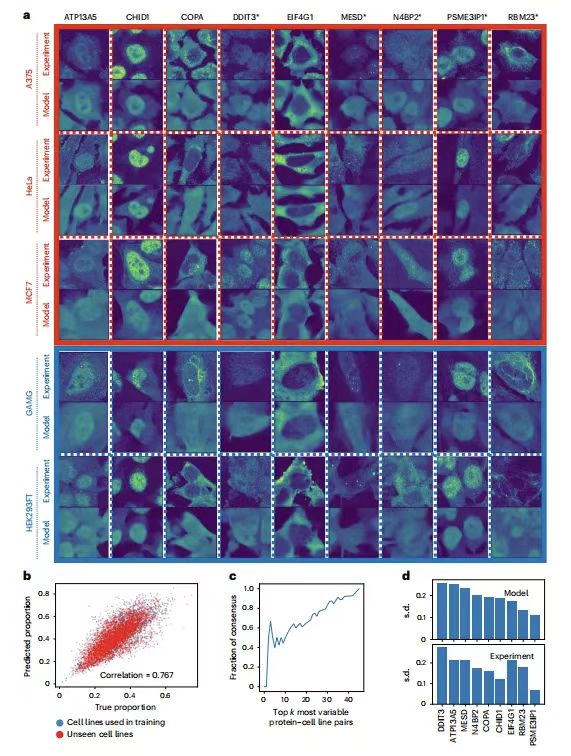
ATP13A5, CHID1, COPA, MESD 및 RBM23은 세포주 간에 가장 큰 변이를 보이는 단백질이며, 이들은 모두 서로 다른 GO 용어를 갖습니다. DDIT3와 N4BP2는 세포주 내 개별 세포에서 가장 큰 변이를 보이는 단백질입니다. EIF4G1과 PSME3IP1은 세포주 간 변이가 가장 적은 단백질로, 전자는 주로 핵 외부에 위치할 것으로 예상되고, 후자는 주로 핵 내부에 위치할 것으로 예상됩니다. 5개 세포주 중 A375를 제외한 나머지 HeLa, MCF7, GAMG 및 HEK293FT는 HPA에 포함됩니다.
결과는 다음과 같습니다PUPS로 예측한 단백질 이미지는 실험적으로 측정한 이미지와 시각적으로 유사합니다.예측된 단백질 이미지를 사용하여 계산된 각 단일 세포의 핵단백질 비율은 실험적으로 측정된 이미지로부터 계산된 비율과 밀접한 상관관계를 보이며, 피어슨 상관 계수는 0.767입니다. 이는 다음을 보여줍니다PUPS는 이전에 실험적으로 측정되지 않았거나 훈련 아틀라스에 사용되지 않았던 단백질의 국소화를 정량적으로 예측하는 데 사용될 수 있습니다.
PUPS는 의미 있는 단백질과 세포 표현을 학습합니다.
실험 결과, PUPS가 알려지지 않은 단백질과 세포주에서 단백질 국소화를 예측하는 능력은 단백질 서열과 세포 랜드마크 이미지의 의미 있는 표현을 학습하는 데서 비롯된다는 것이 입증되었습니다.
연구팀은 12,614개 유전자에 해당하는 40,622개 단백질 형태의 단백질 서열 표현을 매핑했으며, 유사한 위치를 가진 단백질은 유사한 서열 표현을 갖는 경향이 있었습니다. 연구팀은 이 모델이 의미 있는 단백질 서열 패턴을 식별하고 국소화를 예측할 수 있음을 더욱 입증하기 위해 위치적 샤플리 방법을 사용하여 특정 단백질의 각 아미노산 잔류물이 각 세포 구획의 라벨을 예측하는 데 얼마나 중요한지를 계산했습니다. 예를 들어, N4BP2 핵 국소화의 예측된 변동성을 성공적으로 설명했는데, 이는 CUE 도메인이 유비퀴틴 결합을 통해 세포 내 국소화를 변경할 수 있다는 보고와 일치합니다.
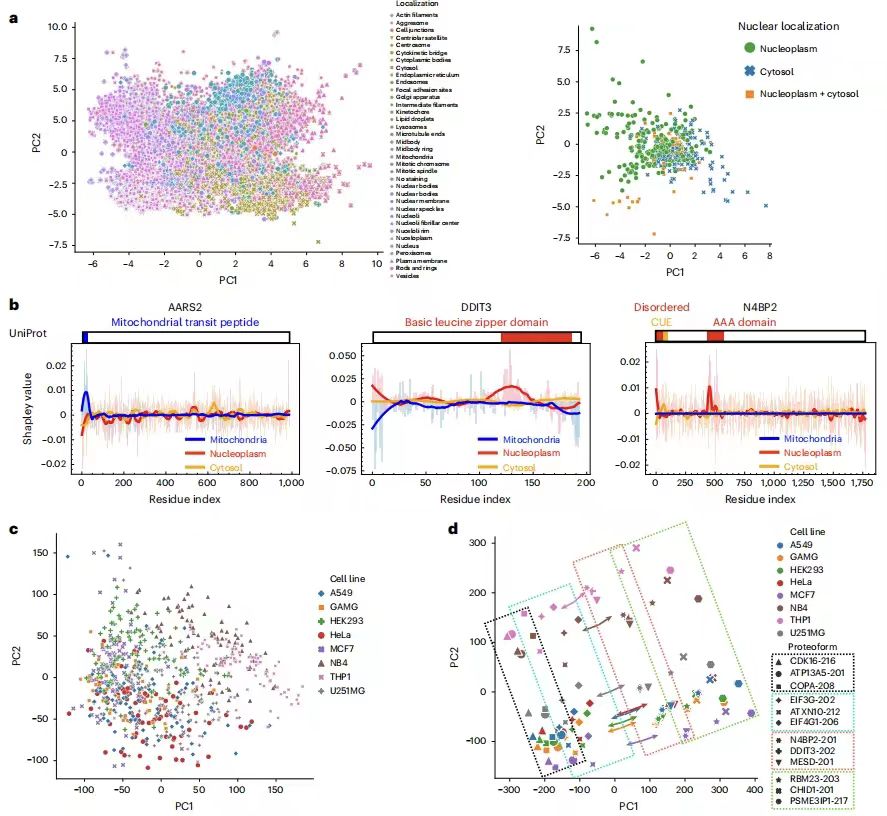
의미 있는 단백질 서열 모티프를 식별하는 것 외에도,연구팀은 PUPS가 세포 특징 염색을 통해 단일 세포의 의미 있는 표현을 학습한다는 것을 추가로 입증했습니다.랜드마크 염색에서 학습한 단일 세포 이미지 표현을 시각화하고, 세포 계통 레이블이 모델에 입력되지 않은 경우에도 동일한 세포 계통의 단일 세포가 유사한 이미지 표현을 갖는다는 것을 발견했습니다. 단백질과 세포 랜드마크 이미지를 함께 표현하면 세포주와 단백질을 분리할 수 있는 반면, 각 세포주 내의 다양한 단백질의 순서는 세포주 간에 유사합니다. 공동 표현 공간에서 각 세포주의 중심이 주어지면, 중심에서 특정 단백질로 가는 벡터는 모든 세포주에 걸쳐 대부분 평행합니다. 즉, 시퀀스 표현이 주어지면 특정 단백질에 대한 이미지를 예측하려면 세포주에 관계없이 표현 공간에서 같은 방향으로 이동해야 합니다.이는 PUPS가 단백질과 세포 이미지의 의미 있는 표현을 학습하여 알려지지 않은 단백질과 세포주로 일반화하는 능력을 설명합니다.
또한,PUPS는 또한 질병을 유발하는 돌연변이가 단백질 국소화에 미치는 영향을 예측할 수 있습니다.예를 들어, 핵에 인코딩된 미토콘드리아 단백질 SDHD와 ETHE1에 대한 돌연변이 연구는 SDHD 돌연변이가 핵 국소화 비율의 증가로 이어진다는 것을 보여주었는데, 이는 질병에서 핵 게놈 불안정성의 메커니즘과 일치합니다. ETHE1 돌연변이는 세포질 국소화 비율의 증가를 보여주는데, 이는 알려진 핵-세포질 왕복 이상과 관련이 있습니다. 이러한 결과는 PUPS가 국소화에 대한 서열 변이의 영향을 분석함으로써 질병 메커니즘을 연구하기 위한 새로운 단서를 제공할 수 있음을 시사합니다.
단백질 세포 내 위치 예측을 위한 새로운 솔루션
위에서 언급했듯이, 단백질의 세포 내 위치 예측은 생물정보학과 생물학 연구 모두에서 매우 중요합니다. PUPS는 다중 모드 정보를 통합하는 방법을 제공하여 이 분야 연구에 큰 진전을 가져왔습니다. 동시에 수십 년간의 개발을 거쳐 이 분야의 연구는 매우 다양한 결과를 낳았습니다.
아일랜드 더블린 대학교 연구팀은 Computational and Structural Biotechology 저널에 연구 결과를 발표했습니다.이 논문에서는 시퀀스 기반, 주석 기반, 하이브리드 및 메타 예측 기반 방법을 포함하여 단백질 세포 내 국소화 예측을 위한 다양한 계산 방법을 소개합니다. 또한 이 논문에서는 진핵생물, 원핵생물, 바이러스 및 다양한 범주별로 세포 내 위치 예측 도구를 분류하고 소개합니다.진핵생물 예측 도구로는 mLASSO-Hum, DeepPSL 등이 있고, 원핵생물 예측 도구로는 PRED-LIPO 등이 있습니다. 본 연구에서는 7개 주요 분야와 28개 하위 범주를 포괄하는 머신러닝과 딥러닝의 분류 지도를 설계하여 단일 범주 및 다중 범주 예측 도구에 대한 분류 체계를 제공하여 사용자가 방법과 예측 도구를 쉽게 찾을 수 있도록 했습니다. 해당 논문은 "단백질 세포 내 위치 예측 도구"라는 제목으로 출판되었습니다.
* 서류 주소:
https://www.sciencedirect.com/science/article/pii/S2001037024001156
4월 12일, 푸단대학교 생물의학연구소의 양리 연구팀과 상하이 인공지능연구소의 둥난칭 연구팀이 공동으로 "단백질 세포 내 국소화를 위한 심층 생성 모델"이라는 제목의 연구 논문을 Briefings in Bioinformatics 저널에 온라인 게재했습니다.이 연구에서는 또한 ESM2 단백질 언어 모델과 U-Net 프레임워크를 기반으로 다중 모드 처리 기능을 갖춘 생성적 딥 러닝 모델 deepGPS를 개발했습니다.
보도에 따르면, deepGPS는 단백질 서열과 세포핵 이미지를 입력으로 받아 단백질 국소화에 대한 텍스트 레이블과 분포 이미지를 생성할 수 있습니다. 이는 단백질 세포 내 국소화 예측을 지원하는 새로운 "텍스트-이미지" 다중 모드 모델입니다.
* 서류 주소:
https://doi.org/10.1093/bib/bbaf152
인공지능과 생물학 연구의 통합이 가속화됨에 따라, 이와 관련된 혁신적인 실험이 끊임없이 등장하여 점차 기존 방법의 단점을 깨고 '두 가지의 장점을 모두 취한' 또는 심지어 '완벽한' 성과를 달성함으로써 생물정보학의 급속한 발전을 촉진하고 있습니다.