효소 반응 속도 매개변수 예측, 병목 현상 식별... 중국과학원 선전선진기술연구소의 뤄샤오저우는 효소 분야에서 AI의 혁신적 응용을 공유했습니다.

단백질은 생명의 초석으로서 생명 활동에 중요한 역할을 합니다. 그 구조와 기능에 대한 연구는 혁신적인 약물 개발, 합성 생물학, 효소 생산 및 기타 분야에 매우 중요합니다. 그러나 전통적인 단백질 설계는 많은 어려움에 직면해 있습니다. 단백질 구조는 복잡하고 서열 공간은 방대합니다. 전문가의 경험과 고성능 스크리닝에 의존하는 설계 방법은 시간과 노동력이 많이 소요될 뿐만 아니라 성공률을 보장하기 어렵습니다.
오늘날 과학을 위한 AI는 글로벌 인공지능 개발의 새로운 영역으로 자리 잡았으며, 과학 연구의 패러다임을 근본적으로 바꾸고 단백질 설계 분야에 엄청난 변화를 가져오고 있습니다. 특히 AlphaFold와 같은 혁신적인 결과가 나온 이후, 관련 연구가 점차 대중의 관심을 끌면서 더 많은 주목을 받게 되었습니다. 동시에 국내외의 더욱 뛰어난 팀이 이에 헌신하고 기술, 응용 등 다양한 측면에서 어려움을 해결하도록 더욱 장려했습니다.
중국과학원 선전선진기술연구소의 연구원인 뤄샤오저우 교수가 그 중 한 명입니다. 그는 이전에 합성생물학에 집중했습니다. 그는 2019년 중국으로 돌아온 후 AI 단백질 연구에 전념하기 시작했습니다. 최근 중국 상하이 교통대학교에서 주최한 "미래는 여기 있다" AI 단백질 설계 서밋에서 뤄샤오저우 교수는 "인공지능 기반 효소 공학"이라는 주제에 대한 자신의 견해를 공유했습니다. 효소 설계에서 다중 모드 학습 및 생성 AI의 잠재적 응용 분야를 탐색합니다.효소 공학 분야에서 AI의 혁신적인 응용 분야와 실무 방식은 UniKP 프레임워크와 ProEnsemble 머신과 같은 다양한 관점에서 설명됩니다.

HyperAI는 원래 의도를 훼손하지 않으면서 심도 있는 공유를 구성하고 요약했습니다. 다음은 연설의 주요 내용을 요약한 것입니다.
자동화된 플랫폼 구축, AI가 단백질 문제를 해결한다
천연물은 광범위한 공급원, 풍부한 구조, 다양한 활성을 특징으로 하는 약재의 보고입니다. 그러나 천연자원으로부터 천연물을 추출하는 전통적인 방법은 비효율적이며, 순수한 화학 합성은 수율이 낮을 뿐만 아니라 많은 양의 독성 및 유해 시약을 사용해야 합니다. 예를 들어, 아르테미시닌은 원래 Artemisia annua에서 추출되었지만 화학 합성 과정에서 많은 문제에 직면했습니다. 이후, 사카로미세스 세레비시아에서 여러 유전자를 조절하여 아르테미시닌의 발현이 이루어졌습니다. 이 획기적인 발견을 통해 우리는 생합성의 잠재력을 확인하게 되었고, 그래서 저는 생물학 분야 연구에 집중하기 시작했습니다. 또한 효소 변형 분야에서는 데이터 부족으로 인해 연구 진행이 심각하게 제한될 것입니다. 이 문제는 우리에게 데이터의 중요성을 깨닫게 해주었고, 저는 이후 AI 연구의 기반을 마련하기 위해 자동화와 데이터 플랫폼을 구축하는 데 전념하고 있습니다.
생명의 기본 분자인 핵산, 저분자 지질, 탄수화물, 대사산물, 이온, 물 및 기타 물질은 모두 단백질에서 생성됩니다. 이러한 특징을 바탕으로 저는 2019년 중국으로 돌아온 후 단백질 분야에 연구를 집중하였고, 세 가지 과학적 의문을 제기했습니다. 첫째, 단백질의 활성과 기능을 서열만으로 직접 예측할 수 있을까요? 두 번째는 사람들이 필요로 하는 단백질을 수요에 따라 생성하거나 진화시키는 것이 가능한가? 세 번째는 보편적이고 표준화된 전략에 따라 효소나 균주를 최적화하는 것이 가능한가?
UniKP 프레임워크는 효소 특성을 더 잘 예측합니다.
교과서에는 다음과 같이 기술되어 있습니다: 단백질의 1차 서열은 3차 구조와 기능을 결정하며, 1차 서열에는 기능 정보가 포함되어야 합니다. 그러므로 시퀀스를 추출하는 방법은 매우 중요합니다. AlphaFold에서 영감을 얻어, 저희 팀은 서열을 통해 단백질 기능을 예측하는 방법을 탐구하기 시작했습니다. 우리 연구에서는 기존 표현 방법과 머신 러닝 기능을 통합하여 통합 모델을 구축하기 위해 Transformer 아키텍처를 도입했습니다.융합 특징과 통합 모델을 기반으로 하는 펩타이드 및 단백질 기능 예측 프레임워크는 8개의 관련 예측 작업에서 SOTA 성능을 달성하여 펩타이드 및 단백질 기능을 정확하게 예측했습니다.항균 펩타이드 등 항균 활성 물질의 스크리닝 과정을 가속화하고 실험 비용을 절감합니다.
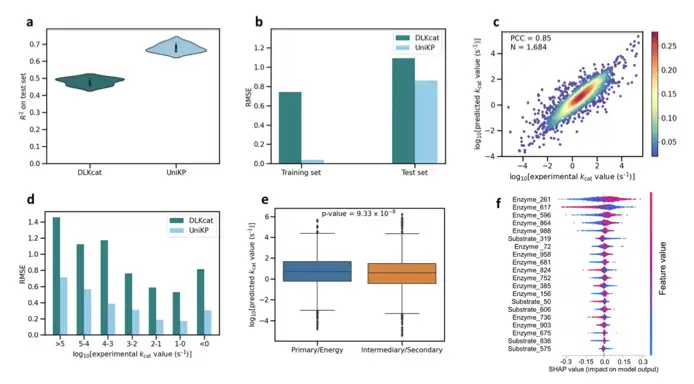
이후 연구팀은 UniKP 프레임워크를 사용하여 Transformer 임베딩 효소 매개변수 예측 도구를 기반으로 효소의 속성을 예측하려고 시도했습니다. ProtT5와 기존 SMILE Transformer 모델을 사용하여 시퀀스를 벡터화하고 이를 간단한 머신 러닝 모델과 결합하여 SOTA 결과를 얻습니다.
연구팀은 UniKP의 성능과 가치를 검증하기 위해 대표적인 데이터 세트 4개를 선택했습니다.
첫 번째는 DLkcat 데이터 세트입니다.연구진은 851개 생물체에서 7,822개의 고유한 단백질 서열과 2,672개의 고유한 기질을 포함하여 총 16,838개의 샘플을 검사했습니다. 데이터 세트는 9:1의 비율로 훈련 세트와 테스트 세트로 나뉩니다.
다음은 pH 및 온도 데이터 세트입니다.pH 데이터 세트에는 261개의 고유한 효소 시퀀스와 331개의 고유한 기질로 구성된 636개의 샘플이 포함되어 있습니다. 온도 데이터 세트에는 243개의 고유한 효소 서열과 302개의 고유한 기질로 구성된 572개의 샘플이 포함되어 있습니다. 데이터 세트는 8:2의 비율로 훈련 세트와 테스트 세트로 나뉩니다.
세 번째는 Michaelis 상수(Km) 데이터 세트입니다.여기에는 효소 서열, 기질 분자 지문 및 해당 Km 값을 포함한 11,722개의 샘플이 포함되어 있습니다. 데이터 세트는 8:2의 비율로 훈련 세트와 테스트 세트로 나뉩니다.
네 번째는 kcat/Km 데이터 세트입니다.효소 서열, 기질 구조 및 해당 kcat/Km 값으로 구성된 910개의 샘플이 포함되어 있습니다.
UniKP는 kcat 예측에서 기존 모델보다 상당히 우수하고, 처음으로 kcat/Km 예측을 달성한 것으로 검증되었습니다.예를 들어 kcat을 살펴보면, 가장 큰 공개 데이터 집합에서 결정 계수는 현재 SOTA 결과보다 20퍼센트 포인트 더 높습니다. 동시에, 다양한 데이터 세트 분할, 다양한 간격 분할, 다양한 효소 범주 분할 등 여러 작업에서도 상당히 더 나은 성능을 보입니다.
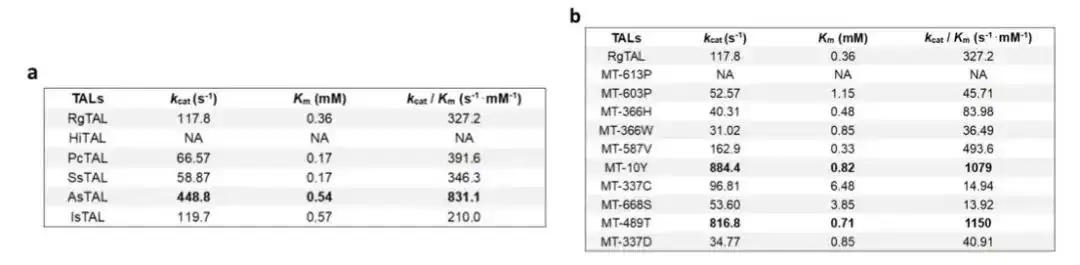
이 아키텍처를 사용하여 우리는 1,000개의 Blast 시퀀스에서 지금까지 가장 높은 효소 활성을 가진 야생형 TAL 효소를 찾았고, 단일 부위 돌연변이를 예측하여 효소 활성이 더 높은 돌연변이를 얻었으며, 이를 통해 효소 엔지니어링 프로세스를 크게 가속화했습니다.
또한, 단백질의 열 안정성을 목표로, 우리는 단백질 서열에 기반한 호열성 단백질 예측 모델인 Thermal Finer를 제안했습니다. 이 모델은 3개의 분류 데이터 세트에 대해 SOTA 성능을 달성했으며, 단백질 서열에 기반한 해당 최적 촉매 온도(회귀)를 처음으로 예측했습니다. 즉, 우리는 최초로 단백질 서열로부터 최적 온도를 직접 예측하는 데 성공했으며, 이는 효소 채굴과 진화에 대한 강력한 뒷받침을 제공합니다.
ProGPT-2 미세 조정을 통해 필요에 따라 단백질을 생성하거나 진화시킵니다.
현재 단백질 생산, 특히 효소 생산을 위한 두 가지 주요 모델이 있습니다.
* 생성적 적대 신경망(GAN): ProteinGAN
* 사전 학습된 생성적 대규모 언어 모델(LLM): ProtGPT2, ProGen
하지만,이러한 단백질 생성 도구는 모두 유사한 시퀀스를 생성한다는 문제가 있으며, 새로운 기능과 새로운 활성을 가진 효소를 생성하는 요구를 충족시킬 수 없습니다.이론적 분석에는 몇 가지 불합리한 측면도 있습니다. 첫째, 이미지의 픽셀 값은 연속적이어서 그래디언트 최적화에 더 적합합니다. 두 번째로, 텍스트(아미노산 서열)가 불연속적이고, 그래디언트 최적화는 임베딩을 업데이트하는 데 의미가 없으며 매우 비효율적입니다.
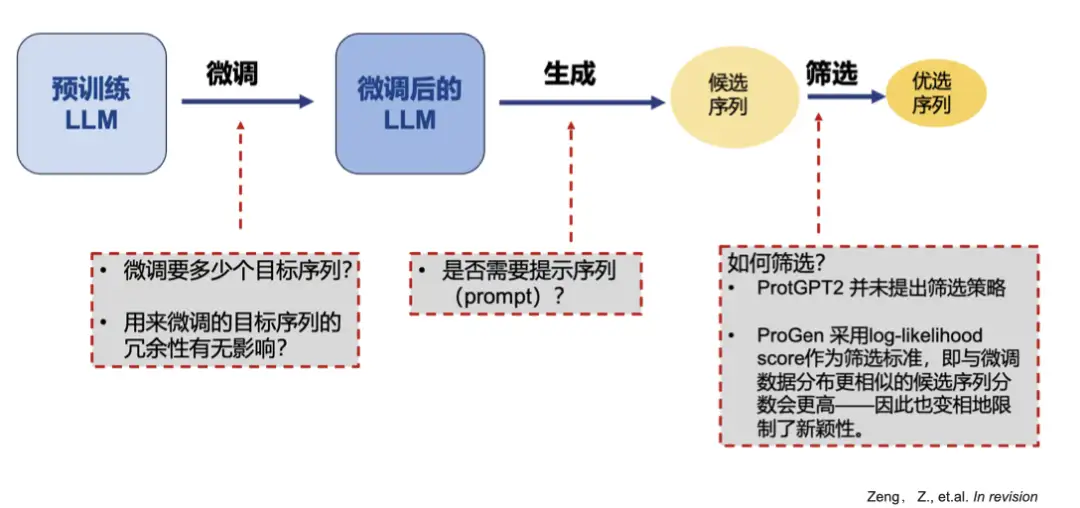
이러한 문제에 대해 우리는 기존 모델의 단점을 심층적으로 분석하고 새로운 최적화 프레임워크를 제안합니다.
저희 팀은 ProGPT-2를 미세 조정하고 CNN 신경망을 판별기로 사용하여 생성된 시퀀스를 필터링하고 우선순위를 지정했습니다. 실험을 통해 다음 사실이 밝혀졌습니다.시퀀스를 미세하게 조정하는 데는 2000개 또는 그 이하만 필요하며, 힌트 단어가 없는 생성된 시퀀스는 천연 효소에 더 가깝습니다. 동시에 중복된 데이터를 줄이면 생성된 시퀀스의 참신성이 향상될 수 있습니다.
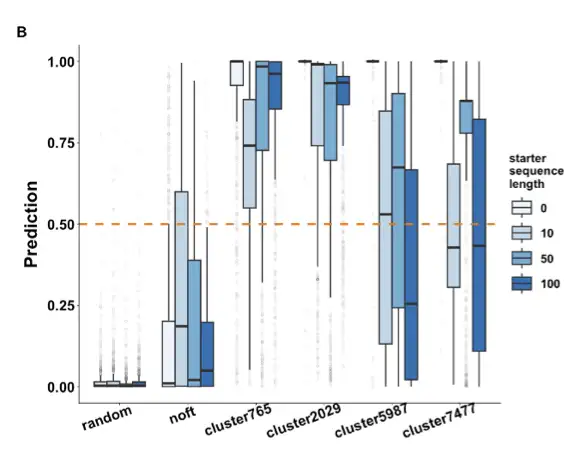
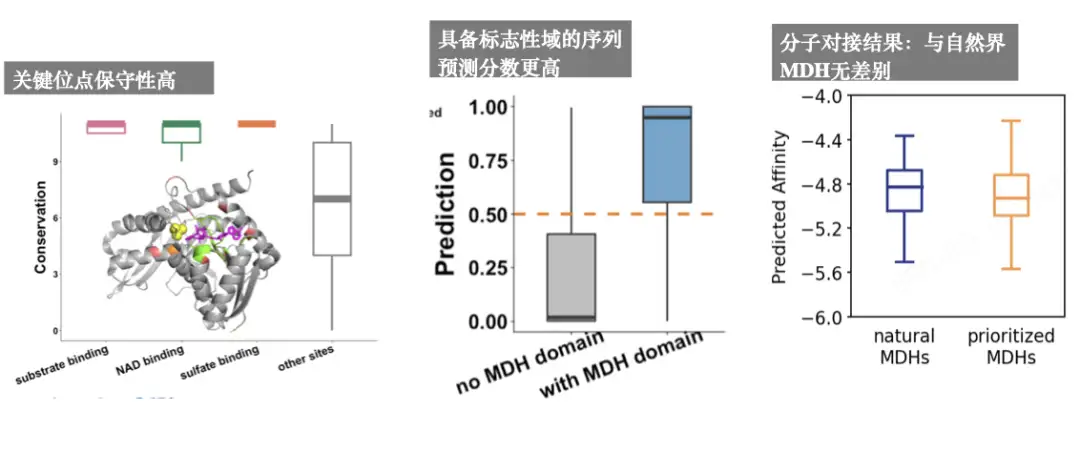
우리는 새로운 구조와 기능을 가진 새로운 효소를 원하므로 중복된 시퀀스는 필요하지 않습니다. 예측 항균 펩타이드를 통해 우리는 모델이 기본적으로 잘 작동한다는 것을 확인했고, 그런 다음 MDH 분석을 수행하여 다음 사실을 발견했습니다.주요 유적지는 높은 수준으로 보존되어 있습니다. 시그니처 도메인을 가진 사람들의 예측 점수가 더 높습니다. 분자 도킹 결과는 기본적으로 자연의 MDH와 동일합니다.다음 그림과 같이:
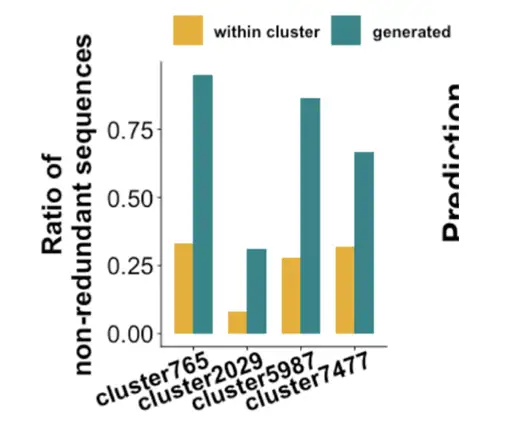
그런 다음 우리는 모델 이후에 생성된 다른 효소가 기능하는지 확인했습니다. ProteinGAN의 원래 데이터를 기준으로, 80%의 유사도를 가진 효소는 우선순위가 지정된 MDHs 모델을 적용한 후 40% 미만의 유사도에 도달할 수 있습니다. 자연에서 무작위로 선택한 10가지 효소와 비교해 보면 불용성, 발현 없음, 가용성 측면에서 기본적으로 동일하지만 여전히 매우 좋은 효소 활성을 가지고 있습니다. 다시 말해서,이 모델을 사용하여 우리 팀이 생성한 효소는 천연 효소와 유사성이 낮으며, 대부분 효소 활성을 가지고 있습니다.
ProEnsemble은 대사 병목 현상을 식별하고 효소 생산을 최적화합니다.
생합성 과정에서는 대사 경로에 있는 여러 효소의 낮은 촉매 효율성과 효소 간의 상호작용 효과 등 일련의 대사적 병목 현상으로 인해 최적화 과정이 복잡하고 불확실해집니다. 경로 효소의 과발현은 세포 성장과 생성물 발현에 영향을 미치는 경우가 많으며, 일부 효소는 부정적인 영향을 미칠 수 있습니다. 이러한 목적을 위해, 효소나 균주를 최적화하기 위한 보편적이고 표준화된 전략이 있는지 물었습니다.
먼저 과잉발현이 정말 나쁜 것인지 확인해 보겠습니다.연구팀은 특정 효소의 발현 수준을 인위적으로 감소시켜 인위적인 대사 병목 현상을 만들어냈고, 이를 통해 제어 가능한 진화적 공간을 얻었습니다.
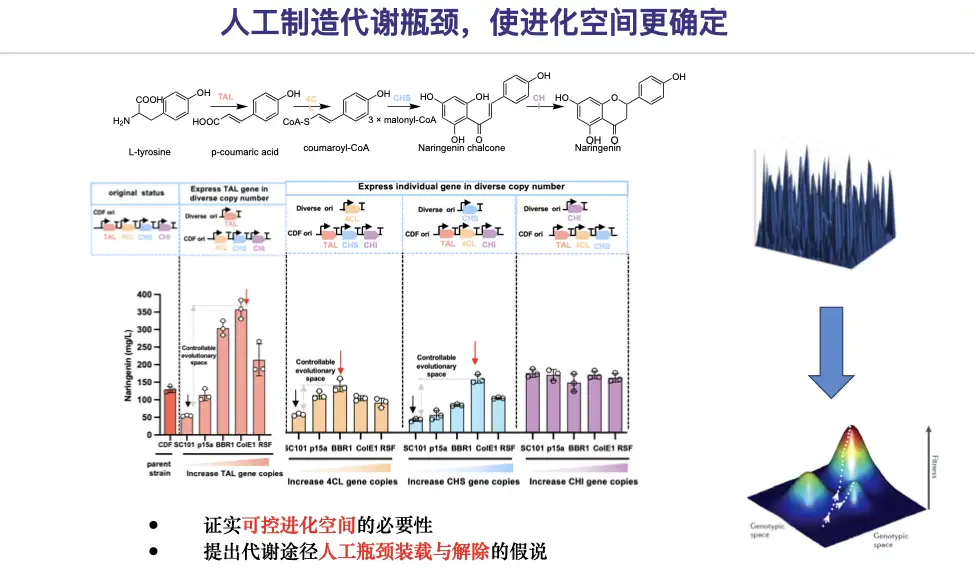
따라서 나린제닌을 예로 들어 경로 병목 현상 설계 및 제거 전략에 대한 솔루션을 제안합니다.
* 1단계에서는 자동화된 대규모 시설 플랫폼 기술을 사용하여 나린제닌 합성과 관련된 유전자가 낮은 수준(낮은 복제수 배경)으로 발현되도록 하여 나린제닌 합성을 위한 인위적인 대사 병목 현상을 구축합니다.
* 두 번째 단계에서는 후보 돌연변이체 4CL-11C1과 CHS-9H9를 선별하여 원래 돌연변이체와 비슷한 수준의 나린제닌을 생성했고, 이를 통해 나린제닌 경로의 병목 현상을 제거했습니다.
* 세 번째 단계에서는 AI를 이용한 프로모터 엔지니어링을 통해 단일 유전자 돌연변이체를 원래 경로로 다시 배치하고 대사 흐름을 균형 있게 조절합니다.
연구 결과는 다음과 같습니다.인공적인 병목 현상 생성 및 제거 전략을 통해 명확한 궤적 범위 내에서 대사 경로의 효율적인 진화가 가능해집니다.이는 또한 상위효과가 경로 진화의 경계를 제한할 수 있음을 추가로 확인시켜 줍니다.
이를 바탕으로 우리는 지침, 복제, 박테리아 스크리닝 테스트를 포함한 자동화된 프로세스를 설계했습니다.실험 결과, 성장, 선별, 제품 추출 측면에서 수동 작업과 유의미한 차이가 없는 것으로 나타났습니다.그러나 자동화 지원 대사 경로 진화 방법다중 효소 병렬 진화에 걸리는 시간이 대폭 단축되어, 한 라운드의 병렬 진화를 2주 안에 완료할 수 있습니다.
연구팀은 축적된 방대한 양의 데이터를 바탕으로 신진대사 증가를 최적화하는 머신러닝 통합 모델인 ProEnsemble을 개발했습니다. 실험 결과, 머신 러닝을 기반으로 한 통합 모델은 대사 경로를 균형 있게 조절하고 최적화되지 않은 모델에 비해 나린제닌 생산을 5.16배 증가시켰으며, 96웰 플레이트에서는 1.21g/L, 발효기에서는 3.65g/L에 도달하여 보고된 최고 수준에 도달했습니다. 단순히 핵심 합성 유전자를 과발현시킴으로써, 다양한 변형된 화합물 섀시의 생산이 문헌에 보고된 수준(대사 공학 전략의 도움)보다 더 높았습니다.
ProEnsemble의 학습 전략은 대사 병목 현상 식별-최적화 폐쇄 루프 시스템을 구축하여 현재 업계 수준보다 몇 배나 높은 고수율의 나린제닌 대장균 섀시를 성공적으로 개발했으며 복잡한 대사 네트워크의 균형을 위한 보편적 솔루션을 제공합니다.
산학연 협력을 촉진하는 대규모 자동화 플랫폼 구축
마지막으로 이러한 성과의 산업적 구현에 대해 소개하고자 합니다. 우리는 중국 선전에 있는 합성생물학 연구를 위한 주요 과학기술시설이라는 대규모 완전 자동화 플랫폼을 구축했습니다. 이 시설에는 설계 학습, 합성 테스트, 사용자 테스트 등 여러 플랫폼을 포괄하는 대규모 자동화 플랫폼이 포함되어 있습니다. 이 플랫폼은 강력한 기능을 갖추고 있으며, 클라우드에서 머신 러닝을 위한 표준화된 데이터 처리 및 실험 설계를 수행할 수 있습니다. 로봇은 실험 작업을 완료하는 데 도움을 줄 수 있습니다. 스펙트럼 준비 및 검출 속도가 빠르며, 단 10초 만에 하나의 샘플을 생성할 수 있어 고처리량 검출이 가능합니다.
또한, 이 플랫폼은 자동화된 지원 소프트웨어 설계를 제공하여 사용자가 구성 요소 라이브러리에서 필요한 구성 요소를 직접 선택하고 실험 지침을 생성할 수 있도록 합니다. 이제 우리는 많은 산업계와 학계와 협력하고 있습니다. 우리는 스트렙토마이세스 자동화의 전체 프로세스를 실현한 업계 최초의 플랫폼입니다. 우리는 모든 분들의 협조를 환영합니다.
Luo Xiaozhou 교수 소개
뤄샤오저우 교수는 중국과학원 선전선진기술연구소 합성생물학연구소의 연구원이자 박사과정 지도교수, 부소장입니다. 그는 국가중점인재프로젝트-청년프로젝트의 선정 전문가이자, 국가생물제조산업혁신센터의 CTO이며, 중국 선전시 합성생물학중점과학기술센터의 부수석공정엔지니어입니다.
그는 박사학위를 받았습니다. 2016년 스크립스 연구소에서 화학 박사 학위를 받았고(지도교수: 학자 피터 G. 슐츠), 그 후 캘리포니아 대학교 버클리에서 박사후 연구를 마쳤습니다(공동 지도교수: 학자 제이 D. 키슬링). 2019년에 그는 중국과학원 선전선진기술연구소에 합류했습니다. 그는 국가 청소년 인재 계획, 광둥성 우수 청년 학자, 선전시 우수 청년 학자로 선정되었습니다.
그의 연구는 효소의 지시적 진화, 단백질 공학, 고처리량 스크리닝, 천연 및 비천연 화합물의 총 생합성을 포함한 합성 생물학 분야에서 생물체의 생화학적 과정에 중점을 두고 있습니다. 그는 Nature Metabolism, Advanced Science, Nature Synthesis, Nature Communications, Angew에 20편의 논문을 게재했습니다. 화학 국제 Ed. 등 총 50여 편의 SCI 논문을 발표하였고, 30여 건의 특허를 출원하였으며, 6건의 특허를 승인 받았습니다.