Command Palette
Search for a command to run...
새로운 암 치료법이 개발될 수도 있습니다! 듀크 대학교는 PepPrCLIP을 사용하여 "치료 불가능한" 문제를 해결합니다.
2021년에 OpenAI는 혁신적인 CLIP(Contrastive Language-Image Pre-training) 모델을 출시했습니다. CLIP은 비지도 학습을 통해 추가적인 주석 정보가 필요 없이 이미지와 텍스트 간의 관계를 효과적으로 이해하고 연관시킬 수 있습니다.
몇 년 후, 생물의학 과학자 그룹은 이 아이디어에서 영감을 얻었습니다. CLIP이 이미지와 언어를 일치시키듯이, 같은 아이디어를 펩타이드와 단백질을 일치시키는 데에도 사용할 수 있지 않을까요?
대조적 언어-이미지 사전 학습을 통해 현실적인 이미지를 생성하는 OpenAI의 획기적인 연구를 활용하여,듀크 대학 생체공학과의 연구팀은 PepPrCLIP(CLIP을 통한 펩타이드 우선순위 지정) 파이프라인을 구축했습니다. 이 파이프라인은 이전에는 약물로 치료할 수 없었던 질병 유발 단백질에 결합하여 파괴할 수 있는 짧은 단백질(펩타이드)을 설계할 수 있습니다.표적 3D 구조를 사용하여 펩타이드를 생성하는 기존 플랫폼인 RFDiffusion과 비교했을 때 PepPrCLIP은 더 빠르고, 표적 단백질과 거의 항상 더 일치하는 펩타이드를 생성합니다. 연구진은 또한 PepPrCLIP이 선택한 "가이드 펩타이드"가 시험관 내에서 억제 펩타이드로 사용되거나 E3 유비퀴틴 리가제 도메인과 융합될 경우 강력하고 우수한 표적 결합 및 조절을 달성할 수 있음을 실험을 통해 추가로 검증했습니다.
관련 결과는 올해 1월 Science Advances에 "대조 언어 모델링을 통한 구조적으로 다양한 표적에 대한 펩타이드 결합제의 새로운 설계"라는 제목으로 게재되었습니다.
서류 주소:
https://www.science.org/doi/10.1126/sciadv.adr8638
관련 데이터세트 다운로드 주소:
https://go.hyper.ai/AT5m9
오픈소스 프로젝트인 "awesome-ai4s"는 200개 이상의 AI4S 논문 해석을 모아 방대한 데이터 세트와 도구를 제공합니다.
https://github.com/hyperai/awesome-ai4s
"치료 불가능한" 문제를 해결하는 새로운 접근 방식
질병을 치료하는 한 가지 방법은 질병을 유발하는 단백질을 특별히 표적으로 삼아 파괴할 수 있는 약물을 개발하는 것입니다. 때로 이러한 핵심 단백질은 마치 조심스럽게 접힌 종이학처럼 잘 정의된 구조를 가지고 있어서 기존의 소분자 치료제가 쉽게 결합할 수 있습니다.
그러나 80% 이상의 병원성 단백질은 무질서하고 얽혀 있는 "엉킨 덩어리"와 같아서 표준 치료법으로는 표면의 결합 부위를 찾아 작용하는 것이 거의 불가능합니다. 소위 "치료 불가능"이라는 말은 구조적, 기능적 특성으로 인해 약물 표적이 되기 어려운 전통적인 약물 개발 과정의 단백질을 설명하는 데 자주 사용됩니다.
공개된 정보에 따르면 약물 사용이 어려운 표적은 대체로 다음과 같은 특징을 갖고 있습니다.
* 잘 정의된 리간드 결합 포켓이 없고 확장되고 평평한 기능적 인터페이스가 있습니다.
* 표적 단백질의 기능을 가능하게 하는 특정 리간드가 부족함
* 표적은 질병 억제제로, 단백질 활동을 활성화하는 약물이 필요하므로 약물 개발이 더욱 어렵습니다.
* 약물 개발이 어려운 표적은 종종 복잡한 생리적 기능을 가지고 있어 약물 설계 및 개발이 더욱 어려워집니다.
* 약물 개발 전략의 한계.
이런 문제를 해결하기 위해 많은 연구자들은 펩타이드를 사용해 질병을 유발하는 단백질에 결합하고 분해하는 방법을 연구해 왔습니다. 펩타이드는 단백질의 소형 버전이기 때문에 결합을 위한 표면 주머니가 필요하지 않습니다. 그 대신, 펩타이드는 단백질의 다른 아미노산 서열에 결합될 수 있습니다.
하지만 이러한 접근 방식에도 한계가 있습니다. 기존의 "기성품" 결합제는 불안정하거나 지나치게 얽힌 단백질 구조에 부착되도록 설계되지 않았기 때문입니다. 과학자들이 새로운 결합 단백질을 개발하기 위해 열심히 노력하고 있지만, 이러한 접근 방식은 여전히 표적 단백질의 3차원 구조 정보를 매핑하는 데 의존하고 있으며, 이는 무질서한 표적에는 사용할 수 없습니다.
이 기사에서 소개한 듀크 대학 생체공학과 연구팀은 다른 접근 방식을 취했습니다. 그들은 병원성 단백질의 구조를 파악하려고 시도하는 대신 대규모 언어 모델(LLM)에서 영감을 얻어 PepPrCLIP을 구축했습니다. 첫 번째 구성 요소인 PepPr은 대규모의 자연 단백질 서열 라이브러리를 기반으로 학습된 생성 알고리즘을 사용하여 특정 기능을 갖춘 새로운 "가이드" 단백질을 설계합니다. 두 번째 구성 요소인 CLIP은 OpenAI에서 원래 개발한 알고리즘 프레임워크를 사용하여 이러한 펩타이드가 표적 단백질과 일치하는지 테스트하고 선별합니다.
CLIP 기반 펩타이드 우선순위 지정 프로세스 구축 - PepPrCLIP
PepPrCLIP은 어떻게 구성되었나요?
간단히 말해서, 연구진은 먼저 ESM-2 단백질 언어 모델(pLM)을 사용하여 실제 펩타이드 바인더 시퀀스의 임베딩에 가우시안 노이즈 교란을 수행하여 자연스러운 특성을 가진 후보 펩타이드 시퀀스를 생성했습니다. 그런 다음, 이러한 후보 펩타이드는 CLIP 기반 대조 학습 아키텍처를 통해 잠재 공간에서 스크리닝되어 보완적인 펩타이드-단백질 쌍을 공동 인코딩하는 모델을 학습했습니다. 마지막으로, 구축된 PepPrCLIP은 표적 서열에 결합할 수 있는 완전히 새로운 펩타이드 후보 서열을 선별하기 위해 세대-구별 프레임워크를 통합했습니다.
다음 그림은 PepPrCLIP 모델 학습의 구체적인 프로세스를 보여줍니다.
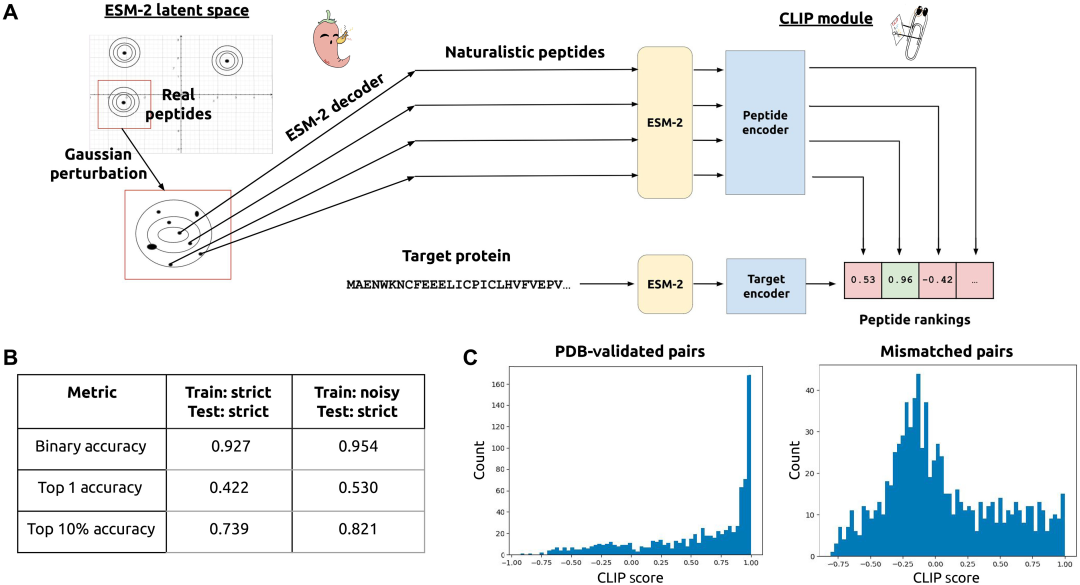
위 그림에서 보듯이, ESM-2에 삽입된 천연 펩타이드를 샘플링하여 가우시안 분포를 생성한 후, 이를 다시 아미노산 서열로 디코딩합니다. 훈련된 CLIP 모듈은 해당 펩타이드-단백질 임베딩을 공동으로 인코딩하고, 수천 개의 펩타이드를 스크리닝하고, 특히 다음 사항을 타겟에 대한 특정 결합 활성을 평가합니다.
* CLIP 아키텍처 및 교육
먼저, 입력 시퀀스는 동결된 ESM-2-650M 모델을 통해 내장되어 입력 임베딩을 생성합니다. 다음으로, 입력 임베딩을 시퀀스 길이에 걸쳐 평균화하여 펩타이드와 단백질에 적합한 임베딩 벡터를 얻습니다. h MLP 레이어가 적용되고, 임베딩 벡터는 ReLU(Rectified Linear Unit) 활성화 함수를 사용하여 처리되어 출력 임베딩을 얻습니다. CLIP 점수는 펩타이드와 단백질 벡터 임베딩 간의 내적을 수행하여 얻어지며, 값은 -1과 1 사이입니다. 이 모델은 펩타이드-단백질 결합 쌍이 높은 CLIP 점수를 갖도록 학습됩니다.
* 펩타이드 후보 서열 생성
후보 펩타이드는 훈련 세트의 모든 펩타이드에서 생성되며, 각 펩타이드는 PyTorch의 ESM-2-650M pLM을 사용하여 내장됩니다. 주어진 펩타이드 임베딩에 대해 임베딩의 모든 차원의 분산이 계산됩니다. 소스 펩타이드의 각 잔류물에 대해 표준 정규 분포에서 노이즈를 샘플링하고 분산을 곱하여 섭동을 생성한 다음, 이를 해당 잔류물의 임베딩에 추가합니다. 추론 시점에서 소스 펩타이드는 훈련 세트에서 무작위로 샘플링되었으며, 각 소스 펩타이드에 대해 위에 설명된 노이즈 방법을 사용하여 1,000개의 펩타이드가 생성되었습니다. 마지막으로, 이러한 펩타이드(약 10만 개)를 CLIP 모델에 입력하고 사용자가 제공한 표적 시퀀스와의 예측 결합을 기준으로 순위를 매깁니다.
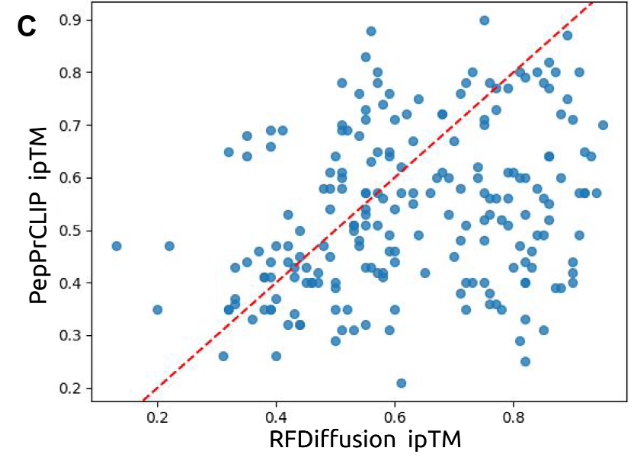
연구진은 컴퓨터 시뮬레이션에서 PepPrCLIP의 성능을 RFDiffusion의 성능과 비교했습니다. 연구진은 PepPrCLIP으로 생성된 펩타이드와 RFDiffusion으로 생성된 펩타이드의 ipTM 점수를 비교한 결과, 아래 그림에서 볼 수 있듯이 표적에 33%가 있는 펩타이드의 경우 PepPrCLIP이 RFDiffusion보다 우수한 것으로 나타났습니다. 더욱이 PepPrCLIP은 시퀀스 임베딩만을 사용하여 생성 및 우선순위 지정 속도를 크게 높여 분당 약 1,000개의 펩타이드를 생성하고 약 1분 안에 단백질 표적당 100,000개의 펩타이드를 순위를 매길 수 있습니다. 이에 비해 RFDiffusion은 단일 바인더를 설계하는 데 약 2분이 걸립니다.이러한 효율성 덕분에 PepPrCLIP은 구조 정보가 있거나 없는 대규모 펩타이드 라이브러리를 스크리닝하는 데 특히 유리합니다.
연구팀은 PepPrCLIP이 정렬된 단백질 표적과 무질서한 단백질 표적에 미치는 효과를 추가로 평가하기 위해 듀크 대학교 의과대학, 코넬 대학교 및 샌포드 번햄 프레비스 의학 발견 연구소의 연구팀과 협력하여 플랫폼을 실험적으로 테스트했습니다.
첫 번째 테스트에서 연구팀은PepPrCLIP에 의해 생성된 펩타이드는 비교적 간단하고 안정적인 효소 단백질인 UltraID에 효과적으로 결합하여 그 활동을 억제할 수 있습니다.
다음으로, 그들은 PepPrCLIP을 사용하여 여러 유형의 암에서 신호 전달에 관여하는 무질서하고 복잡한 단백질인 β-카테닌에 부착될 수 있는 펩타이드를 설계했습니다. 아래 그림에서 보듯이, 연구팀은 CLIP이 단백질에 결합할 수 있는 6개의 펩타이드를 생성했고, 그 중 4개가 표적에 효과적으로 결합하여 분해할 수 있음을 보였습니다. 단백질을 방해함으로써 암세포의 신호전달을 늦출 수 있습니다.
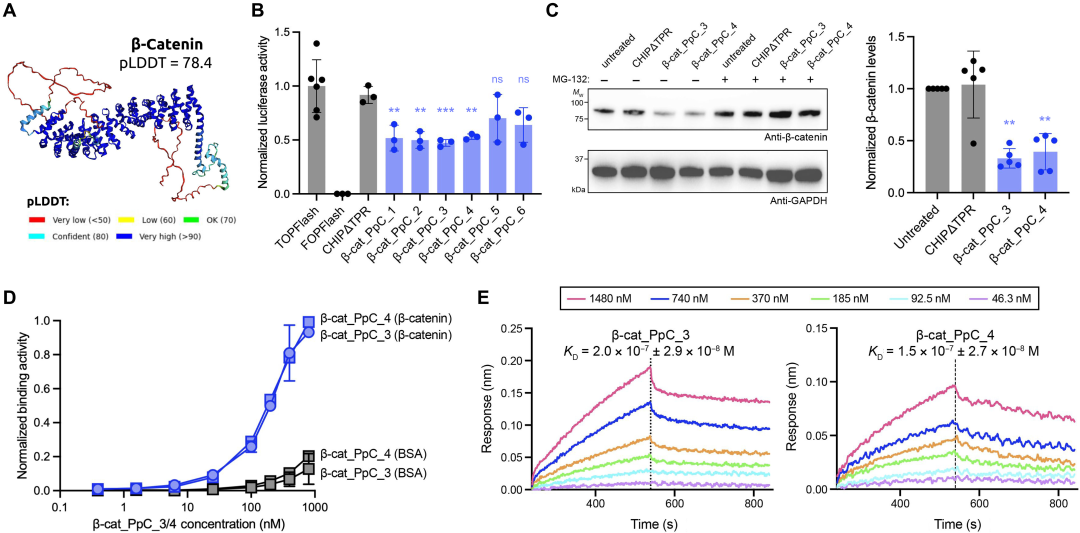
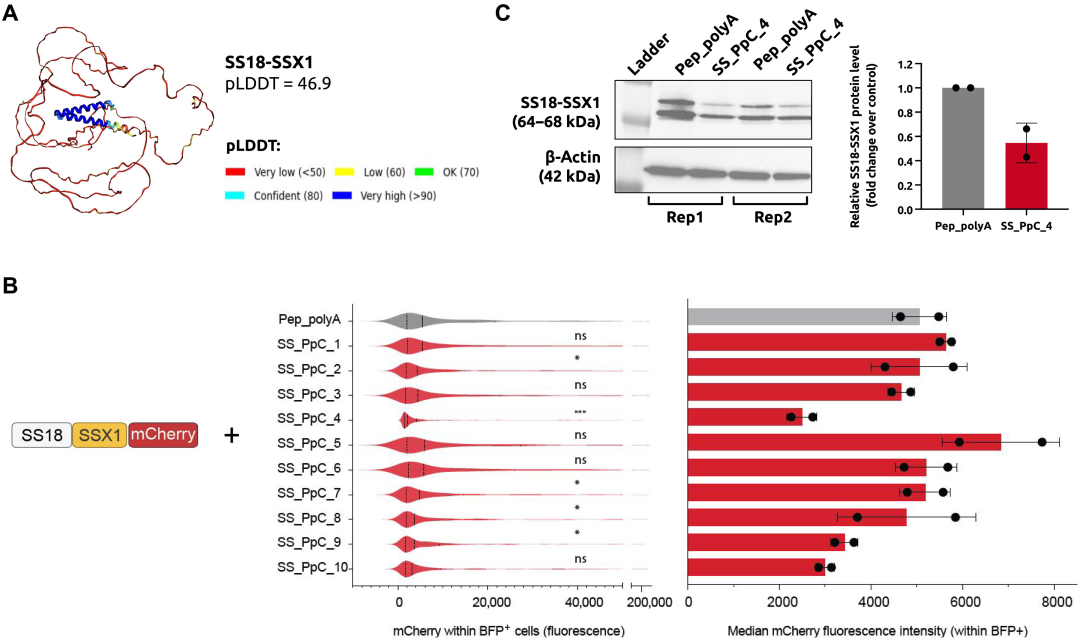
가장 복잡한 실험에서 연구팀은 활막육종과 관련된 고도로 무질서한 단백질에 결합할 수 있는 펩타이드를 설계했습니다. 활막육종은 모든 연부조직 종양 중 5%~10%를 차지하는 희귀 악성 암입니다. 활막육종은 연부조직에서 발생하며 주로 어린이와 청소년에게 영향을 미칩니다. 이 질병은 독특하고 고도로 무질서한 종양 융합 단백질인 SS18-SSX의 존재를 특징으로 합니다.
연구팀은 펩타이드를 활막육종 세포에 넣고 10가지 디자인을 테스트했습니다. 아래 결과에서 볼 수 있듯이, PepPrCLIP에 의해 SS18-SSX1에 결합할 것으로 예측된 펩타이드 중 SS_PpC_4는 SS18-SSX1-mCherry 형광을 현저히 감소시켰습니다. 다음으로 연구진은 SS_PpC_4 과잉발현이 내인성 SS18-SSX1 융합 단백질 수준에 미치는 영향을 테스트했습니다. SS_PpC_4 펩타이드의 과잉발현은 SS18-SSX1 단백질 수치를 상당히 감소시켰다는 점이 주목할 만합니다(>40%).
다시 말해서,PepPrCLIP은 단백질에 결합하고 단백질을 분해할 수 있는 펩타이드를 설계합니다.연구자들이 이 단백질을 파괴할 수 있다면, 이전에는 치료할 수 없었던 암에 대한 치료법을 개발할 수 있는 기회를 얻게 되고, 이는 많은 흥미로운 임상적 가능성을 열어줄 것입니다.
AI는 '치료 불가능한' 질병을 극복하는 새로운 도구를 제공합니다.
2023년 9월 Nature 저널에 게재된 최신 리뷰에서는 "치료 불가능한" 단백질을 표적으로 하는 약물 발견과 그 임상 적용 분야의 최신 진행 상황을 포괄적으로 소개합니다.유사한 약물화 불가능한 특성을 가진 다양한 분자는 다음 범주로 나뉘었습니다.
① 소형 GTPase: KRAS, HRAS 및 NRAS를 포함한 RAS 계열 단백질과 같이 표면에 표적화 가능한 포켓이 없기 때문에 약물로 치료할 수 없는 것으로 간주됨
② 포스파타아제: 각 포스파타아제는 구조상 많은 유사성을 가지고 있어 선택성이 낮고 불가피한 부작용이 있어 약물 발견의 진행에 큰 방해가 됩니다.
③ 전사 인자(TF): 다양한 인간 질병은 많은 생물학적 과정에 관여하는 전사 인자의 조절 장애와 관련이 있으며, 이 중 대부분은 구조적 이질성과 가공 가능한 결합 부위의 부족으로 인해 기존의 소분자로는 표적이 될 수 없습니다.
④ 후성유전학적 표적: 후성유전학적 표적은 유전자 발현 패턴을 조절하는 데 중요한 역할을 하며 다양한 생물학적 과정과 질병에 영향을 미칩니다.
⑤ 기타 단백질: 단백질-단백질 상호작용(PPI)과 그 네트워크는 생물학적 과정과 세포주기 조절에 매우 중요합니다. 평평한 상호작용 표면을 가진 일부 PPI는 다른 PPI보다 타겟팅하기가 더 어려워 어느 정도까지는 "치료가 불가능합니다".
오늘날 학계에서는 소위 '치료 불가능한' 표적에 직면하여 수십 가지의 혁신적인 방법을 개발했습니다. 그들은 약물화 불가능한 단백질의 메커니즘에 따라 단편 기반 약물 발견(FBDD), 컴퓨터 지원 약물 설계(CADD), 가상 스크리닝(VS), DNA 인코딩 라이브러리(DEL) 등의 최첨단 기술을 도입하여 체계적인 약물 설계 전략을 수립했습니다. 오늘날 인공지능 기술의 발전과 단백질 대규모 언어 모델의 등장으로 이 문제를 극복할 수 있는 새로운 도구가 제공되었습니다.최근 몇 년 동안 업계와 학계 모두에서 중요한 혁신이 이루어졌습니다.
산업,2023년 12월, 생성적 AI 항체 발견 분야의 선두주자인 Absci Corporation은 종양 표적에 대한 AI 설계 항체를 개발하기 위해 AstraZeneca와 협력한다고 발표했습니다. 이 협력은 Absci의 통합 약물 창조 플랫폼과 AstraZeneca의 종양학 전문 지식을 결합하여 잠재적인 새로운 암 치료 후보 물질의 발견을 가속화합니다. Absci의 통합 약물 생성 플랫폼은 수백만 개의 단백질-단백질 상호작용을 측정하여 독점 데이터를 생성합니다. 이 데이터는 Absci의 독점 AI 모델을 훈련하는 데 사용되며, 이후 반복 작업에서는 새로운 AI 모델을 사용하여 설계된 항체를 검증합니다. 이 플랫폼은 데이터 수집, AI 기반 설계, 실험실 검증을 약 6주 만에 완료하여 약물 개발을 가속화하고, 이전에는 약물 개발이 불가능하다고 여겨졌던 표적에 대한 약물 개발을 포함하여 약물 표적 범위를 확대할 수 있는 잠재력을 가지고 있습니다.
학계,2025년 1월, AI 제약 분야의 선두주자인 Insilico Medicine과 캐나다 토론토 대학이 공동으로 주도한 연구에서는 양자 컴퓨팅 모델과 기존 컴퓨팅 모델, 생성적 인공 지능을 결합하여 방대한 데이터 세트의 훈련, 생성 및 검토를 통해 더 광범위한 화학적 가능성을 탐색했으며, "치료 불가능한" 암 유발 단백질 KRAS를 표적으로 하는 새로운 분자를 발견했습니다.
KRAS 돌연변이는 암에서 가장 흔한 돌연변이 중 하나로, 인간 종양의 약 4분의 1에서 발생합니다. KRAS 돌연변이는 통제되지 않는 세포 증식을 초래하고, 결국 암으로 이어질 수 있습니다. 이 연구에서 연구진은 잠재적인 새로운 KRAS 억제제를 생성하기 위해 양자 변이 생성 모델(QCBM)과 장단기 기억 네트워크(LSTM)를 결합한 양자-고전적 하이브리드 프레임워크 모델을 제안했으며, 양자 컴퓨팅과 고전적 컴퓨팅 방법을 결합하여 새로운 분자를 설계했습니다. 이 연구는 또한 세인트 주드 어린이 연구 병원을 포함한 여러 연구 기관의 지원을 받았습니다. 관련 연구 결과는 "양자 컴퓨팅 강화 알고리즘이 잠재적인 KRAS 억제제를 밝혀냈다"라는 제목으로 Nature Biotechnology에 게재되었습니다.

관련 기술의 획기적인 발전으로 인류는 질병을 물리치는 데 있어 새로운 상상력의 공간과 무한한 가능성을 열었습니다.