데모를 가장 먼저 경험해 보세요! 게놈 기반 모델 Evo가 Science 표지에 실려 분자 수준에서 게놈 수준까지 예측 및 생성이 가능해졌습니다.

최근 AI는 생물학 분야에서 큰 진전을 이루었습니다.미국 스탠포드 대학과 아크 연구소의 연구팀은 DNA, RNA, 단백질의 다중 모드 작업에서 제로 샘플 예측과 고정밀 생성을 달성할 수 있는 게놈 기반 모델 Evo를 제안했습니다.
관련 연구는 "Evo를 활용한 분자 규모에서 유전체 규모까지의 시퀀스 모델링 및 설계"라는 제목으로 Science에 표지 논문으로 게재되었습니다.
서류 주소:
https://www.science.org/doi/10.1126/science.ado9336
공식 계정을 팔로우하고 "Evo"라고 답글을 달면 전체 PDF를 받을 수 있습니다.
논문의 첫 번째 저자인 에릭 응우옌은 Evo의 성과를 소개하는 여러 업데이트를 게시하고, 팀원들에게 여러 번 감사를 표했으며, 솔직하게 "이렇게 훌륭한 팀과 함께 일하게 되어 영광입니다!"라고 말했습니다.

논문에 따르면, Evo는 StripedHyena 아키텍처를 사용하며 80,000개 이상의 박테리아 및 고균 게놈을 포함하는 대규모 게놈 데이터 세트를 통해 학습되었습니다.그리고 3,000억 개의 뉴클레오티드 토큰을 포함하는 수백만 개의 예측된 파지 및 플라스미드 시퀀스를 통해 1메가베이스 이상의 길이를 갖는 합리적인 게놈 아키텍처를 갖춘 DNA 시퀀스를 생성할 수 있습니다.
또한,70억의 매개변수 규모와 131,072개의 토큰의 최대 컨텍스트 길이를 갖춘 Evo는 코딩 및 비코딩 시퀀스 간의 복잡한 공진화를 밝혀내고 복잡한 생물학적 시스템을 설계할 수 있습니다.예를 들어 CRISPR-Cas 복합체와 IS200 및 IS605 트랜스포존 등이 있습니다.
요약하자면, Evo가 전체 게놈 시퀀스를 예측, 생성, 설계하는 능력은 생명 과학에 새로운 이론적 뒷받침을 제공할 뿐만 아니라, 유전자 편집, 약물 발견, 질병 진단, 농업 및 기타 분야에 적용되어 여러 분야에서 획기적인 결과를 도출하는 데 도움이 될 것으로 기대됩니다.
많은 네티즌들은 Evo의 출시에 충격을 받았고, 이 모델이 구체적으로 어떤 용도로 사용될지 기대했습니다.
모든 분들이 Evo 모델의 강력한 기능을 처음으로 경험하실 수 있도록,HyperAI의 HyperNeural 튜토리얼 섹션 "Evo: 분자 규모에서 유전체 규모까지의 예측 및 생성"이 이제 온라인으로 제공됩니다.어떤 명령도 입력할 필요 없이, 한 번의 클릭으로 복제하여 빠르게 경험해보세요!
튜토리얼 링크:
데모 실행
1. hyper.ai에 로그인하고 튜토리얼 페이지에서 Evo: 분자에서 게놈 규모까지 예측 및 생성을 선택한 후 이 튜토리얼을 온라인으로 실행을 클릭합니다.
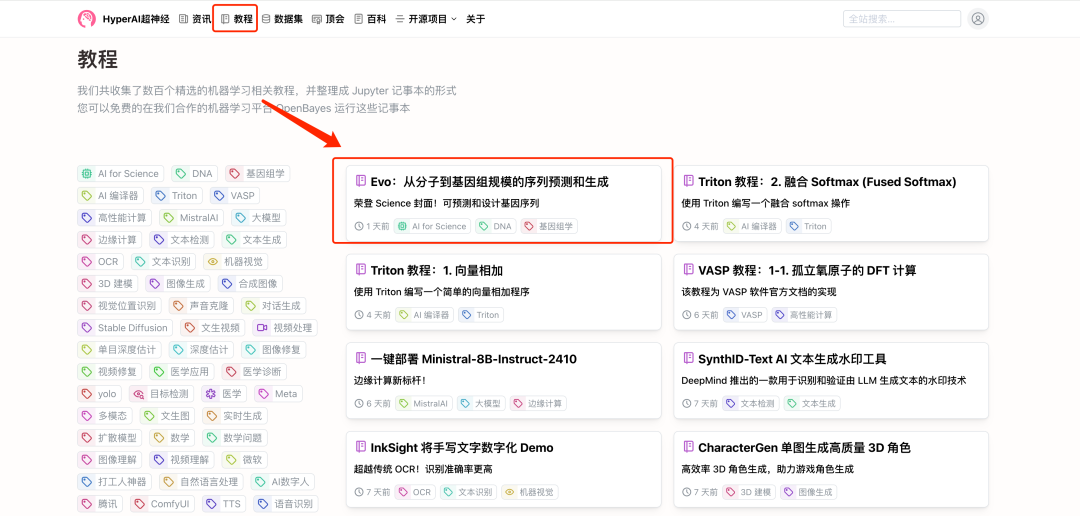
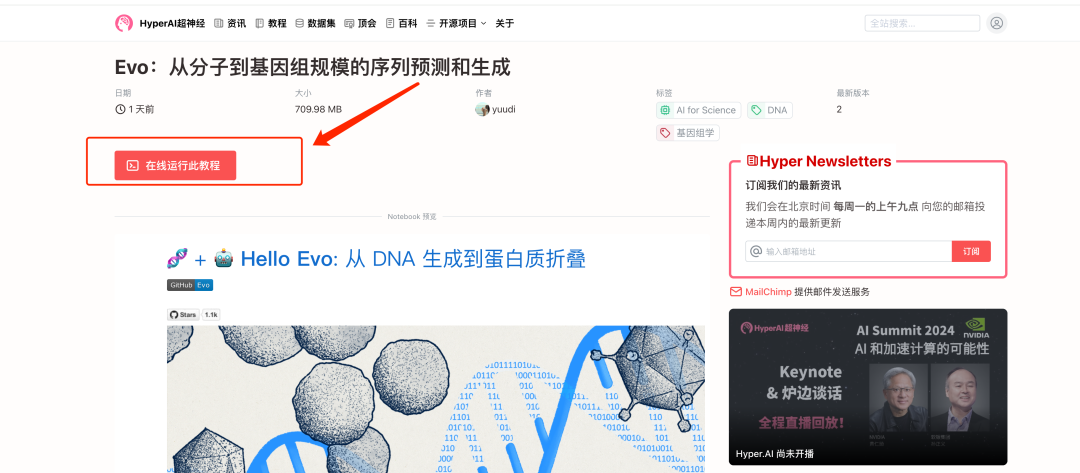
2. 페이지가 이동한 후 오른쪽 상단의 "복제"를 클릭하여 튜토리얼을 자신의 컨테이너로 복제합니다.
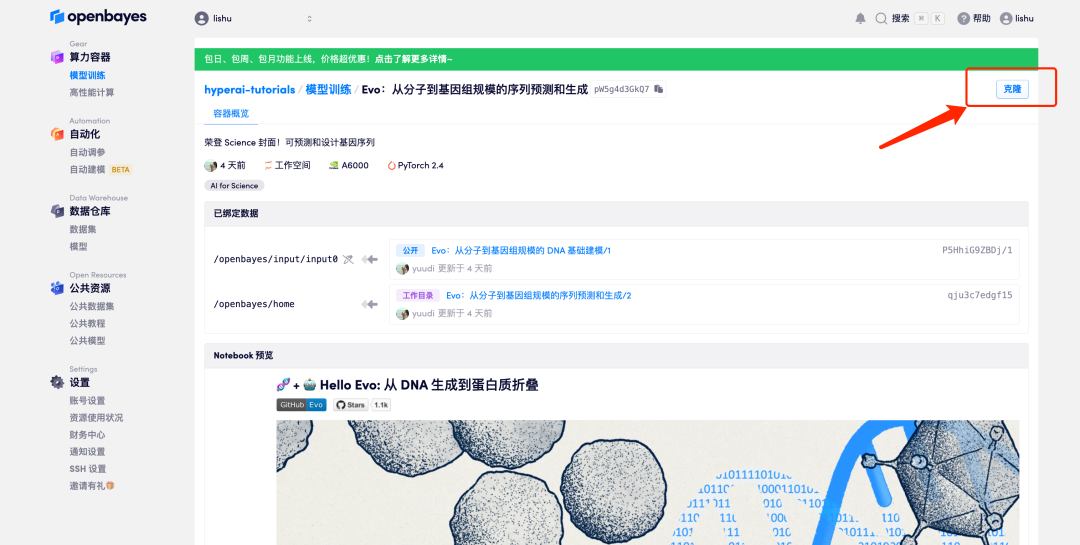
3. 오른쪽 하단에 있는 "다음: 해시레이트 선택"을 클릭합니다.
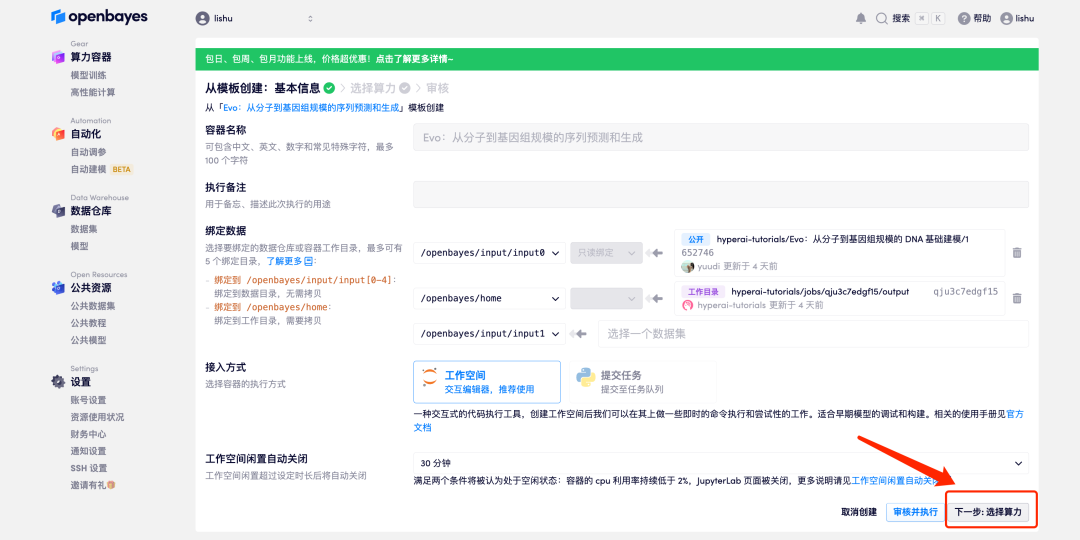
4. 페이지가 이동한 후 "NVIDIA RTX A6000"을 선택하고 필요에 따라 "Pay as you go" 또는 "Daily/Weekly/Weekly Package"를 선택하세요. "PyTorch" 이미지를 선택한 후 "다음: 검토"를 클릭합니다. 신규 사용자는 아래 초대 링크를 사용하여 등록하고 RTX 4090 4시간 + CPU 자유 시간 5시간을 받으세요!
HyperAI 독점 초대 링크(복사하여 브라우저에서 열기):
https://openbayes.com/console/signup?r=Ada0322_QZy7
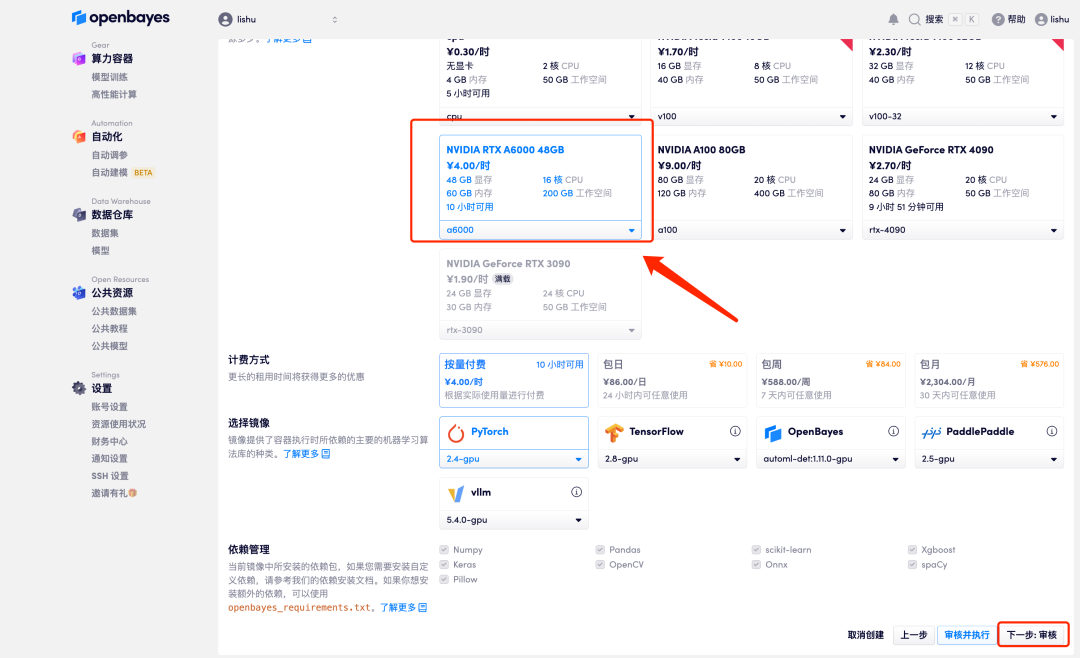
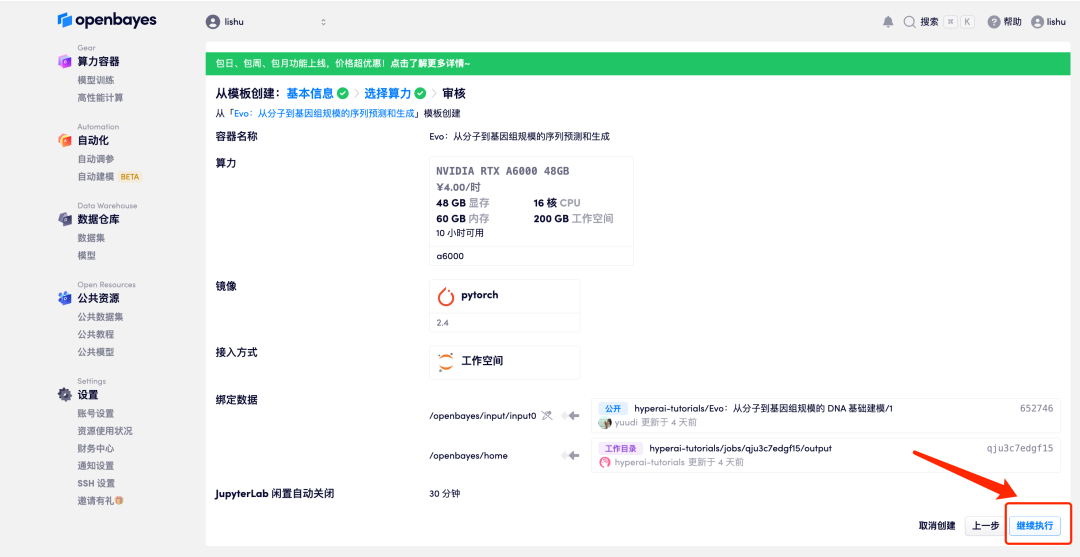
5. 확인 후 "계속"을 클릭하고 리소스가 할당될 때까지 기다리세요. 첫 번째 복제 과정은 약 2분 정도 걸립니다.
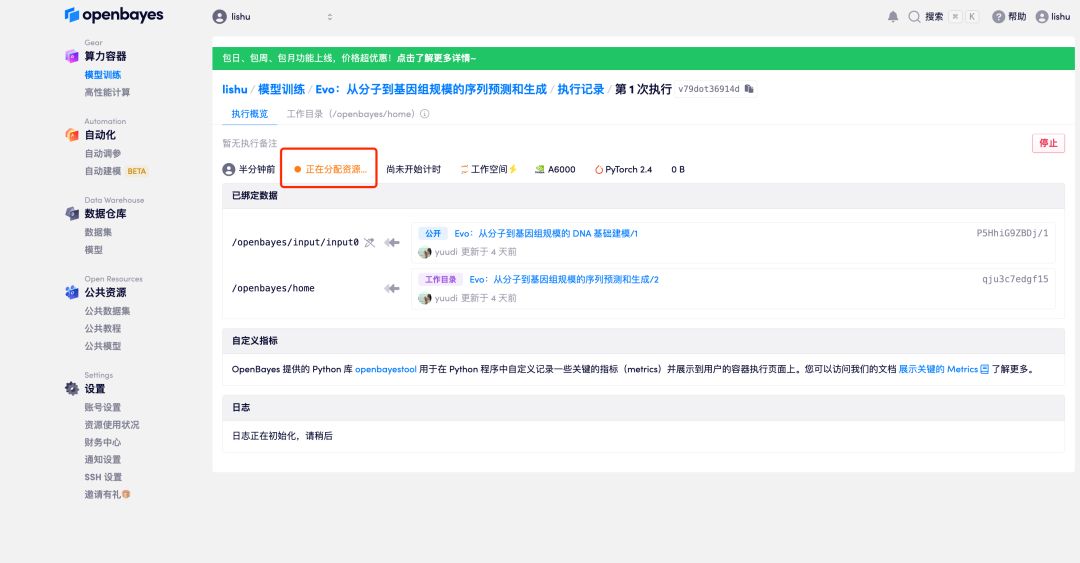
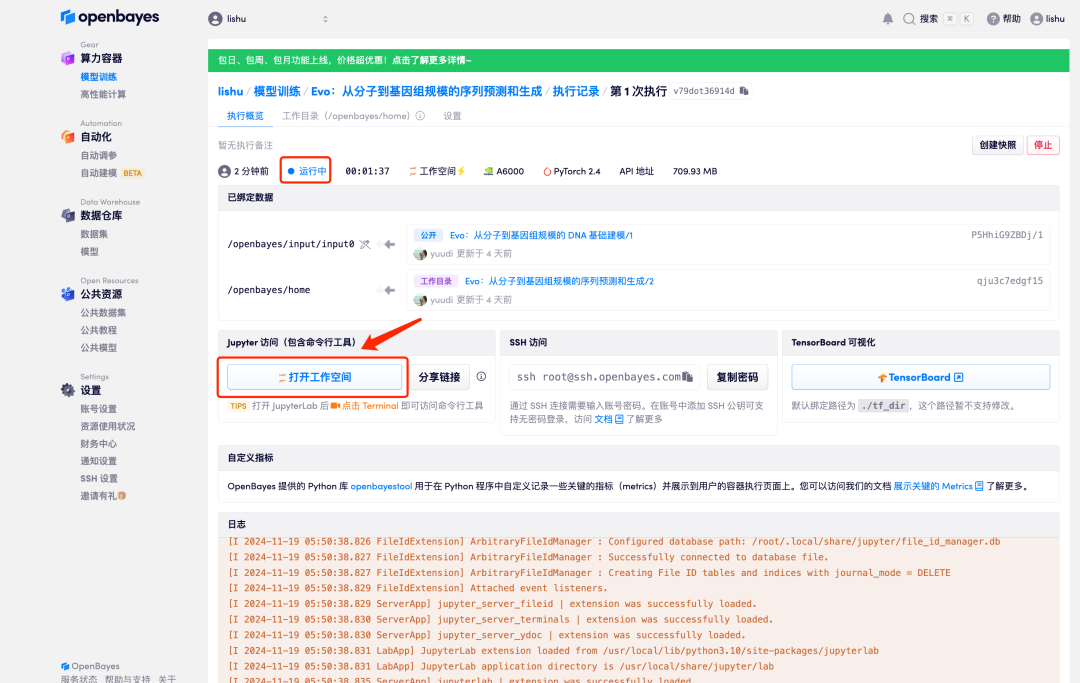
6. 상태가 "실행 중"으로 변경되면 "작업 공간 열기" 옵션을 클릭합니다.
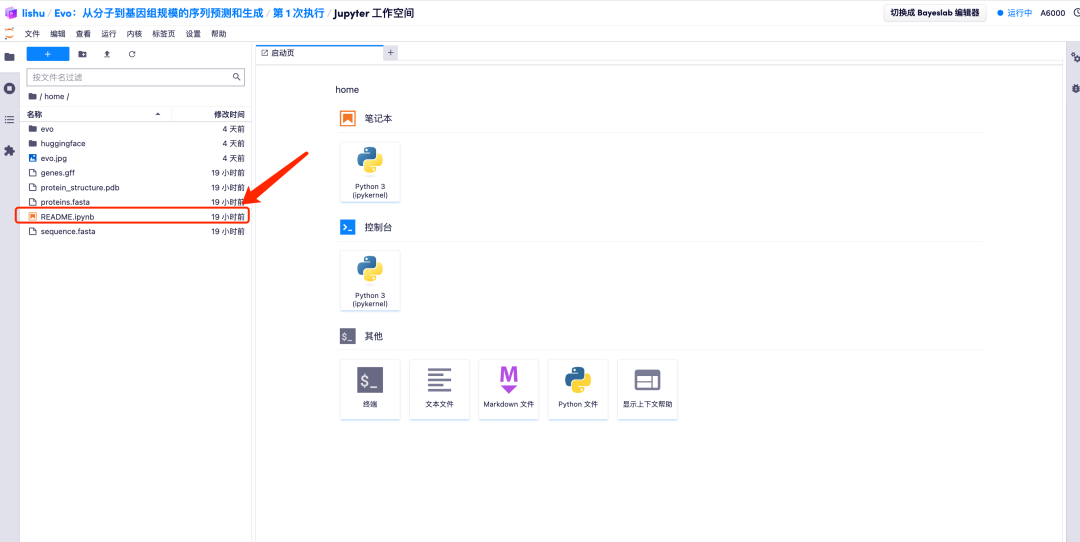
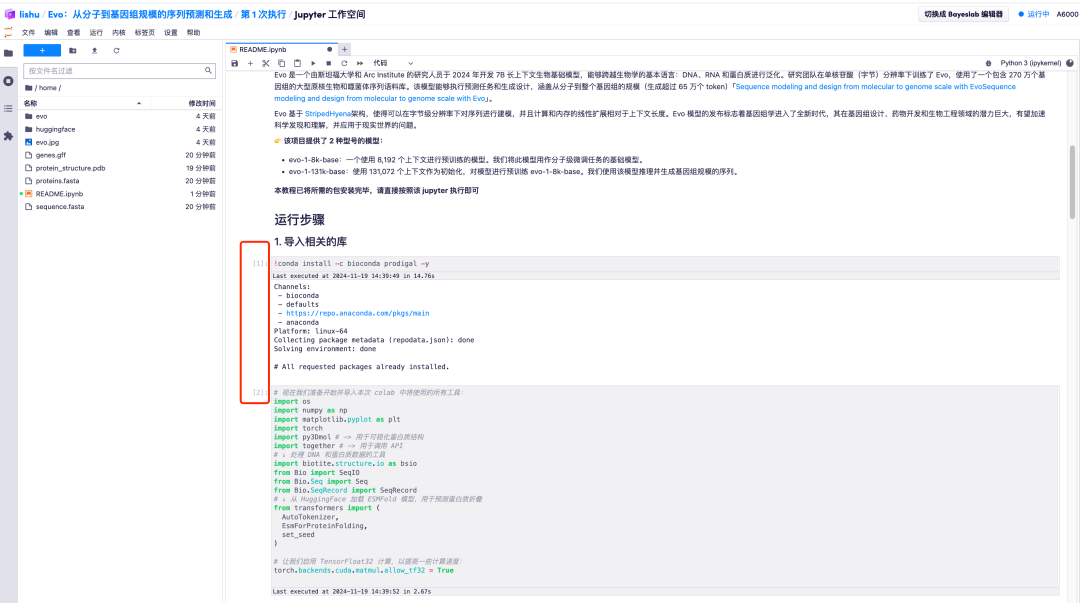
7. Jupyter 작업 공간에 들어간 후, "README" 파일을 두 번 클릭하여 공식적으로 Evo 모델 실행 페이지로 들어갑니다.
효과 시연
1. Evo 모델 실행 페이지에 들어가면 모든 매개변수가 기본 모드에 있습니다. "2. 모델 시작 및 관련 매개변수 입력"까지 아래로 스크롤하여 필요에 따라 프롬프트 매개변수 값을 조정하세요.prompt의 기본값은 "ACGT"이며, 이는 DNA 염기쌍(A, C, G, T)으로 구성된 초기 시퀀스를 나타냅니다.필요에 따라 이 값을 수정하여 다른 DNA 시퀀스를 생성할 수 있습니다.
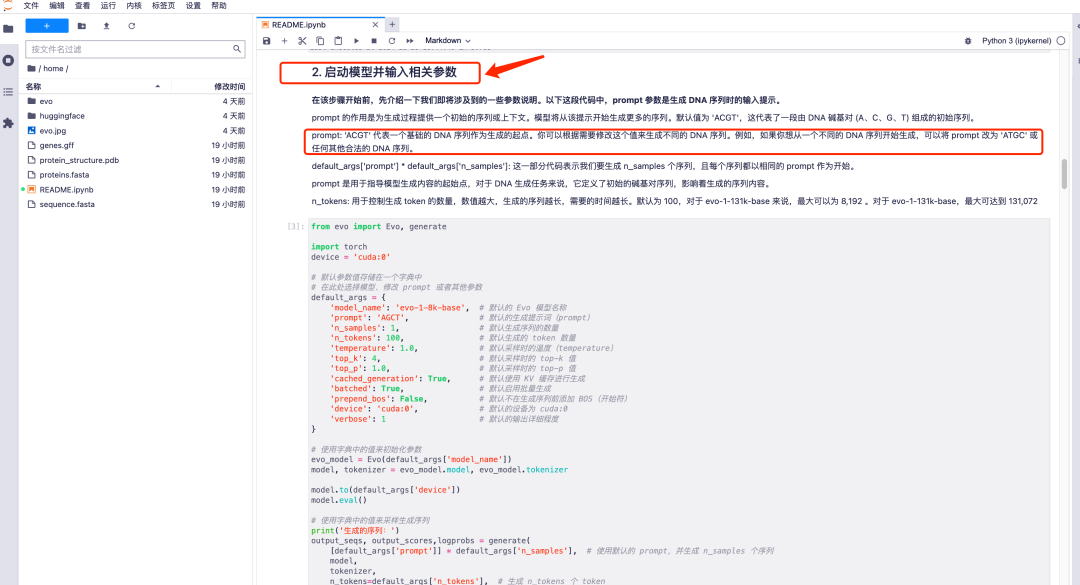
2. 예를 들어 prompt의 기본값을 AGCT로 변경합니다. 기본 매개변수 값을 조정한 후 "모든 셀을 다시 시작하고 실행" 옵션을 클릭하고 "다시 시작"을 선택하여 실행합니다.
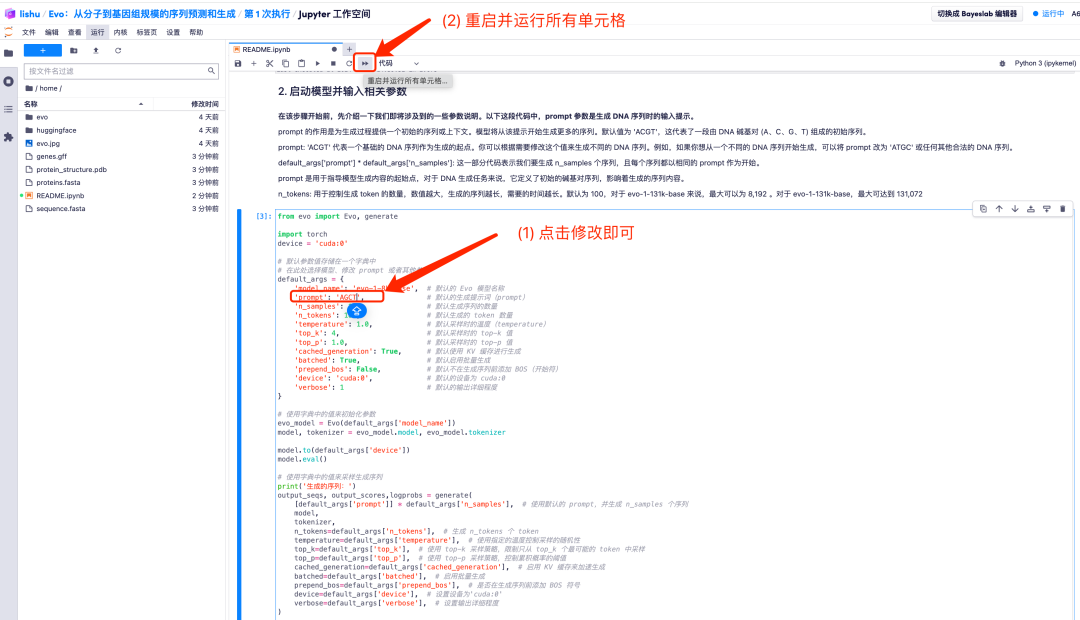
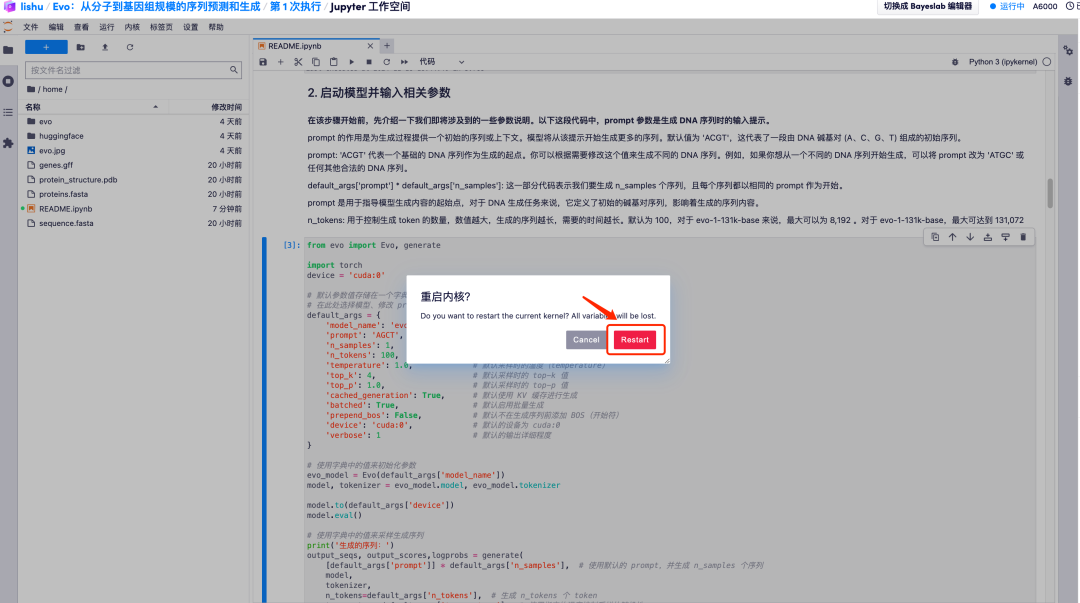
3. 잠시 기다리세요. [*] 기호가 숫자로 바뀌면 작업이 완료됩니다. "2. 모델 시작 및 관련 매개변수 입력" 하단에서 생성된 시퀀스를 볼 수 있습니다.
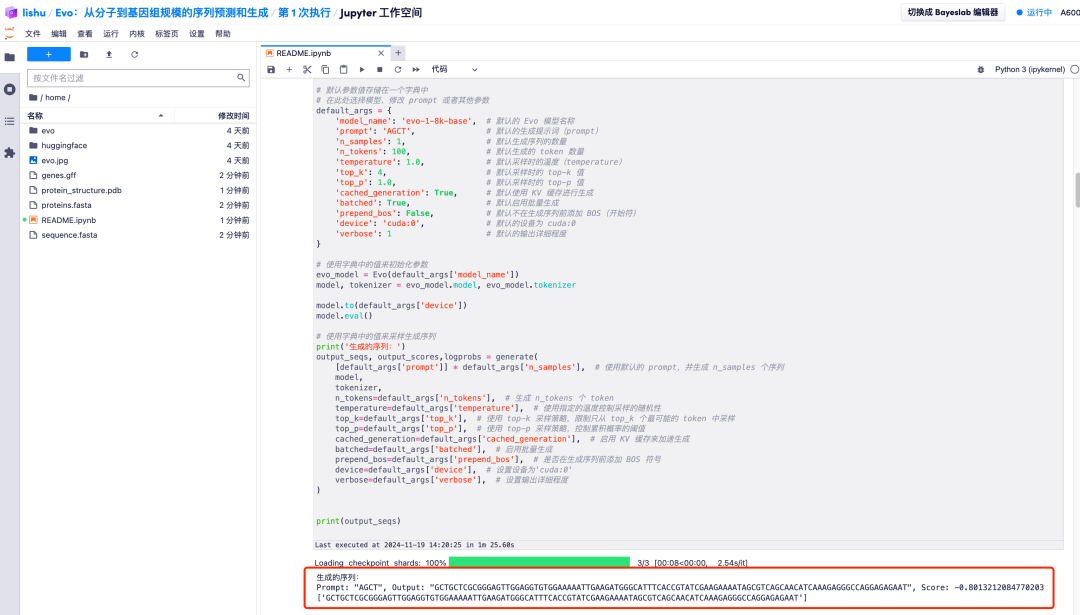
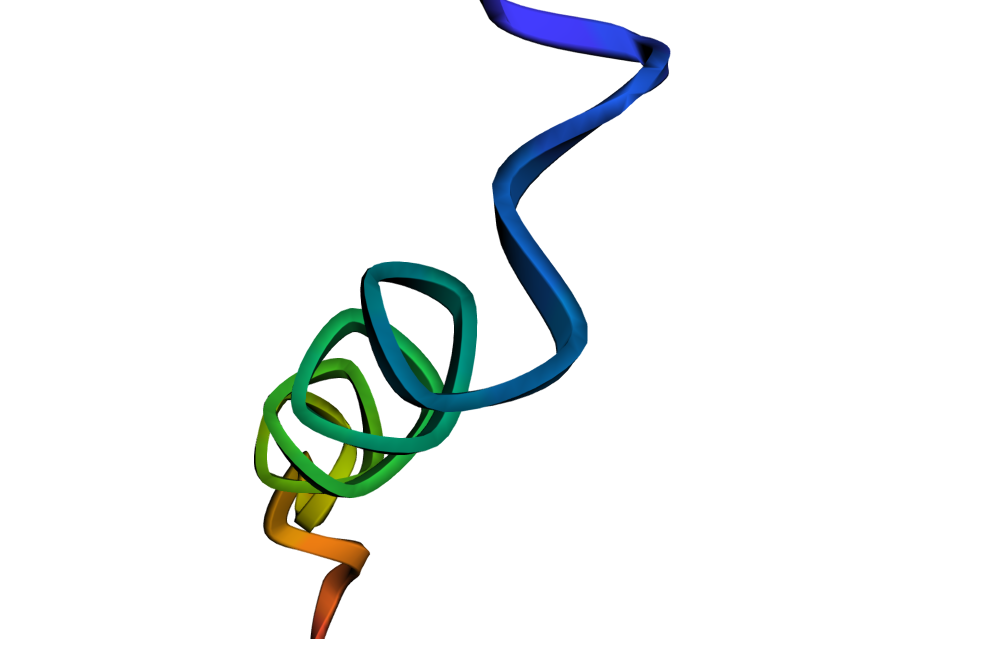
4. 또한, Evo 모델은 생성된 DNA 서열을 분석하고 코딩 서열과 비코딩 서열 간의 공진화 관계를 학습할 수도 있습니다. 또한 DNA 서열에서 단백질 코딩 유전자를 예측하고, RNA 시스템을 인코딩하고 설계하여 결과적으로 생성되는 단백질 접힘 구조를 예측할 수 있으며, 이는 궁극적으로 이미지 형태로 표현될 수 있습니다.
우리는 "안정 확산 튜토리얼 교환 그룹"을 만들었습니다. 다양한 기술적인 문제를 논의하고 신청 결과를 공유하기 위해 그룹에 가입해 주세요~
아래의 QR 코드를 스캔하여 WeChat에 HyperaiXingXing을 추가하세요(WeChat ID: Hyperai01). 그리고 "SD 튜토리얼 교환 그룹"을 메모하여 그룹 채팅에 참여하세요.
