Command Palette
Search for a command to run...
56%로 민감도 향상, CUHK/Fudan/Yale 등 공동으로 새로운 단백질 동족체 검출 방법 제안

단백질은 생명의 물질적 기초이며 생명 활동의 주요 운반체입니다. 포스트 게놈 시대에는 단백질 결정 기술이 발달하면서 단백질 서열 데이터베이스의 크기가 폭발적으로 증가했습니다. 단백질의 다양성과 기능을 더 깊이 이해하기 위해서는 생물학에서 단백질 식별이 특히 중요합니다.
단백질 인식 과정에서 단백질 서열 상동성 확인은 가장 중요한 과제 중 하나입니다.이는 과학자들이 단백질의 진화적 관계, 구조적 특성, 기능을 이해하는 데 도움이 될 수 있습니다. 기존의 단백질 서열 정렬 방법은 많은 경우 좋은 성과를 보이지만, 멀리 떨어진 동족체에 대처할 수는 없습니다. 이러한 먼 상동체는 서열 유사성이 낮기 때문에 일상적인 정렬에서 간과되는 경우가 많아 연구자들이 단백질 다양성과 복잡성을 종합적으로 이해하는 데 제한이 됩니다.
홍콩 중국 대학의 리위(Li Yu)는 푸단 대학의 지능 복잡 시스템 연구실과 상하이 인공지능 연구소의 젊은 연구원인 쑨스치(Sun Siqi), 예일 대학의 마크 거스타인(Mark Gerstein)과 함께 단백질 언어 모델과 고밀도 검색 기술을 기반으로 먼 단백질 상동성 연구의 문제점을 해결하기 위해 초고속 고감도 상동성 감지 프레임워크인 고밀도 상동성 검색기(DHR)를 제안했습니다.
DHR은 듀얼 인코더 구조와 단백질 언어 모델의 강력한 기능을 통해 기존의 서열 정렬에 의존하지 않고도 서열 깊숙이 숨겨진 먼 동족체를 식별할 수 있으며, 동족체 식별에 전례 없는 속도와 민감성을 제공합니다. 해당 연구는 "딥밀도 검색을 이용한 단백질 동족체의 빠르고 민감한 검출"이라는 제목으로 국제적으로 유명한 저널인 Nature Biotechnology에 게재되었습니다.
연구 하이라이트:
* 기존 방식과 비교했을 때 DHR은 민감도를 10% 이상 향상시키고 정렬 기반 방식으로 식별하기 어려운 샘플의 경우 슈퍼패밀리 수준에서 민감도를 56% 이상 향상시킵니다.
* DHR 코드는 PSI-BLAST 및 DIAMOND와 같은 기존 방식보다 22배 빠르고 HMMER보다 28,700배 더 빠른 시퀀스 및 데이터베이스 쿼리를 수행합니다.

서류 주소:
https://doi.org/10.1038/s41587-024-02353-6
오픈소스 프로젝트인 "awesome-ai4s"는 100개가 넘는 AI4S 논문 해석을 모아 방대한 데이터 세트와 도구를 제공합니다.
https://github.com/hyperai/awesome-ai4s
더 넓은 범위의 단백질 서열을 탐색하기 위해 다차원 데이터 세트 구축
본 연구에서 구축한 훈련 세트에는 UR90에서 신중하게 선택한 200만 개의 쿼리 시퀀스가 포함되어 있습니다.이 연구에서는 JackHMMER 알고리즘을 사용하여 Uni-Clust30에서 후보 시퀀스를 반복적으로 검색하고 후보 시퀀스를 다중 시퀀스 정렬(MSA)로 정렬했습니다. 각 MSA에는 1,000개의 동족체가 포함되어 있어 가장 관련성이 높은 시퀀스만 유지되었습니다. 엄격한 검토를 거친 후, JackHMMER는 획득한 다양한 시퀀스를 처리하기 위해 재배치되었으며, 공정한 비교를 용이하게 하기 위해 AF2(AlphaFold 2)와 동일한 하이퍼파라미터 설정을 사용했습니다.
대규모 데이터 세트를 연구하기 위해 본 연구에서는 BFD/MGnify 데이터 세트를 선택했습니다.이는 약 3억 개의 단백질을 보유한 거대한 데이터베이스로, 이를 통해 더 광범위한 단백질 서열을 탐색할 수 있습니다.
DHR 방법: 초고속 및 고감도 단백질 동족성 검색 파이프라인
DHR 방법의 핵심 아이디어는 단백질 서열을 고밀도 임베딩 벡터로 인코딩하여 서열 간의 유사성을 효과적으로 계산하는 것입니다.구체적으로, 이 연구는 ESM을 초기화하고 대조 학습 기술을 통합하여 시퀀스 인코더를 효과적으로 훈련시켰고, 이를 통해 단백질 언어 모델을 구축할 수 있는 조건을 만들고 DHR을 사용하여 동족체를 더 효과적으로 검색할 수 있게 했습니다.
아래 그림 a에서 볼 수 있듯이, 듀얼 인코더 학습 단계가 완료되면 이 연구에서는 고품질의 오프라인 단백질 시퀀스 임베딩을 생성할 수 있습니다. 그런 다음 연구에서는 이러한 임베딩과 유사성 검색 알고리즘을 사용하여 각 쿼리 단백질에 대한 동족체를 검색했습니다. 유사성을 검색 지표로 지정하면 기존 방법보다 유사한 단백질을 더 정확하게 찾을 수 있으며, 두 단백질 간의 유사성을 추가 분석에 사용할 수 있습니다. 마지막으로, JackHMMER는 검색된 동족체의 MSA를 구축했고, 이 연구에서는 동족체를 빠르고 효과적으로 발견할 수 있는 DHR 기술을 얻었습니다.
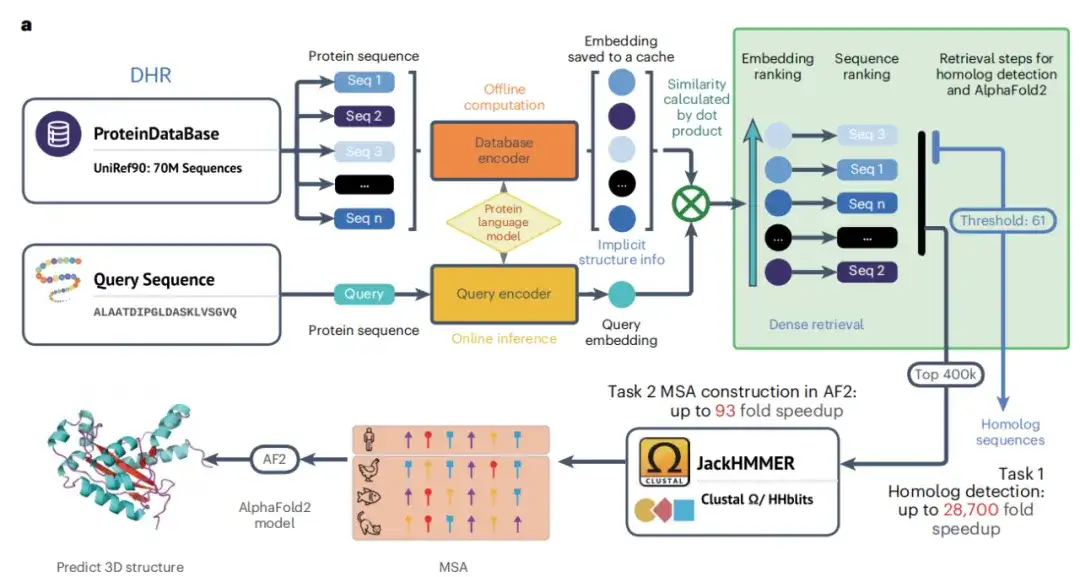
그뿐만 아니라, 이 연구에서는 DHR과 AF2 기본값을 결합하여 CASP13DM(도메인 시퀀스) 및 CASP14DM 타겟에 대한 개별 파이프라인보다 우수한 성능을 보이는 하이브리드 모델 DHR-meta도 개발했습니다.
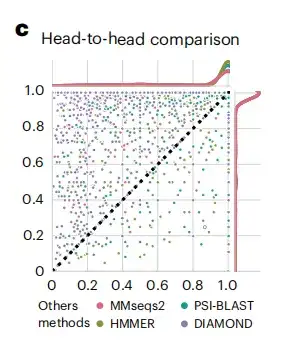
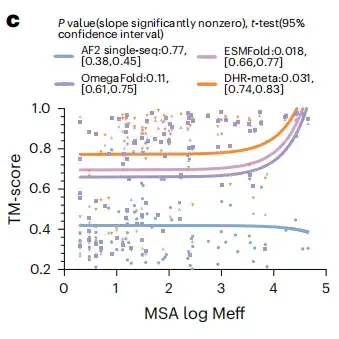
생성된 단백질 임베딩을 얻은 후, 본 연구에서는 표준 SCOPe(단백질 구조 분류) 데이터 세트의 방법과 비교하여 DHR의 성능을 평가했습니다.아래 그림 c에서 볼 수 있듯이 DHR 데이터의 민감도는 다른 방법보다 더 좋습니다.
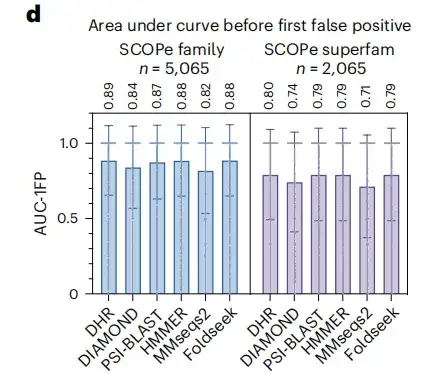
또한, 아래 그림 h에서 보듯이, d1w0ha 쿼리의 구체적인 예에서 PSI-BLAST나 MMseqs2는 어떤 결과와도 일치하지 않았지만, DHR은 5개의 동족체를 검색했고, 이들 역시 SCOPe에서 d1w0ha와 동일한 패밀리로 분류되었습니다. 즉, DHR은 더 많은 구조적 정보를 포착할 수 있습니다. PSI-BLAST, MMseqs2, DIAMOND, HMMER 등의 기존 방법과 비교했을 때, DHR은 가장 많은 동족체를 감지했습니다(감도 93%).이는 DHR이 풍부한 구조적 정보를 통합하고 많은 경우 100% 감도를 달성할 수 있음을 보여줍니다.
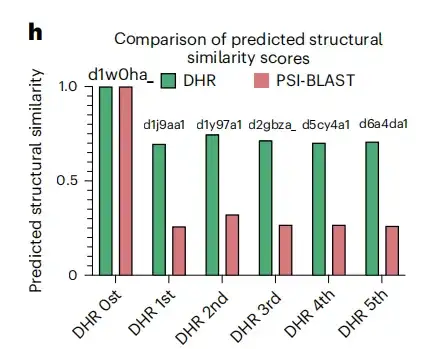
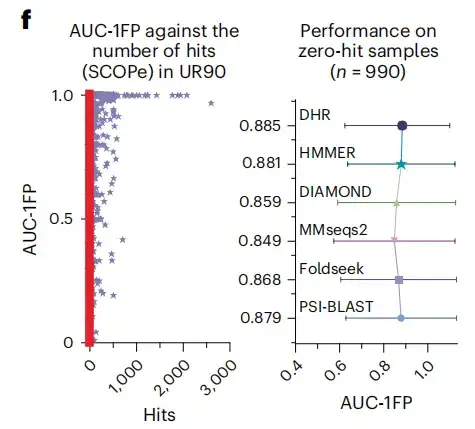
연구 결과의 신뢰성을 강화하기 위해 이 연구에서는 첫 번째 FP 이전 곡선 아래의 면적이라는 또 다른 표준 지표를 포함했습니다. 결과는 아래 그림 d에서 볼 수 있듯이 DHR이 89%의 점수를 달성한다는 것을 보여줍니다.한편, 다른 방법들도 DHR과 비슷한 성능을 보였지만, 실행 시간이 훨씬 더 길었습니다.더욱 어려운 먼 동족체를 분석하기 위해 슈퍼패밀리 수준으로 이동했을 때, 모든 방법에서 성능이 크게 떨어졌으며, 전체적으로 약 10%가 감소했습니다. 이러한 사실에도 불구하고 DHR은 여전히 80%라는 높은 AUC-1FP 점수를 기록하며 선두적인 성과를 유지하고 있습니다.
이 연구에서는 BLAST를 사용하여 SCOPe 데이터베이스와 UniRef90을 비교했을 때 대부분 샘플에서 100개 미만의 일치 항목이 생성되었고, 심지어 약 500개 샘플에서는 일치하는 항목이 전혀 발견되지 않았음을 발견했습니다. 이는 이러한 샘플이 훈련 데이터 세트에서 "보이지 않는" 구조였음을 나타냅니다. 이와 대조적으로 DHR은 여전히 이러한 구조에 대해 고품질 예측을 달성하여 AUC-1FP 점수 89%에 도달했습니다.이는 DHR이 완전히 새로운 데이터를 처리할 수 있는 능력을 보여줍니다.
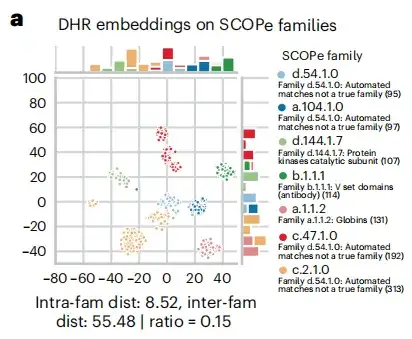
아래 그림 a에 표시된 것처럼 상동성 검색 과정에서 DHR 시퀀스 임베딩에는 많은 양의 구조적 정보가 포함되어 있으며, 상동 물질을 검색하는 데 있어 DHR의 정확도는 구조 기반 정렬 방법보다 더 높다는 것을 연구에서 발견했습니다. 이 결과를 바탕으로,이 연구는 DHR의 서열 유사성 순위와 구조적 유사성 사이의 상관관계를 더욱 명확히 밝혔습니다.
연구 결과: DHR은 정확도와 효율성이 더 뛰어나며 대규모 데이터 세트에서 고품질 MSA를 구축할 수 있습니다.
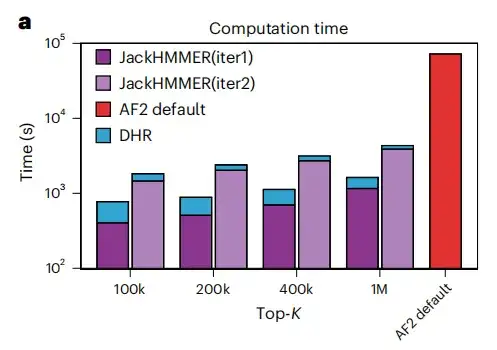
우리는 DHR에서 제공한 동족체를 사용하여 JackHMMER의 MSA를 만들고 이를 AF2 기본 파이프라인과 비교했습니다. 아래 그림 a에서 볼 수 있듯이, 모든 구성의 DHR + JackHMMER의 평균 주행 속도는 AF2의 일반 JackHMMER보다 빠릅니다. 게다가, UniRef90에서 MSA를 구성할 때 DHR은 JackHMMER와 약 80%만큼 겹칩니다.이는 MSA와 관련된 많은 하위 작업을 DHR을 사용하여 수행할 수 있으며, 비슷한 결과를 더 빠르게 얻을 수 있음을 시사합니다.
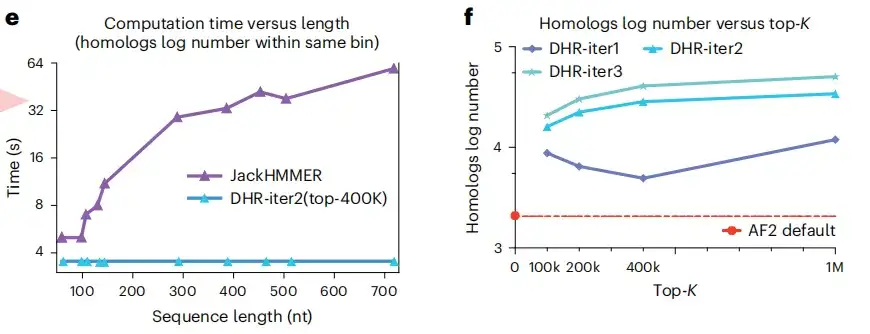
아래 그림 e와 f에서 볼 수 있듯이 DHR의 또 다른 장점은 JackHMMER가 선형적으로 확장되는 반면, DHR은 일정한 시간 내에 서로 다른 길이의 동족체를 같은 수로 구성할 수 있다는 점입니다. 게다가 DHR은 AF2에 비해 쿼리 임베딩을 위한 더 많은 동족체와 MSA를 제공할 수 있습니다. 이러한 결과는 다음을 나타냅니다.DHR은 모든 종류의 MSA 건설에 유망한 접근 방식입니다.
DHR은 다양한 MSA를 생성할 수 있지만, 이 연구에서는 이것이 AF2 기준 MSA를 보완하는 역할을 할 수 있는지 여부를 추가로 분석했습니다. 연구 결과에 따르면, 아래 그림 a와 b에서 볼 수 있듯이, 다양한 DHR 설정에서 모든 MSA와 AF2를 결합하는 것이 가장 좋은 성능을 보입니다.즉, DHR은 AF2의 MSA 파이프라인을 빠르고 정확하게 보충할 수 있습니다.
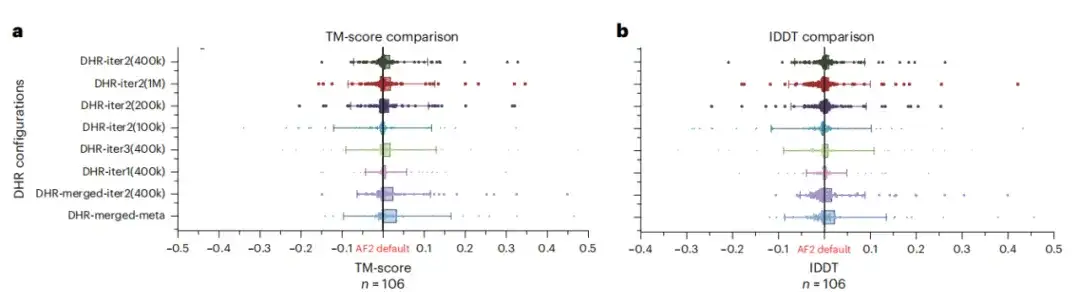
단백질 구조 예측을 위한 대규모 언어 모델의 잠재적 이점을 조사하기 위해 이 연구에서는 모든 CASP14DM 대상에서 MSA를 대규모 언어 모델로 대체하면 더 나은 결과가 나오는지 평가했습니다. 아래 그림 c에서 보듯이, 사용 가능한 MSA의 수가 많은 간단한 사례에서 언어 모델은 MSA만큼 많은 정보를 전달할 수 있습니다. 그러나 시퀀스 길이가 늘어날수록 DHR-meta의 성능은 점점 더 좋아져 거의 모든 경우에서 ESMFold보다 우수한 성능을 보였습니다. 이는 언어 모델 기반 방법과 비교했을 때,MSA 기반 모델은 예측의 정확도와 효과를 크게 향상시킬 수 있습니다.
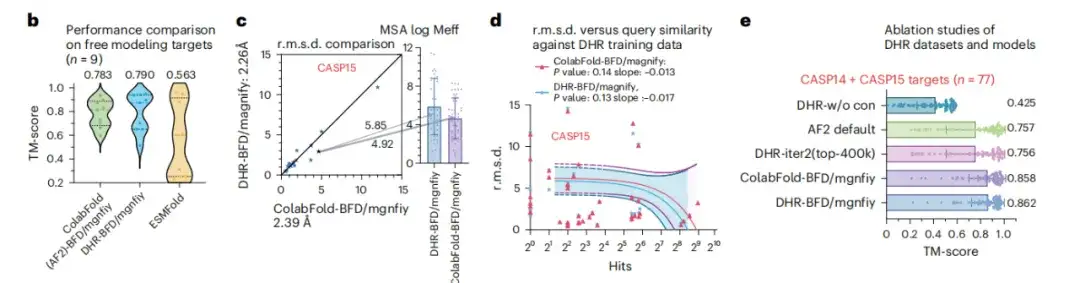
본 연구에서는 대규모 데이터 세트에서 DHR의 확장성을 연구하기 위해 BFM/MGnify를 기반으로 DHR에 대한 심층 분석을 수행했습니다. 아래 그림 b에서 볼 수 있듯이 FM 타겟의 구조를 예측하는 복잡한 시나리오에서 DHR은 더욱 의미 있는 MSA를 생성하여 두각을 나타낼 수 있으며, ColabFold 방법은 MMseqs2를 사용하여 MSA를 구성하여 TM 점수보다 0.007 더 높은 성능을 보였습니다.
그림 2c에서 DHR은 ColabFold-MMseqs2에 비해 약간의 성능 향상을 보여줍니다. 아래 그림 d는 CASP14와 SCOPe의 유사성 테스트를 거친 후 DHR이 단순히 쿼리나 히트의 결과를 기억하는 것이 아니라 모든 대상에 대한 포괄적인 유사성 평가를 수행했다는 것을 보여줍니다. 이러한 결과는 다음을 증명합니다.DHR을 사용하면 다양성이 높은 대규모 검색 데이터 세트에서 무질서한 단백질의 MSA를 구성할 수 있습니다.
단백질 구조 예측 분야의 젊은 세력
단백질 구조 예측이 약물 개발 및 항체 설계와 같은 응용 분야에서 중요한 역할을 한다는 것은 의심의 여지가 없습니다. AI는 단백질 구조 예측의 정확도가 제한적이라는 역사적 문제를 해결하는 열쇠가 될 수도 있습니다. 이 핵심 분야에서 국내 과학 연구진은 점차 백학파의 흐름을 형성하고 있으며, 떠오르는 젊은 연구자들은 무시할 수 없는 세력으로 자리 잡았습니다. 위의 연구 결과를 주도한 리위(Li Yu)와 쑨스치(Sun Siqi)는 둘 다 최고에 속합니다.

리위는 2015년 중국 과학기술대학교 북석장 엘리트 학급에서 생물학 학사 학위(우등)를 받았고, 2016년 12월 사우디아라비아 킹압둘라 과학기술대학교(KAUST)에서 컴퓨터 과학 석사 학위를 받았고, 2020년 같은 대학에서 컴퓨터 과학 박사 학위를 받았습니다.
같은 해 12월 중국으로 돌아와 홍콩 중국대학 컴퓨터공학과에 조교수로 부임하여 의료 인공지능(AIH) 그룹을 이끌었습니다. 그는 머신 러닝, 의료, 생물정보학의 교차점에서 심층 연구를 수행했으며, 팀을 이끌고 생물학과 의료 분야의 계산 문제, 특히 구조화된 학습 문제를 해결하기 위한 새로운 머신 러닝 방법을 개발했습니다.
리위는 자신이 깊이 관여하고 있는 생물학과 의료 분야에 대해 "저의 장기적인 목표는 의료 시스템을 개선하고 사람들의 건강과 복지를 향상시켜 사회에 직접적인 혜택을 주는 것입니다."라고 말했습니다.그는 2022년 포브스 아시아 "30세 이하 30인" 목록(헬스케어 및 과학)에도 선정되었다는 점도 언급할 가치가 있습니다.

쑨스치는 글로벌 단백질 구조 예측 경진대회에서 우수한 성적을 거두었으며, 현재 복단대학교 지능형 복잡시스템 기초이론 및 핵심기술 실험실과 상하이 인공지능 실험실의 청년 연구원으로 활동하고 있습니다.그는 생명과학과 자연어 처리와 같은 학제간 분야에서 딥러닝을 응용하는 연구에 전념하고 있으며, 모델의 정확도와 속도를 개선하고 모델 구현에서 발생하는 특정 문제를 해결하는 데 중점을 두고 있습니다.
단백질 예측 측면에서 그는 딥 러닝 모델을 통해 단백질의 구조와 서열을 예측하고, 서열의 패턴과 규칙성을 식별하여 단백질의 서열과 접힘을 예측하는 모델을 학습시키는 데 중점을 두고 있습니다. 이를 통해 단백질 신생 시퀀싱 및 구조 예측의 정확도와 효율성을 개선하고 약물 설계 및 질병 치료에 새로운 가능성을 창출합니다.
국내 AI4S 분야에서는 젊은 세력의 활동이 점점 더 활발해지고 있습니다. AI 기술이 단백질 구조 예측 분야에서 더욱 중요한 역할을 할 것으로 예상되지만, 앞으로의 길은 길고 험난할 것입니다. 국내 과학 연구진이 끈기 있는 탐구 정신과 혁신 능력을 보여준 것은 고무적인 일입니다. 그들은 알고리즘 최적화와 모델 구축에 열심히 노력했을 뿐만 아니라, 데이터 처리, 실험 검증 등에 대한 심층적인 연구를 수행하여 연구 결과의 과학성과 실용성을 확보했습니다. 이러한 노력은 점차 실용적인 응용 분야로 전환되어 의학 연구 개발, 생명 공학 등의 분야에 새로운 활력과 희망을 가져다주고 있습니다.
마지막으로, 학문적 공유 활동을 추천해 드리겠습니다!
Meet AI4S의 세 번째 라이브 방송에는 상하이 교통대학교 자연과학연구소와 상하이 응용수학 국가센터의 박사후 연구원인 주쯔이가 초대되었습니다. 생방송을 시청하기 위한 예약을 하려면 여기를 클릭하세요!
https://hdxu.cn/6Bjomhdxu.cn/6Bjom
