Command Palette
Search for a command to run...
라이브 미리보기: 청화대학교 박사후연구원 Li Yuzhe가 Cell/Nature 하위 저널 논문을 자세히 설명하며 유전체학 분야의 AI 응용 분야를 탐구합니다.

"AI4S를 만나다" 라이브 시리즈의 두 번째 에피소드가 8월 21일 오후 7시에 시작됩니다! HyperAI는 청화대학교 장창펑 연구실의 박사후 연구원인 리위저를 초대하여 공간 전사체학 및 단일 세포 오믹스 연구 분야의 AI 방법을 공유하게 되어 영광입니다.
첫 번째 저자인 Li Yuzhe 박사의 연구 결과인 "세포-세포 상호작용을 인식하는 세포 임베딩을 통한 단일 세포 해상도 공간 전사체 데이터에서 조직 모듈 발견"이 Cell Systems에 게재되었습니다.그는 또한 이 연구에서 제안된 혁신적인 방법, 즉 그래프 오토인코더 딥러닝 프레임워크를 기반으로 한 인공지능 알고리즘 SPACE를 소개할 것입니다.
또한,이위저 박사는 변분 자동 인코더 딥러닝 프레임워크를 기반으로 한 인공지능 알고리즘 연구에 참여했습니다. 스케일렉스,이 논문은 "이질적인 데이터 세트를 공통 세포 임베딩 공간으로 투영하여 온라인 단일 세포 데이터 통합"이라는 제목으로 Nature Communications에 게재되었습니다. 그는 또한 생방송을 통해 이 성과의 배경에 있는 연구 아이디어를 모든 사람과 공유할 것입니다.
이벤트 세부 정보

주제를 공유하세요
게놈학에서 AI 응용 프로그램 탐색: 공간 전사체 데이터 표현 알고리즘 SPACE를 예로 들어보자
소개
생명과학 연구와 인공지능(AI) 기술의 급속한 발전으로 AI는 생물의학 연구에서 점점 더 중요한 역할을 수행하고 있으며, "과학을 위한 AI"라는 새로운 학제간 연구 패러다임이 생겨나고 있습니다.
이 생방송에서는 유전체학 연구, 특히 공간 전사체학 및 단일 세포 오믹스 연구에서 인공지능 방법을 공유합니다.
청중에게 주는 혜택
1. 게놈 연구에서 인공지능 방법의 개발 과정과 최첨단 진행 상황에 대한 사전 이해를 얻습니다.
2. 생성 모델을 기반으로 한 단일 세포 오믹스 데이터 통합 알고리즘인 SCALEX를 이해합니다.
3. 그래프 신경망과 세포 군집 개념을 기반으로 한 공간 전사체 데이터 표현 알고리즘 SPACE를 이해합니다.
논문 리뷰
HyperAI는 이전에 "세포-세포 상호작용을 인식하는 세포 임베딩을 통한 단일 세포 해상도 공간 전사체 데이터에서 조직 모듈 발견"이라는 제목의 연구 논문을 해석하여 공유했으며, 이 논문의 첫 번째 저자는 Li Yuzhe 박사입니다.
연구 하이라이트
* 공간 전사체 데이터를 위한 인공지능 분석 도구인 SPACE를 개발했습니다. 이 도구는 단일 세포 분해능으로 공간 세포 유형을 식별하고 공간 전사체 데이터에서 조직 모듈을 발견할 수 있습니다.
* SPACE는 세포 유형 식별 및 조직 모듈 발견 측면에서 다른 도구보다 훨씬 우수한 성능을 보이며, 특히 여러 세포 유형이 포함된 복잡한 조직에서 그 성능이 뛰어납니다.
* SPACE는 세포 커뮤니티, 즉 인식 가능한 경계를 가진 공간적으로 동질적인 세포 유형으로 구성된 조직 모듈을 정의하고 발견합니다.
* 세포 공동체는 이를 구성하는 세포들 간의 유사한 상호작용 네트워크로 정의되며, 이를 사용하여 세포 통신에 대한 리간드-수용체 기반 추론을 구체화할 수 있습니다.
* SPACE는 공간적으로 인접한 세포 간의 상호작용이 세포 유형과 조직 모듈의 생물학적 기능에 어떤 영향을 미치는지 이해하기 위한 대규모 공간 전사체 연구에 사용될 수 있습니다.
데이터셋 수집
SPACE의 역량을 검증하기 위해 이 연구에서는 다양한 데이터 세트를 사용했습니다. 다운로드 주소:
모델 아키텍처: 세포 간 상호 작용을 인식하는 세포 내장 모델
SPACE는 그래프 자동 인코더 프레임워크를 사용하여 공간 전사체 데이터에서 각 세포의 유전자 발현 정보와 공간적 이웃 세포와의 상호작용 정보를 기술하는 저차원 세포 임베딩을 학습합니다(따라서 세포 임베딩을 세포-세포 상호작용 인식 세포 임베딩이라고 합니다). 이러한 세포 임베딩을 기반으로 SPACE는 클러스터링 알고리즘을 사용하여 공간 세포 하위 유형을 식별하고 조직 모듈을 발견합니다.
아키텍처 관점에서 SPACE 모델은 인코더(3층 그래프 주의 네트워크), 이웃 그래프 디코더, 유전자 발현 디코더의 세 부분으로 구성됩니다. 다음 그림은 모델의 전반적인 프레임워크를 보여줍니다.
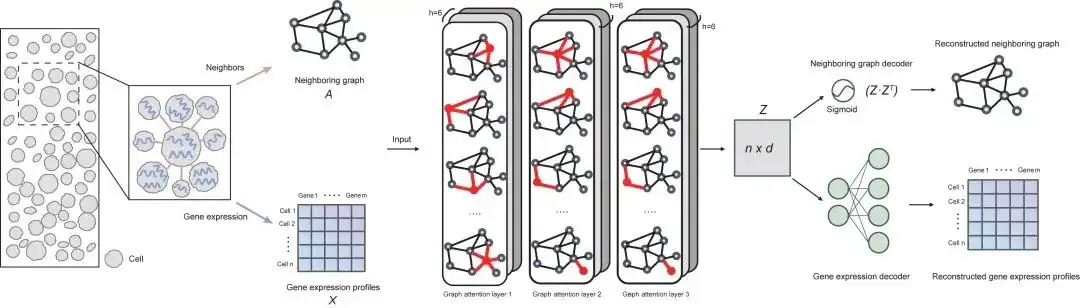
성과 평가
* SPACE는 ST 데이터 세트의 공간 정보를 기반으로 생물학적으로 구별되는 세포 유형을 식별할 수 있습니다.
* SPACE는 ST 데이터에서 공간적 정보를 제공하는 세포 유형을 구별하는 데 현재 사용 가능한 도구보다 성능이 뛰어납니다.
* SPACE는 조직 모듈 발견에 있어서 최첨단 도구보다 우수한 성능을 발휘합니다.
칭화대학교 장창펑 연구실

장창펑의 연구실은 청화대학교 생명과학부에 소속되어 있습니다. 또한 이곳은 청화-베이징 대학 생명과학 공동 센터와 베이징 구조생물학 선진혁신 센터의 중요한 부분입니다.
이 연구실의 연구는 구조 생물학, 유전체학, 머신 러닝, 빅데이터 분석과 같은 학제간 분야에 중점을 두고 있습니다. 주요 연구 방향은 구조 생물학과 시스템 생물학을 결합하고, 계산적 및 실험적 방법을 결합하여 생물학적 거대 분자(예: 단백질, RNA, DNA)의 구조와 기능 관계를 해석하고, 이들의 상호작용 네트워크를 재구성하고, 단백질과 RNA 구조의 변화 및 비정상적인 거대 분자 상호작용과 관련된 복잡한 질병(암 및 감염성 질환 포함)의 병인과 가능한 치료법을 발견하는 것입니다.
이 연구실은 독특한 단백질 및 RNA 구조 모델링, 차세대 시퀀싱을 기반으로 한 RNA 구조 측정, 고처리량 RNA-단백질 상호작용 검출 기술, 강력한 계산 및 실험 플랫폼을 갖추고 있어 연구자들의 최첨단 연구를 발전시키고 있습니다.
AI4S 라이브 시리즈를 만나보세요
HyperAI(hyper.ai)는 데이터 과학 분야에서 중국 최대의 검색 엔진입니다. AI for Science의 최신 과학 연구 결과에 초점을 맞추고 Nature, Science 등 최고 저널에 실린 학술 논문을 실시간으로 추적합니다. 지금까지 100편이 넘는 AI for Science 논문의 해석을 완료했습니다.
또한, 우리는 중국 유일의 AI for Science 오픈소스 프로젝트인 awesome-ai4s도 운영하고 있습니다.
* 프로젝트 주소:
https://github.com/hyperai/awesome-ai4s
AI4S의 대중화를 더욱 촉진하고, 학술 기관의 과학 연구 결과의 보급 장벽을 더욱 낮추고, 이를 더 광범위한 산업 학자, 기술 애호가 및 산업 단위와 공유하기 위해 HyperAI는 "AI4S를 만나보세요" 영상 칼럼을 기획하여 AI for Science 분야에 깊이 관여하는 연구자 또는 관련 단위를 초대하여 영상 형식으로 연구 결과와 방법을 공유하고, 과학 연구 진행 및 홍보 및 구현 과정에서 AI for Science가 직면한 기회와 과제를 함께 논의하여 AI for Science의 대중화와 보급을 촉진합니다.