Command Palette
Search for a command to run...
특징 선택 전략: 유방암 바이오마커를 감지하는 새로운 방법 찾기

내용을 한눈에 보기:마이크로RNA(microRNA)는 짧은 단일 가닥의 비번역 RNA 전사본입니다. 이러한 분자는 다양한 악성 종양에서 통제되지 않는 성장을 보이므로 최근 몇 년 동안 많은 연구를 통해 암 진단을 위한 신뢰할 수 있는 바이오마커로 확인되었습니다. 다양한 병리학적 분석 중에서, 차등 발현 분석은 주요 바이오마커를 탐지하는 효과적인 방법으로 간주되는 경우가 많습니다. 이탈리아 나폴리 페데리코 2세 대학의 연구진은 머신러닝을 기반으로 한 특징 선택 전략이 탐지에 더 효과적일 수 있다고 제안했으며, 발견된 20개의 마이크로RNA를 유방암 진단 바이오마커로 사용할 것을 권장했습니다.
키워드:특징 선택 마이크로RNA 유방암
본 기사는 HyperAI WeChat 공개 플랫폼에 처음 게재되었습니다~
국가보건위원회가 발표한 2022년판 '유방암 치료 지침'에 따르면,유방암은 여성에게 흔한 악성 종양 중 하나이며, 여성 악성 종양 중 발병률이 1위를 차지합니다.세계보건기구의 통계에 따르면, 2020년에 전 세계적으로 230만 명의 여성이 유방암 진단을 받았습니다. 치료방법의 지속적인 개선으로조기 유방암의 5년 생존율은 90% 또는 그 이상에 도달할 수 있습니다. 그러므로 조기 유방암의 정확한 진단이 특히 중요합니다.
microRNA 발현의 변화는 생물학에서 많은 핵심 역할을 하는 것 외에도 다양한 암과 관련이 있으므로 신뢰할 수 있는 추정 진단 바이오마커로 사용될 수 있습니다. 이탈리아 나폴리 페데리코 2세 대학의 연구원들은 머신 러닝을 사용하여특징 선택 전략을 사용하여 세 가지 방법의 안정성과 분류 성능을 분석함으로써,유방암 특이적 진단 바이오마커 패널을 확보하고, 유방암 질환의 발병 및 진행에 관여하는 것으로 추정되는 핵심 유전자를 발견했습니다.
현재, 본 연구 결과는 생물정보학 및 생물통계학 분야 계산 지능 방법에 관한 제18회 학술대회(CIBB 2023) 논문집에 "강력한 특징 선택 전략을 통한 유방암의 추정 진단 바이오마커로서의 마이크로RNA 패널 감지"라는 제목으로 게재되었습니다.

연구 결과는 CIBB 2023에 게재되었습니다.
서류 주소:
https://www.researchgate.net/publication/372083934
실험 개요
이 연구에서 연구자들은 세 가지 특징 선택 방법(이득률, 랜덤 포레스트, 지원 벡터 머신 재귀적 특징 제거)의 도움으로 진단 분자 조합을 더 효율적으로 추출할 수 있다는 것을 발견했습니다. 그들은 20개의 마이크로RNA 패널을 공개했는데, 그 중 hsa-mir-337, hsa-mir-378c, hsa-mir-483은 현재 유방암 진단 바이오마커 중에서 의학계에서 널리 주목받지 못했습니다. 이 방법은 건강한 샘플과 종양 샘플을 구별할 수 있습니다. 일반적으로 사용되는 차등 표현 방식과 비교했을 때 분류 성능이 더 뛰어나고, 쉽게 과소평가되거나 무시되는 특징을 더 쉽게 식별할 수 있습니다.
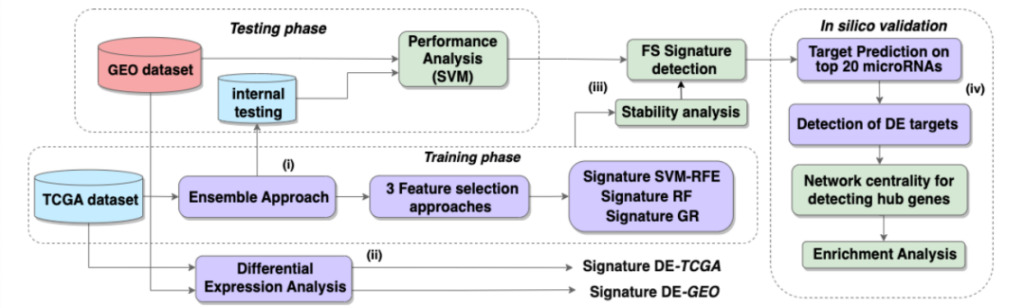
그림 1: 파이프라인 개요
워크플로는 4가지 주요 단계로 구성됩니다.
(나) 훈련 TCGA 하위 집합에 대한 앙상블-FS 계산
(ii) TCGA/GEO 데이터 세트의 차등 발현 분석
(3) 차등 발현 분석과 특징 선택 결과의 분류 성능을 비교하고, 특징 선택 방법의 안정성을 평가합니다.
(4) 컴퓨터 시뮬레이션을 통해 선택된 시그니처의 상위 20개 microRNA를 검증하고 허브 유전자 표적을 감지했습니다.
실험 세부 사항
데이터세트
실험 데이터 소스에는 두 개의 채널이 포함됩니다.미국 GDC 공식 웹사이트와 Gene Expression Omnibus(GEO) 데이터 저장소(GSE97811)의 TCGA-BRCA 프로젝트.
실험팀은 GDC TCGA-BRCA 프로젝트에서 총 1,881개의 microRNA-Seq 데이터를 수집하여 8:2의 비율로 훈련 세트와 테스트 세트로 나누었습니다. 이 데이터는 300개의 고형 원발 종양 샘플(T)과 101개의 정상 인접 종양(NAT) 샘플(관형 및 소엽형 유방 조직 모두)과 관련이 있습니다. 특징 선택을 적용하기 전에,이러한 데이터는 분산 안정화 정규화를 거쳤습니다.
동시에 연구자들은 GEO 데이터베이스(GSE97811)에서 2,565개의 마이크로RNA가 포함된 마이크로어레이 데이터 세트를 다운로드했습니다.이 실험의 검증 세트로 사용됩니다.데이터 세트에는 정상 샘플 16개와 종양 샘플 45개가 포함되어 있으며, 이에 대한 데이터 투입이 수행됩니다.
GEO 데이터(이 실험의 검증 세트)는 성숙한 마이크로RNA의 발현을 포함하고, TCGA 데이터(이 실험의 훈련 세트 및 테스트 세트)는 전구체 형태를 포함하고 있기 때문에, 연구진은 데이터를 통합하기 위해 GEO 데이터 샘플에서 반대 가닥보다 평균 카운트 값이 높은 대체 성숙한 마이크로RNA만을 선택했습니다. 동시에, 마이크로RNA의 이름은 해당 전구체 형태의 이름으로 변환되었습니다.
이 과정을 거친 후,GEO 데이터(검증 세트)의 차원은 1,361개의 microRNA로 축소되었고, 총 20,404개의 유전자를 포함하여 해당 TCGA RNA-Seq 데이터도 수집되었습니다.
1. 특징 선택 방법 및 앙상블 프로그램 적용
연구자들은 차등 발현 분석과 비교하기 위해 세 가지 특징 선택 방법을 선택했습니다.그들은 이득 비율, 랜덤 포레스트, SVM-RFE(지원 벡터 머신 재귀적 특징 제거).500개의 microRNA-Seq 발현 TCGA 데이터 하위 집합에 세 가지 방법을 적용하여 정상 샘플과 종양 샘플을 구별할 수 있는 강력한 기능 패널을 식별했습니다. 관찰 결과, 데이터는 8:2의 비율로 훈련 세트와 테스트 세트로 나뉘었고, 그런 다음 데이터를 다시 샘플링하고 부트스트래핑하여 데이터 섭동 앙상블 절차에 맞게 만들었습니다. 각 계산은 "중요도 점수"의 내림차순으로 정렬된 500개의 microRNA 벡터를 반환합니다.
|비고:중요도 점수는 알고리즘이 계산한 분류에서 각 기능의 영향력을 나타냅니다.
중요도 점수가 높을수록 기능에 할당된 순위는 낮아집니다.그런 다음 연구자들은 집계 절차를 사용하여 각 기능 선택 방법에 대한 합의 서명을 도출하고 궁극적으로 각 microRNA 그룹에 대한 상위 200개 기능을 유지했습니다.
2. 안정성 시험
Kuncheva 지수(KI)와 중복 유전자/특징 비율(POG)은 특성 선택 방법의 일관성을 평가하는 데 사용되었으며, Stot 통계(KI의 쌍별 측정)는 모든 방법 간의 안정성을 확인하는 데 사용되었습니다.이러한 통계는 서명 길이가 늘어남에 따라 계산됩니다.특징의 개수는 2에서 시작하여 200으로 끝나며, 재계산할 때마다 2단위씩 증가합니다.

Stot 통계 공식
3. 차등 발현 분석 및 DE-signature
TCGA 데이터 세트(microRNA-Seq 및 RNA-Seq 포함)에 대해 정확한 검정을 사용하여 원시 계수부터 시작하여 FDR <= 0.01 및 Log2FC 임계값 |0.5|를 사용하여 DE 특징을 유지한 채 차등 발현 분석을 수행했습니다.DE-microRNA의 특징을 얻기 위해 Log2FC 값을 절대값으로 변환하고, microRNA(처음 200개 특징 유지)를 절대값(Log2FC)의 내림차순으로 정렬했습니다.
GEO 검증 세트는 Limma를 사용하여 차등적으로 표현되었으며, 이 데이터 세트에서 DE 서명을 얻기 위한 매개변수와 절차는 TCGA 데이터 세트와 일치했습니다.
4. 분류 성능 분석
각 서명이 건강한 사람과 암 환자를 구별하는 능력을 확인하려면,연구진은 테스트 하위 집합(TCGA)과 검증 집합(GEO) 모두에서 4가지 특징(특징 선택 패널과 차등 발현 패널 포함)에 대한 예측 분석을 수행했습니다.
마지막으로, 각 폴드와 각 시그니처의 여러 길이에 대해 평균 정확도(ACC), K 통계량(KK), 매튜스 상관 계수(MCC)가 계산됩니다.
5.SVM-RFE 마이크로RNA 시그니처 타겟 검출
microRNA의 잠재적인 유전자 표적을 식별하려면연구자들은 다음과 같은 작업을 수행했습니다.
1. 상위 20개 SVM-RFE 마이크로RNA는 종양 샘플에서 상향 조절되었는지 하향 조절되었는지에 따라 분류되었습니다.
2. RNA-Seq 데이터에 대해 차등 발현 분석을 수행하여 차등 발현 유전자(FDR <= 0.05)를 검출했습니다.
3. 스피어만 상관분석을 적용하여 차등적으로 발현되는 유전자와 마이크로RNA 발현을 비교하였고, 하향 마이크로RNA와 음의 상관관계를 갖는 상향 유전자와 상향 마이크로RNA와 음의 상관관계를 갖는 하향 유전자만 유지하였다(rho <= -0.5).
4. 검증된 모든 microRNA 유전자 표적을 수집하였고 DE-상관관계를 보인 표적만 보관했습니다.
6. 네트워크 중심성 및 허브 유전자 식별
선택된 조절 불량 유전자의 상관 행렬(Spearman)이를 사용하여 그래프 구조의 유전자 네트워크를 구축합니다.클라인버그 허브 중심성 점수가 75 이상, rho가 0.8 이상 또는 rho가 -0.6 미만인 허브 유전자는 유지되었습니다. REACTOME 데이터베이스에서 가장 풍부한 경로를 탐색하기 위해 허브 유전자에 대한 유전자 풍부 분석(ORA)을 수행했습니다. FDR 조정 pValue 임계값은 0.005로 설정되었습니다.
실험 결과
실험 결과, 세 가지 특징 선택 방법을 적용한 후 중요도 점수의 내림차순으로 500개의 microRNA 시그니처가 반환되었고, 집계 후 3개의 합의 패널이 얻어졌습니다. 특히 상위 3개의 microRNA(hsa-mir-139, hsa-mir-96, hsa-mir-145)가 모든 패널에 나타났으며, 이는 이러한 분자가 종양 샘플과 건강한 샘플을 구별하는 데 중요하다는 것을 보여줍니다.
결론 1: SVM-RFE는 가장 높은 안정성을 가지고 있습니다.
합의 패널에서 KI와 POG를 계산한 결과,SVM-RFE 방법은 가장 안정적이며, 시그니처 길이가 20개 특징에 도달했을 때 가장 두드러집니다. 마찬가지로 Stot 지수의 결과도 SVE-RFE 방법이 가장 높은 안정성을 가지고 있음을 보여줍니다.
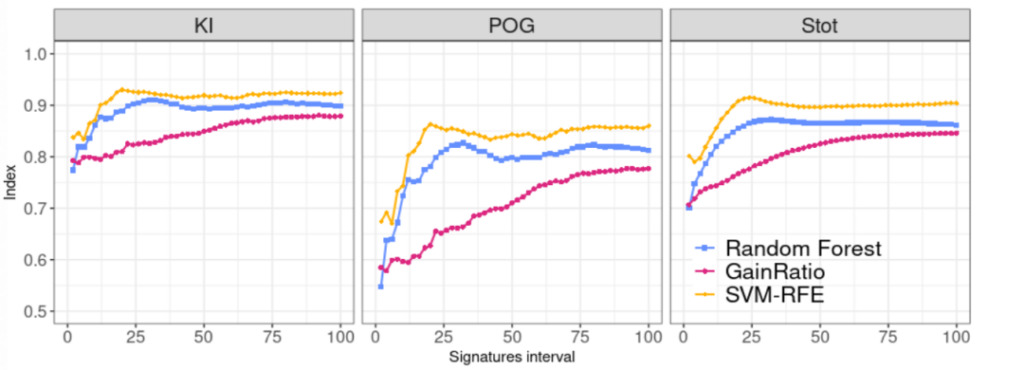
그림 2: 세 가지 특징 선택 방법의 안정성 지수 비교
파란색:랜덤 포레스트
분홍색:이득 비율
노란색:SVM-RFE(지원 벡터 머신 재귀적 특징 제거)
|결론 2: SVM-RFE 시그니처는 분류에서 차등 발현 시그니처보다 더 나은 성능을 보입니다.
모든 개별 패널에 대한 분류 성능 분석 후, 테스트 세트(TCGA)와 검증 세트(GEO) 모두 SVM-RFE로 얻은 시그니처가 가장 높은 예측 능력을 가지고 있음을 보여주었습니다.
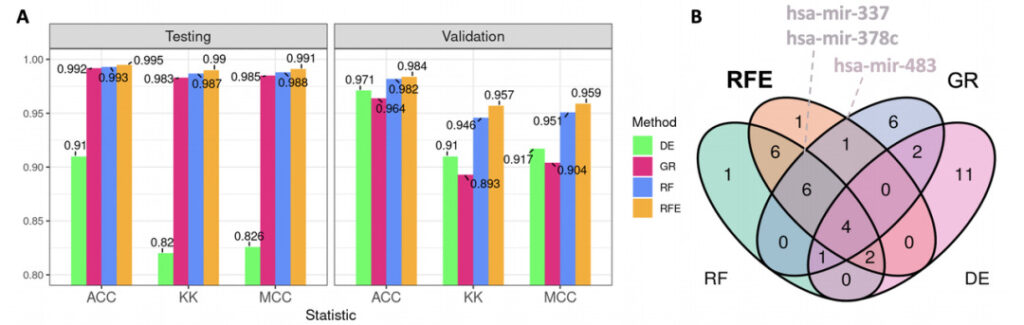
그림 3: 상위 20개 microRNA의 분류 성능 및 벤 다이어그램
에이:막대 그래프는 테스트 하위 집합과 외부 검증 GEO 데이터 집합에서 계산된 평균 통계를 보여줍니다.
ACC:정확성
케이케이:K 통계량
MCC:매튜스 상관 계수
녹색:DE(차등 발현 분석, 본 실험의 제어 방법)
분홍색:GR(이득 비율)
파란색:RF(랜덤 포레스트)
오렌지색:RFE(SVM-RFE, 지원 벡터 머신 재귀적 특징 제거)
비:그림 4: 각 시그니처에 대한 상위 20개 microRNA의 벤 다이어그램. SVM-RFE 패널의 상위 20개에서 흥미로운 microRNA 몇 가지를 hsa-mir-337, hsa-mir-378c, hsa-mir-483으로 표시했습니다. 이 세 가지 마이크로 RNA는 세 가지 특징 선택 방법 모두에서 나타났지만, 유방암에 대한 최근 연구에서는 진단적 증거로서의 신뢰성이 아직 완전히 결정되지 않았습니다.
|결론 3: 네트워크 분석은 질병 진화의 잠재적 핵심 유전자를 밝혀냅니다.
실험 결과, CDC25, TPX2, KIF18B는 다양한 유형의 암과 삼중 음성 유방암 환자의 줄기 세포에서 높게 발현되고, TGFBR2의 다운레귤레이션은 암 진행과 관련이 있는 것으로 나타났습니다.
MicroRNA: 유방암 조기 검진을 위한 또 다른 이상적인 후보
기존의 유방암 검진 방법은 여전히 X선 영상과 조직 생검에 기반을 두고 있어, 암 유전체 전체에 대한 보다 깊고 포괄적인 이해를 확립할 수 없습니다. 이 방법은 침습성이 매우 높고 비용이 많이 들며 부작용이 발생하기 쉬울 뿐만 아니라, 양성 오류나 음성 오류 결과가 나오는 경우가 많습니다. 유방암 조기 검진의 정확성을 높이고 환자 경험을 개선하는 것은 어렵습니다.유방암의 부담을 해소하기 위해 새로운 전략을 개발할 필요가 여전히 있습니다.
1993년 처음 발견된 이래로 micorRNA는 암에 대한 우리의 이해를 지속적으로 심화시켜 왔으며 유방암 진단을 위한 신뢰할 수 있는 바이오마커로서 큰 잠재력을 보여주었습니다.
마이크로RNA는 길이가 약 19~25nt인 작은 비코딩 RNA로, 다양한 표적 유전자를 조절할 수 있습니다.다양한 생물학적, 병리학적 과정의 조절에 관여합니다.암의 형성과 발달 과정을 포함하여 임상에서 유방암 검진을 위한 주류 진단 방법인 현재 엑스선 영상과 조직 생검의 한계를 보완할 것으로 기대됩니다.
그러나 microRNA의 성숙한 임상적 적용은 아직 완전히 개발되지 않았으며, microRNA 사용에 대한 안전성 평가 시스템도 아직 확립되지 않았습니다.마이크로RNA가 암 진단의 주요 기준이 되려면 아마도 시간이 좀 걸릴 것이다.
참고문헌:
[1]https://www.who.int/zh/news-room/fact-sheets/detail/유방암
[2]https://guide.medlive.cn/guideline/25596
[3]https://www.abcam.cn/kits/micrornas-as-biomarkers-in-cancer-1
[4]https://caivd-org.cn/webfile/file/20220508/20220508153691029102.pdf
[5]https://www.sohu.com/a/318088245_100120288
본 기사는 HyperAI WeChat 공개 플랫폼에 처음 게재되었습니다~