Command Palette
Search for a command to run...
Plateforme De Conception d'ingénierie Des Protéines VenusFactory
Date
Taille
6.42 GB
Balises
Licence
Apache 2.0
GitHub
URL du document

1. Introduction au tutoriel

VenusFactory a été développé en 2025 par une équipe conjointe de l'Université Jiao Tong de Shanghai, du Laboratoire d'intelligence artificielle de Shanghai et de l'Université des sciences et technologies de Chine orientale. Les articles de recherche associés sont les suivants : VenusFactory : une plateforme unifiée pour la récupération de données d'ingénierie des protéines et l'optimisation des modèles de langage .
VenusFactory est une plate-forme unifiée conçue spécifiquement pour la communauté de l'ingénierie des protéines, visant à intégrer la récupération de données biologiques, l'analyse comparative des tâches standardisées et le réglage fin modulaire des modèles de langage protéique pré-entraînés (PLM).
La plateforme prend en charge l'exécution en ligne de commande et une interface sans code basée sur Gradio, et intègre plus de 40 ensembles de données liés aux protéines et plus de 40 PLM populaires, ce qui la rend facile à utiliser pour les chercheurs en informatique et en biologie.
Le tutoriel propose 7 modules fonctionnels :
- Formation : formation de modèle sans code, prend en charge plus de 40 grands modèles et utilise des ensembles de données privés pour former vos propres modèles.
- Évaluation : Un outil facile à utiliser pour l’évaluation complète des performances des modèles protéiques.
- Prédiction : utilisez le modèle formé pour prédire la fonction de nouvelles séquences protéiques.
- VenusAgent : un agent d'ingénierie des protéines qui fonctionne avec DeepSeek pour permettre le calcul des protéines par IA.
- Outils rapides : version facile à utiliser, prend en charge la prédiction de mutation à échantillon zéro (évolution dirigée) et la prédiction supervisée (prédiction de fonction ou de propriété).
- Outils avancés : version personnalisée avancée, prenant en charge la prédiction de mutation à échantillon zéro (évolution dirigée) et la prédiction supervisée (prédiction de fonction ou de propriété).
- Téléchargement : Connectez-vous facilement aux données sur les protéines et prenez en charge les téléchargements multithreads à partir des principales bases de données (RCSB, UniProt...).
Les ressources informatiques utilisées dans ce tutoriel sont une seule carte RTX 4090. Le modèle utilisé dans le tutoriel est enregistré dans
/openbayes/input/input1Toutes les données sont stockées dans le répertoire/openbayes/home/VenusFactoryannuaire.
2. Étapes de l'opération
1. Démarrez le conteneur
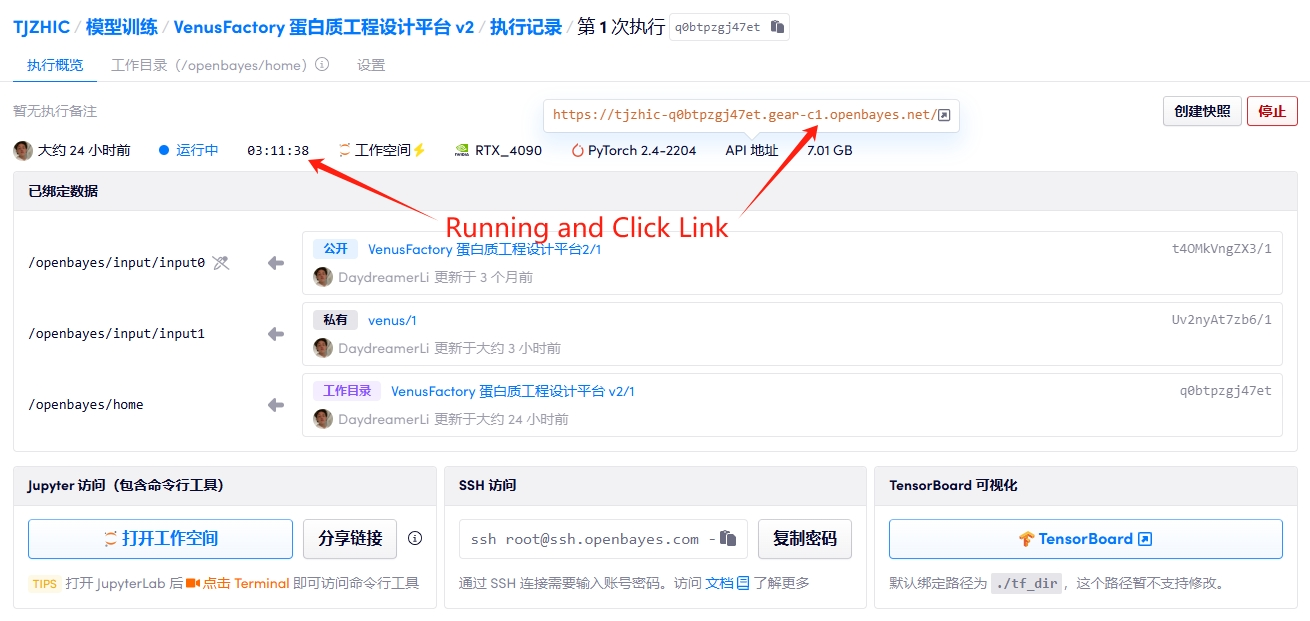
2. Manuel
Si le message « Bad Gateway » s'affiche, cela signifie que le projet est en cours d'initialisation. Veuillez patienter 1 à 2 minutes et actualiser la page.
Le guide d'utilisation « Manuel » de VenusFactory comprend actuellement quatre modules : Formation, Évaluation, Prédiction et Téléchargement.
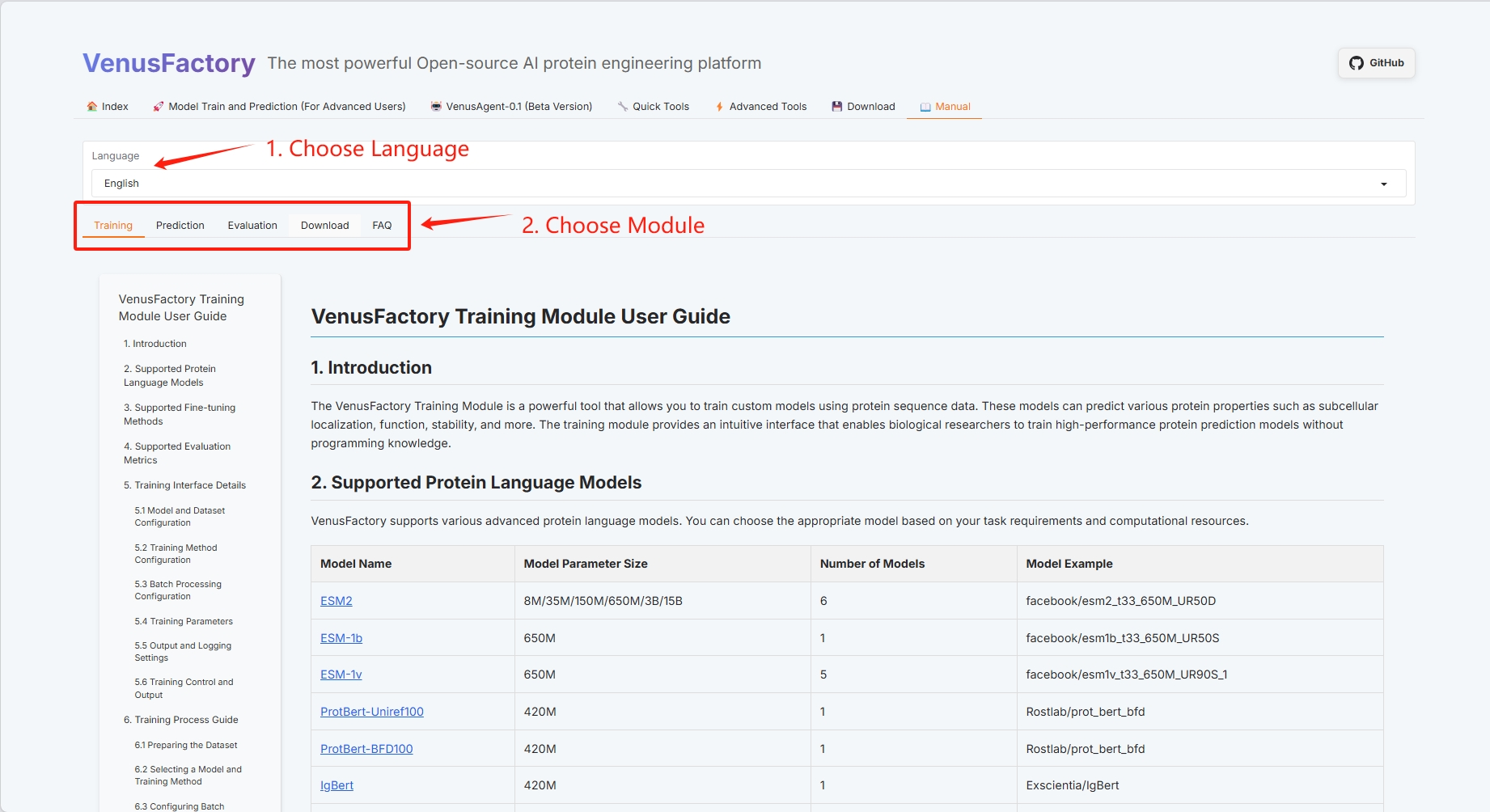
3. Démonstration d'une fonction spécifique
3.1 Formation
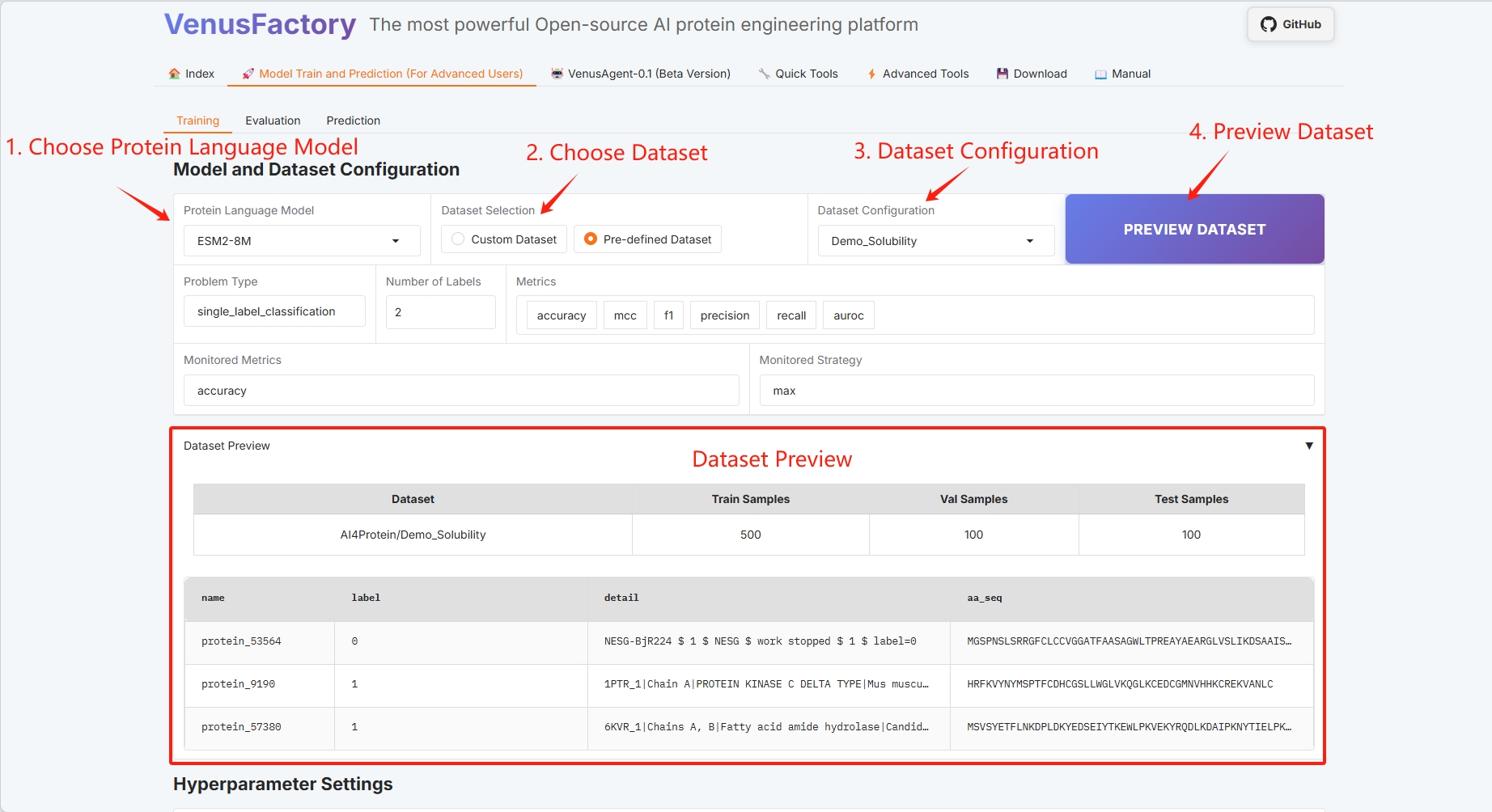
Cliquez sur le module « Formation » dans le module « Formation au modèle et à la prédiction »
- Sélectionner le modèle de langage des protéines
- Sélection des jeux de données
- Aperçu de l'ensemble de données
- Configuration de la méthode de formation (se référer au guide d'utilisation pour des informations spécifiques)
- Configuration par lots (voir le Guide de l'utilisateur pour plus de détails)
Si les paramètres du modèle sélectionné sont importants, veuillez remplacer la carte graphique par une plus grande.
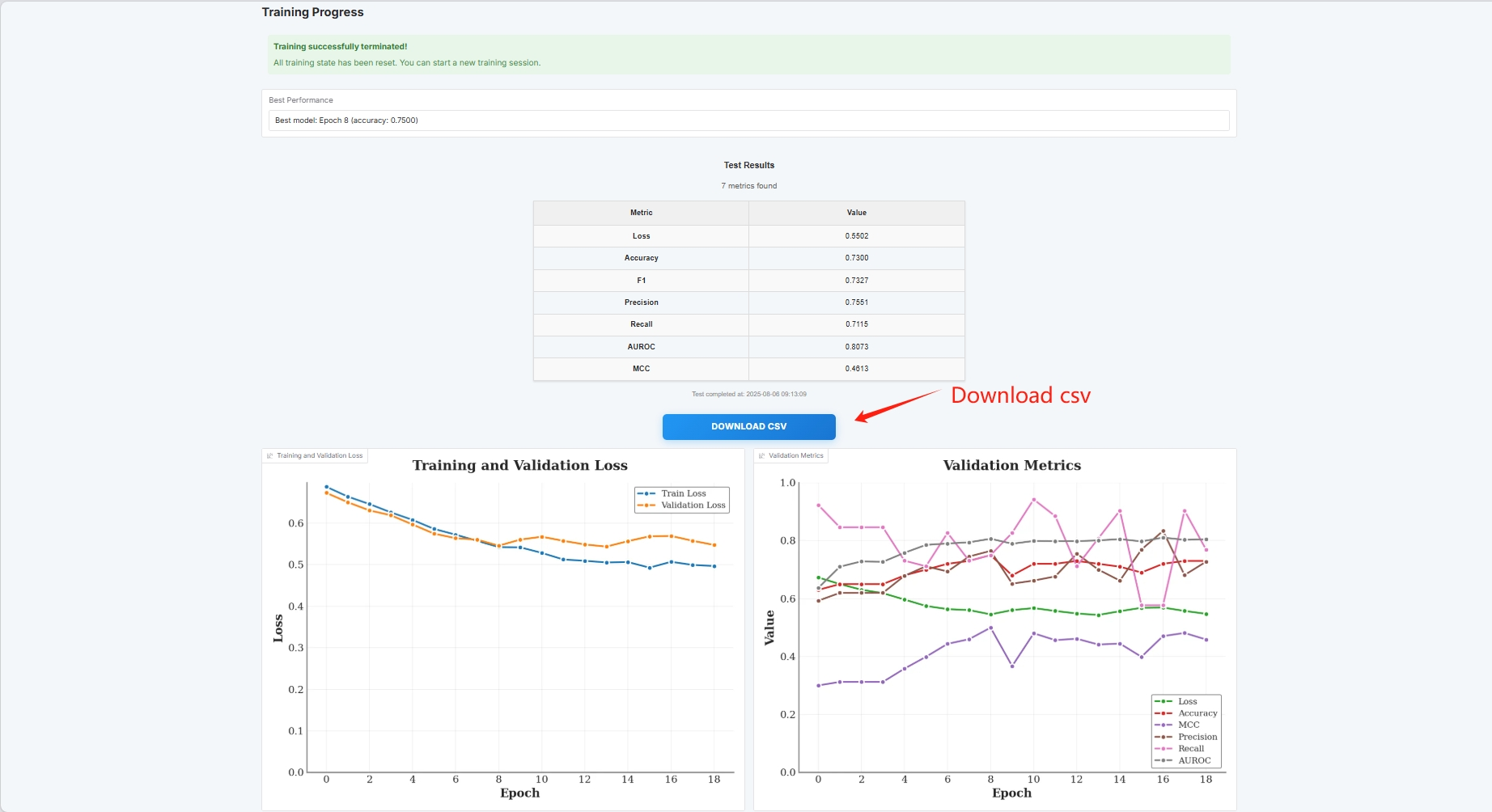
Définissez le chemin d'enregistrement du modèle de formation et cliquez sur « DÉMARRER LA FORMATION » pour démarrer la formation.
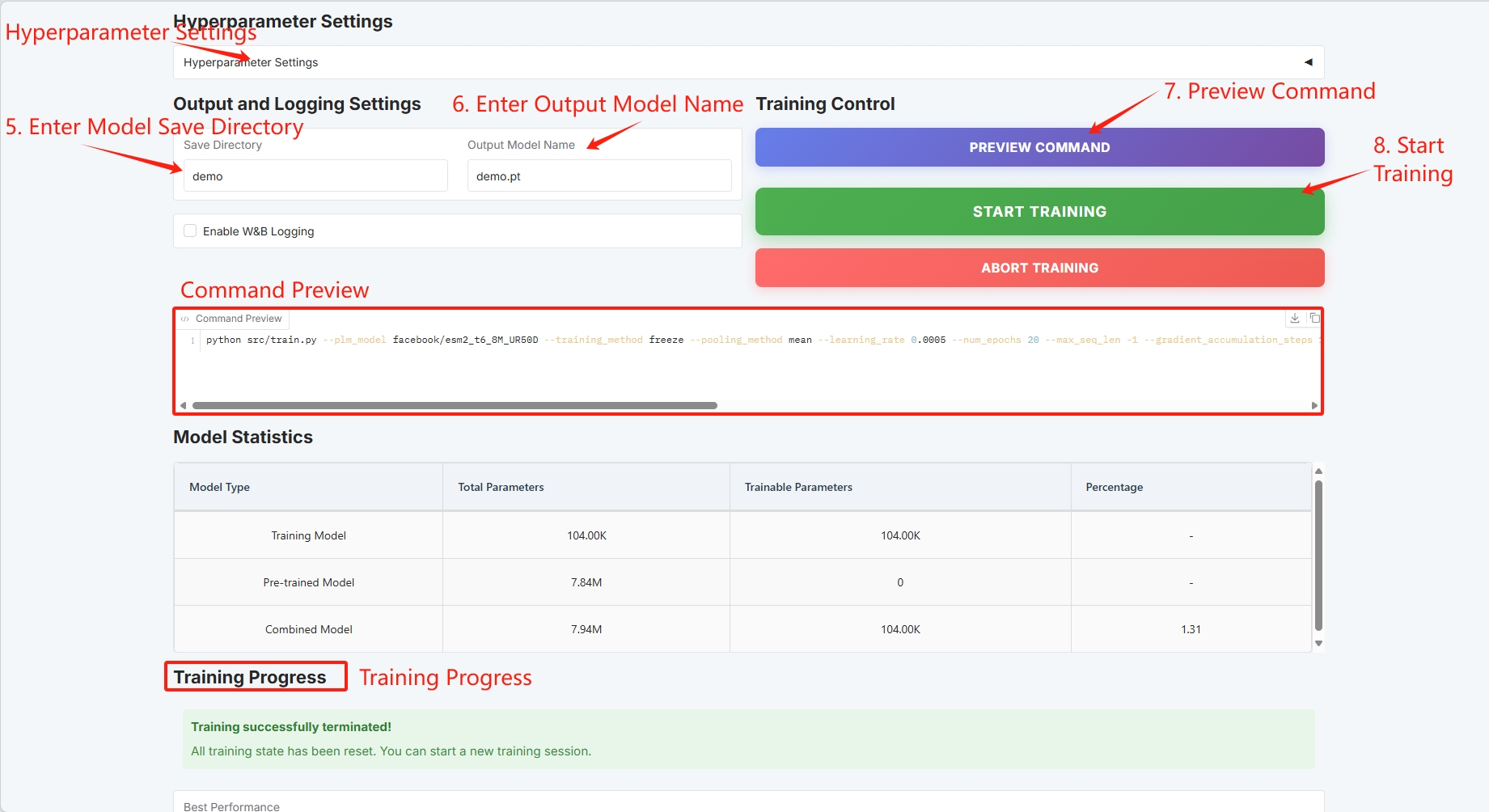
À ce stade, vous pouvez voir les paramètres d’entraînement et la courbe de perte
Si vous souhaitez utiliser votre propre jeu de données, vous pouvez utiliser la configuration Jeu de données personnalisé. Il vous suffit de renseigner le chemin d'accès à votre jeu de données (consultez la documentation du manuel pour plus de détails).
3.2 Évaluation
Cliquez sur le module « Évaluation » dans le module « Formation de modèles et de prédiction »
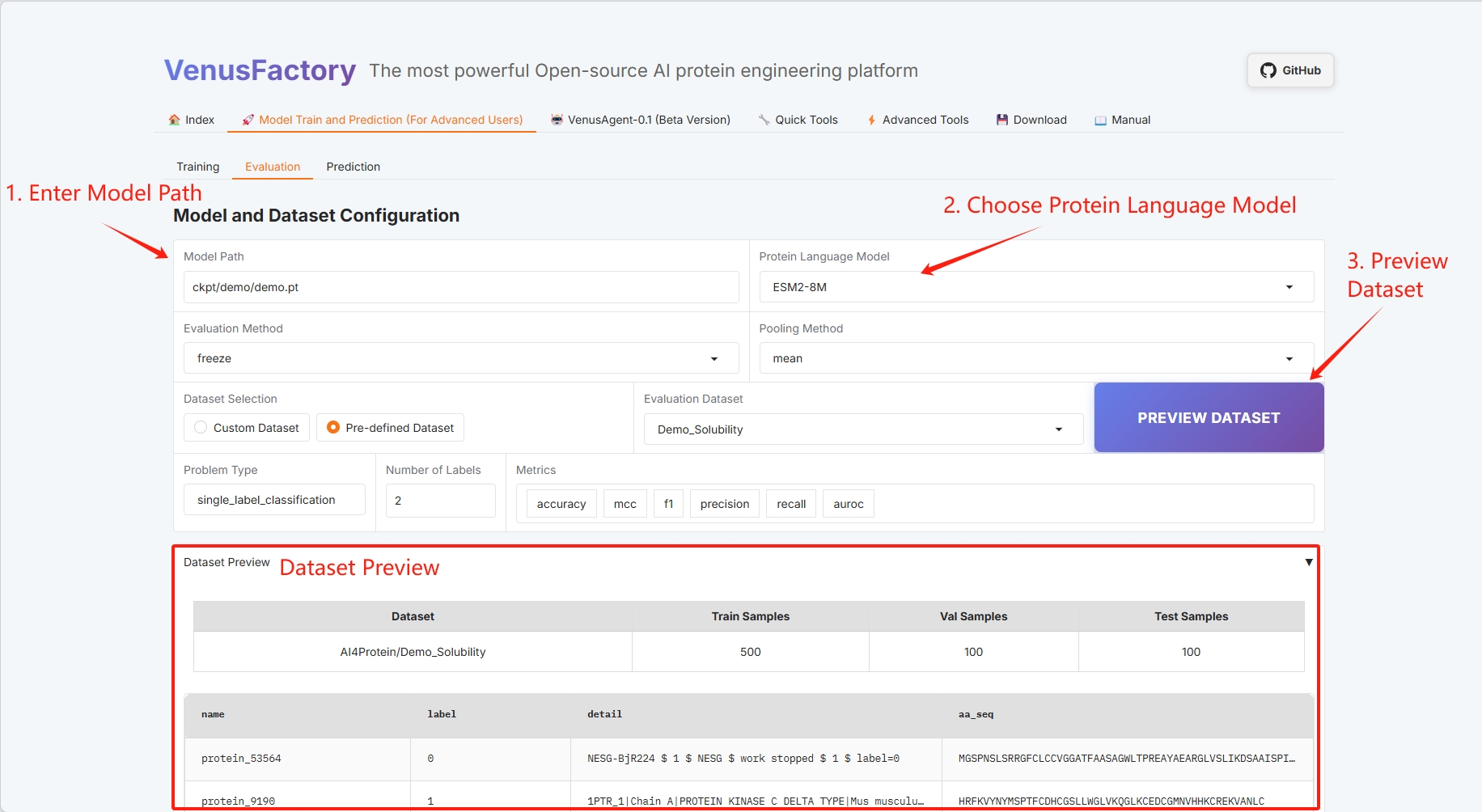
- Chemin du modèle et sélection du modèle de langage des protéines
- Méthode d'évaluation et méthode de mise en commun (se référer au guide d'utilisation pour des informations spécifiques)
- Sélection des jeux de données
- Aperçu de l'ensemble de données
- Types de questions et balises (voir le Guide de l'utilisateur pour plus de détails)
- Configuration par lots (voir le Guide de l'utilisateur pour plus de détails)
Définissez le chemin pour enregistrer le modèle formé et sélectionnez le modèle de langage protéique.
Configuration par lots, cliquez sur « DÉMARRER L'ÉVALUATION » pour démarrer la formation.
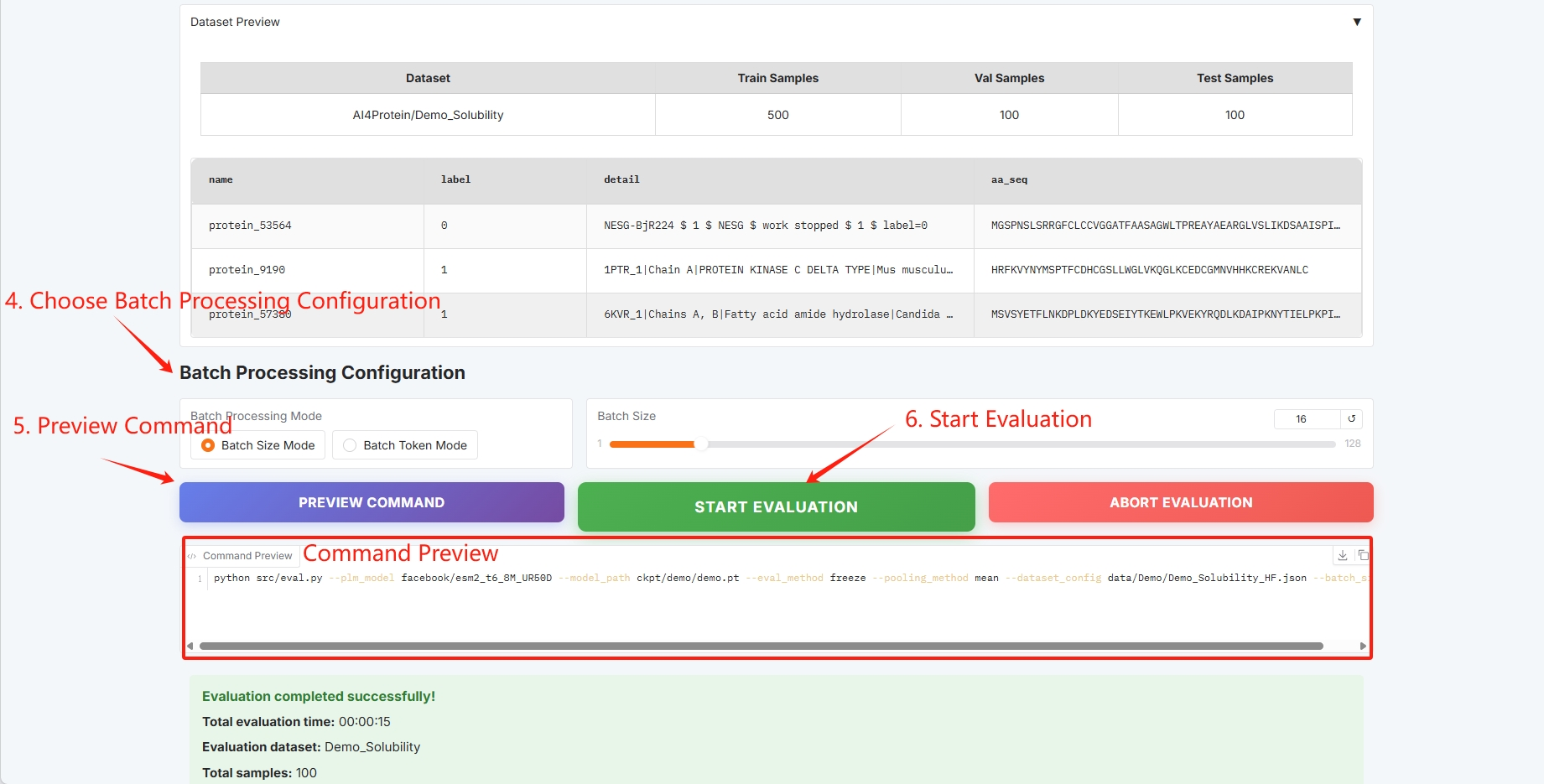
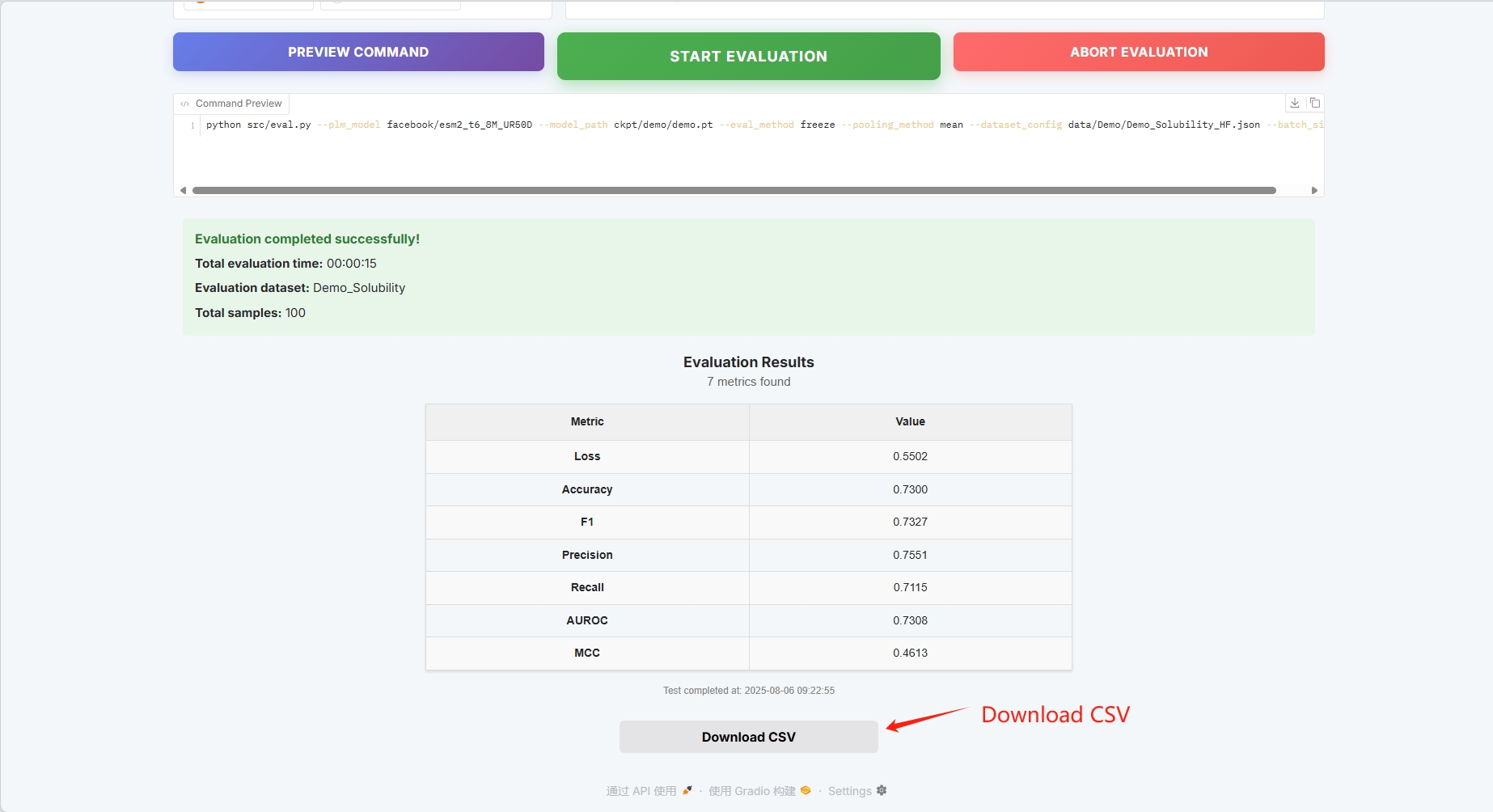
Les résultats de l'évaluation sont les suivants et peuvent être téléchargés au format CSV
Si vous souhaitez utiliser votre propre jeu de données, vous pouvez utiliser la configuration Jeu de données personnalisé. Il vous suffit de renseigner le chemin d'accès à votre jeu de données (consultez la documentation du manuel pour plus de détails).
3.3 Prédiction
Cliquez sur le module « Prédiction » dans le module « Formation de modèles et de prédiction » pour effectuer une prédiction de séquence unique et une prédiction par lots.
- Configuration du modèle
- Sélectionnez le module de prédiction (reportez-vous au guide d'utilisation pour plus de détails)
Définissez le chemin d'enregistrement du modèle de formation, sélectionnez le modèle de langage protéique et cliquez sur « DÉMARRER LA PRÉDICTION » pour démarrer la formation.
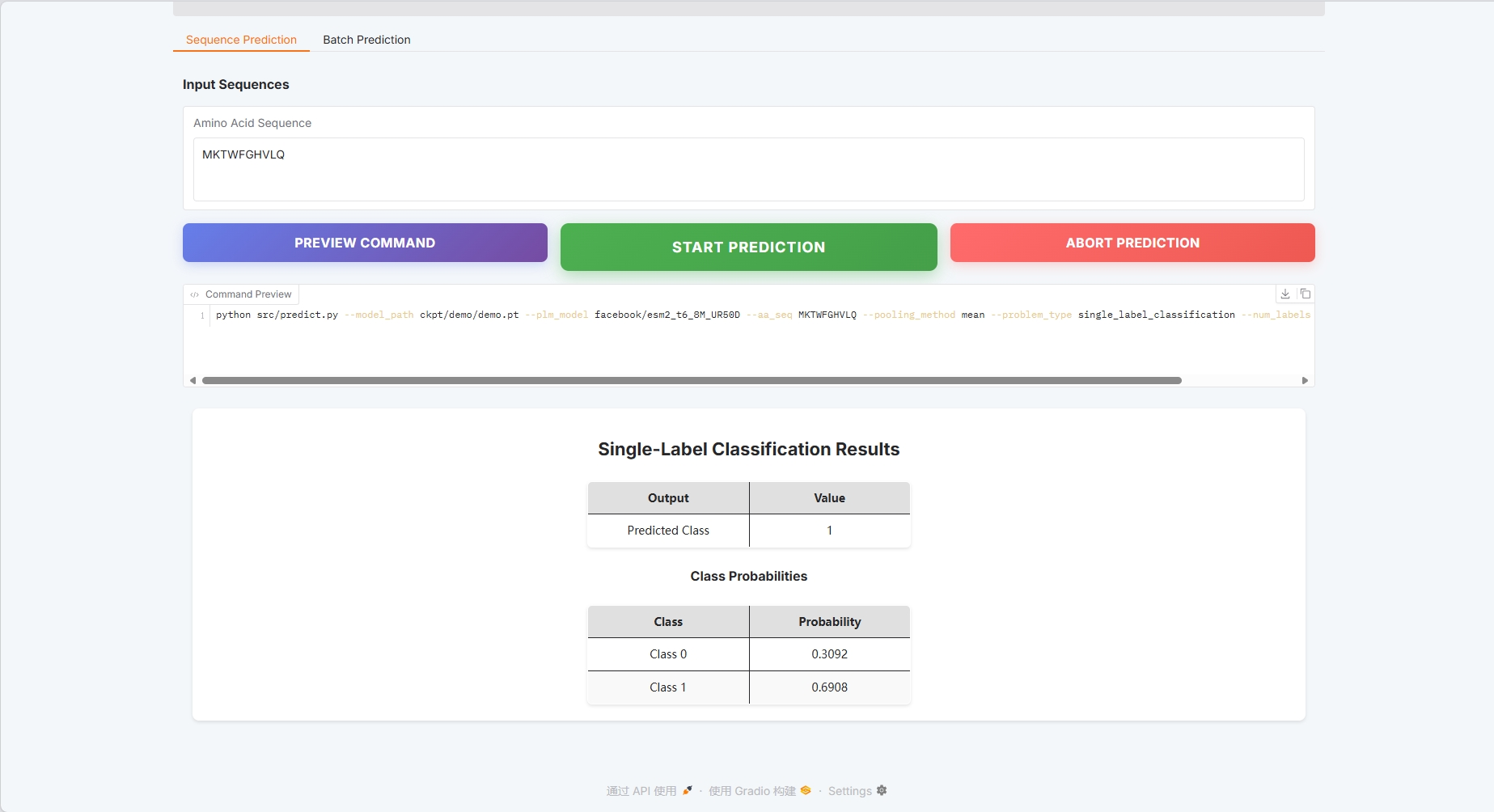
Prédiction de séquence unique
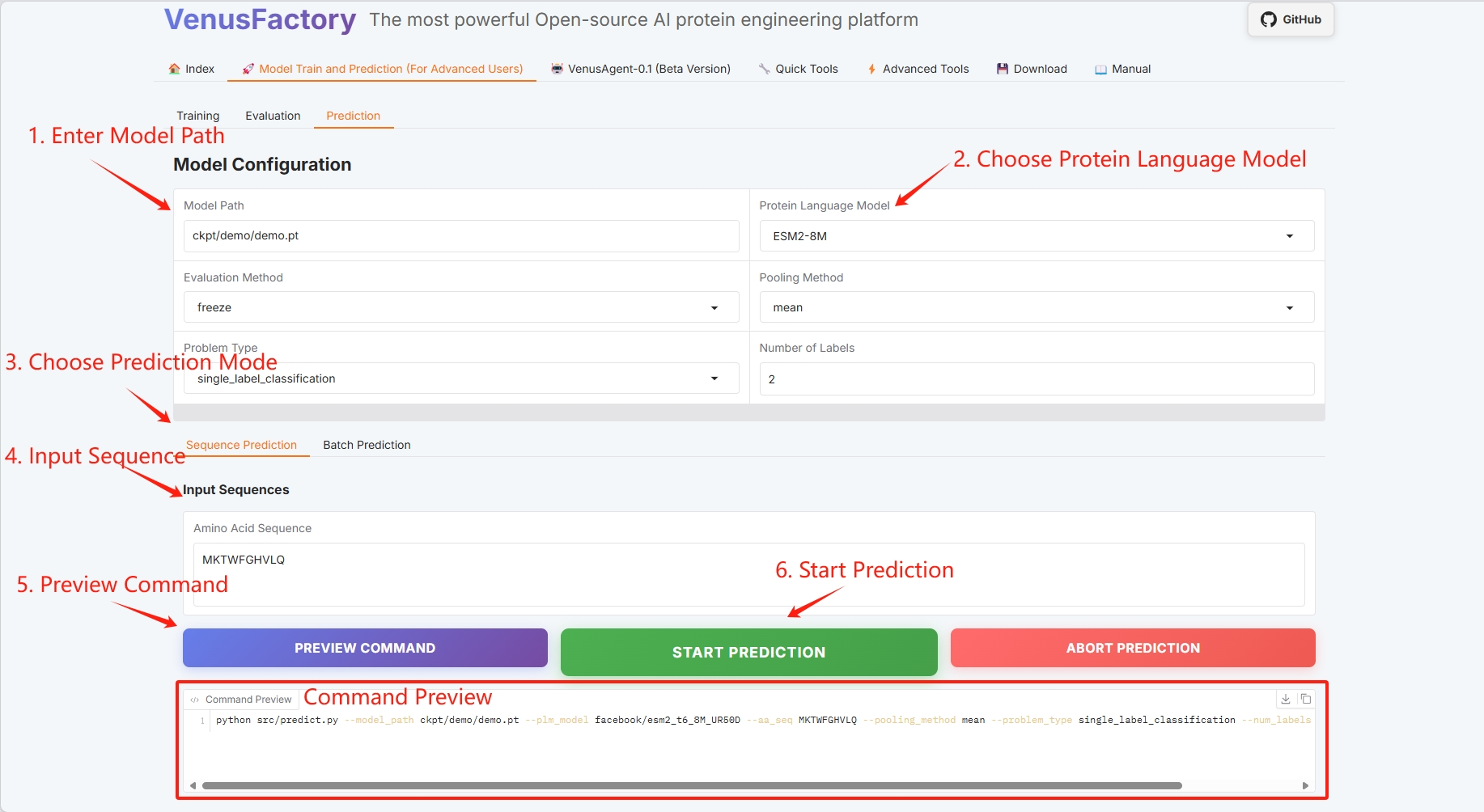
Exemple de séquence protéique : MKTWFGHVLQ
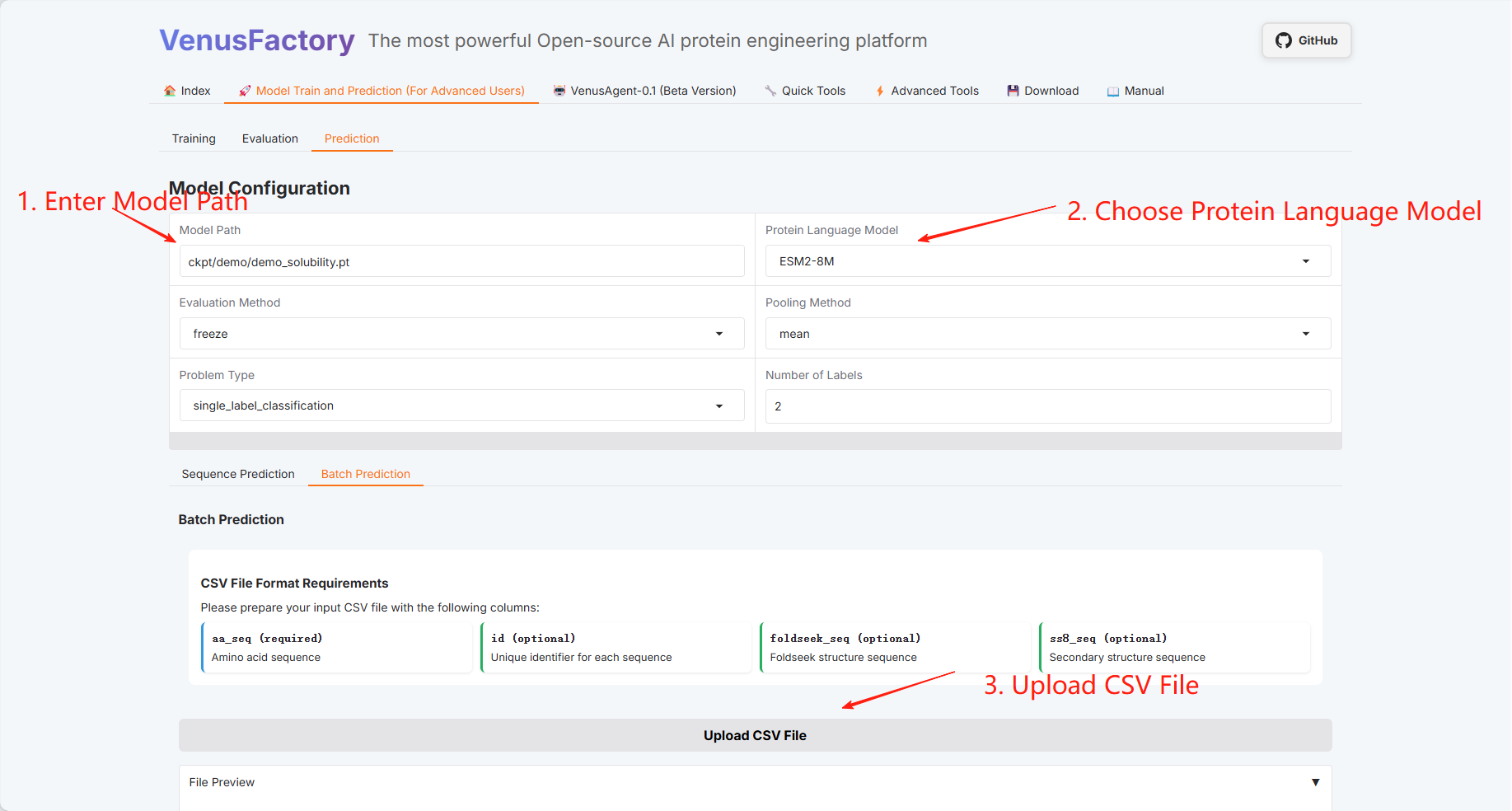
Prédiction par lots
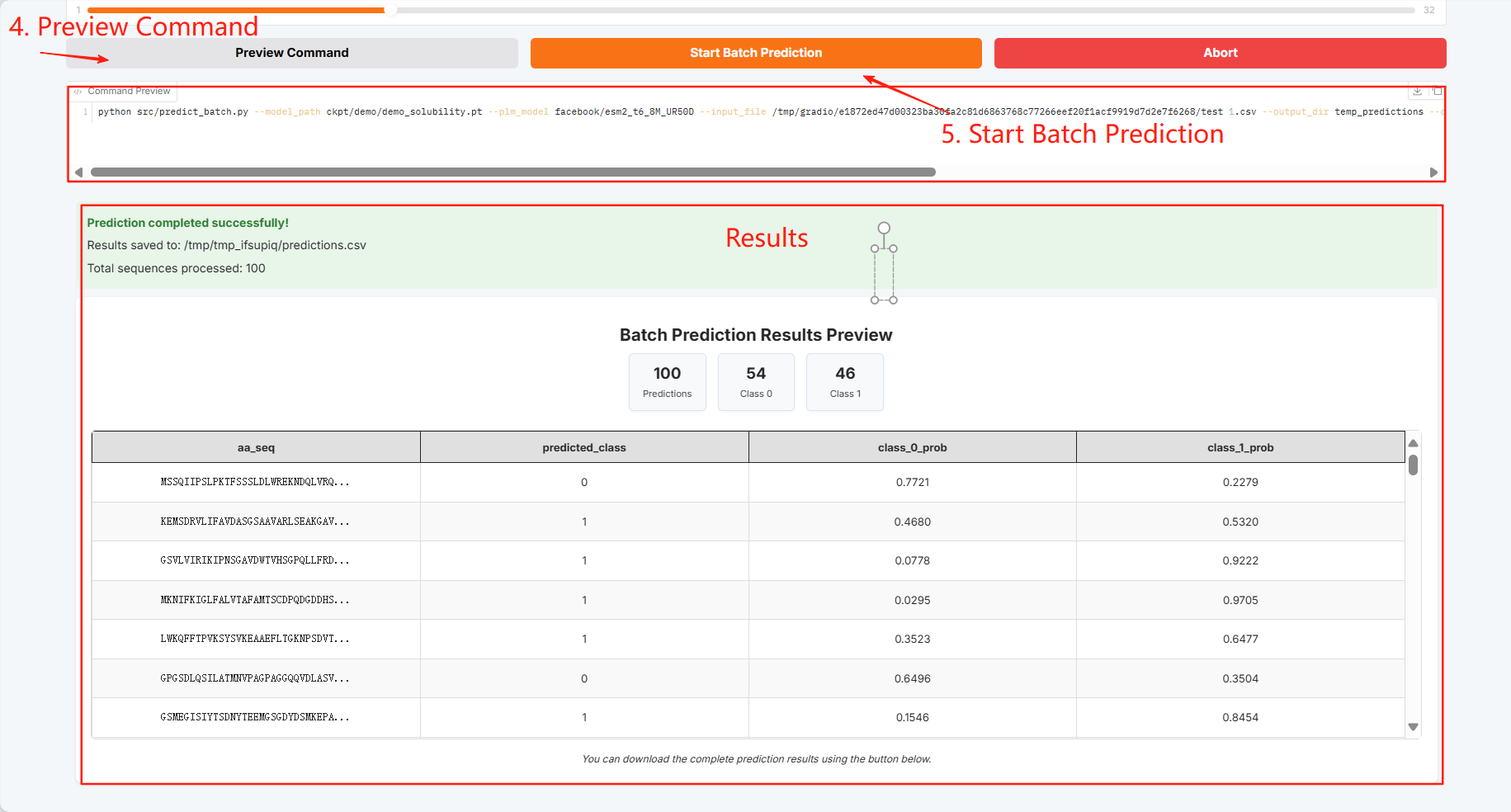
Les résultats de prédiction par lots peuvent être téléchargés et enregistrés
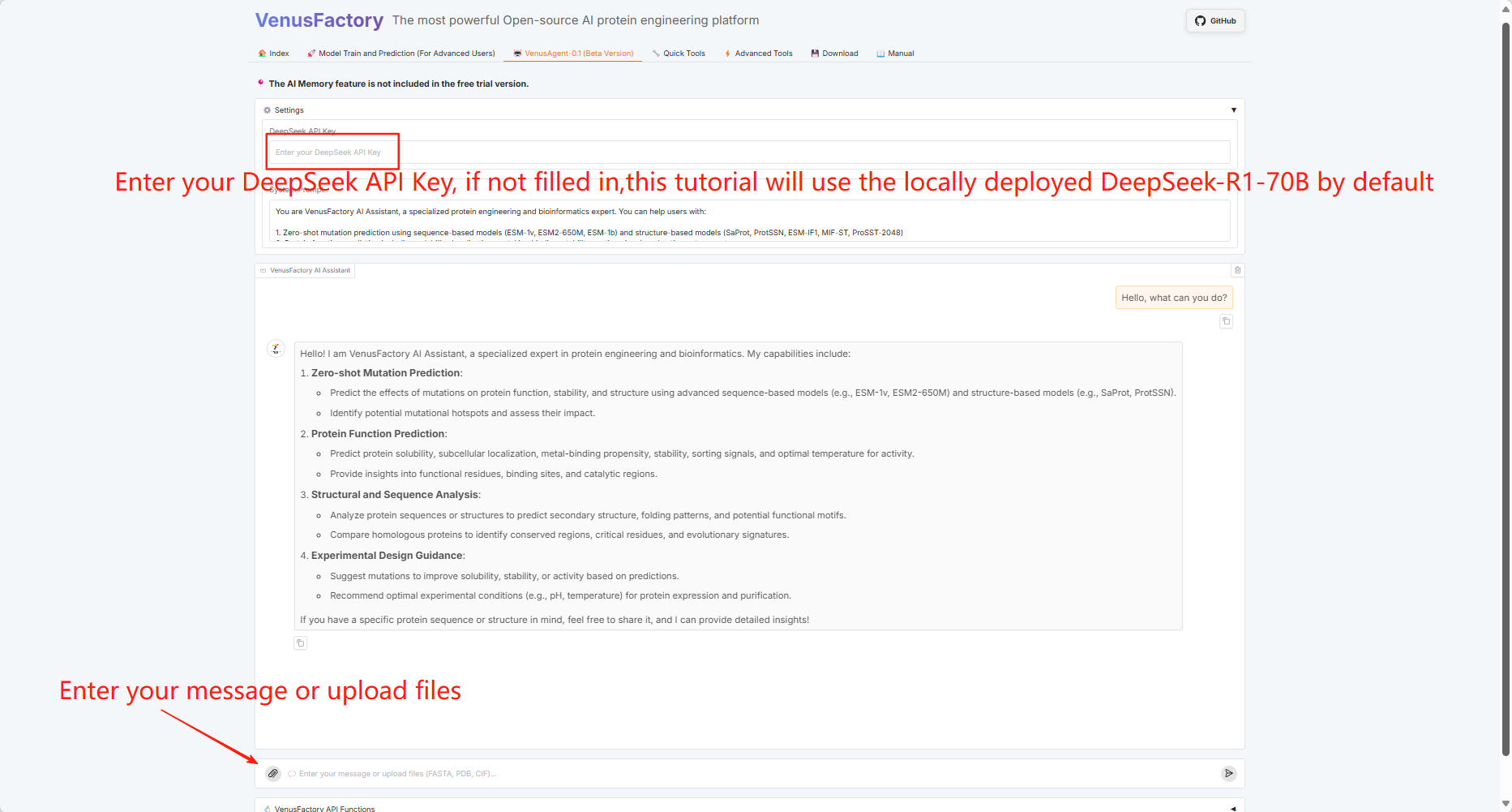
3.4 VenusAgent
Cliquez sur le module « VenusAgent »
Étant donné que VenusAgent doit appeler le grand modèle DeepSeek, ce didacticiel propose deux méthodes d'appel : saisissez vous-même la clé API ou utilisez le modèle DeepSeek-R1-70B déployé sur la plateforme.
Vous pouvez choisir différentes cartes graphiques selon les fonctionnalités requises. Voici les instructions de sélection :
Si vous utilisez une seule carte graphique RTX 4090, la fonction VenusAgent ne prend pas en charge l'utilisation de services de modèles volumineux déployés localement (l'utilisation de la clé API DeepSeek est illimitée).
Si vous utilisez deux cartes graphiques RTX 4090, vous ne pouvez pas utiliser d'autres fonctions immédiatement (après 1 à 2 minutes) après avoir utilisé la fonction VenusAgent (il n'y a aucune restriction lors de l'utilisation de la clé API DeepSeek).
Si vous utilisez deux cartes graphiques RTX A6000, les fonctions de VenusAgent sont illimitées.
Les ressources informatiques utilisées dans ce tutoriel sont une seule carte RTX 4090. Le modèle utilisé dans le tutoriel est enregistré dans
/openbayes/input/input1Toutes les données sont stockées dans le répertoire/openbayes/home/VenusFactoryannuaire.
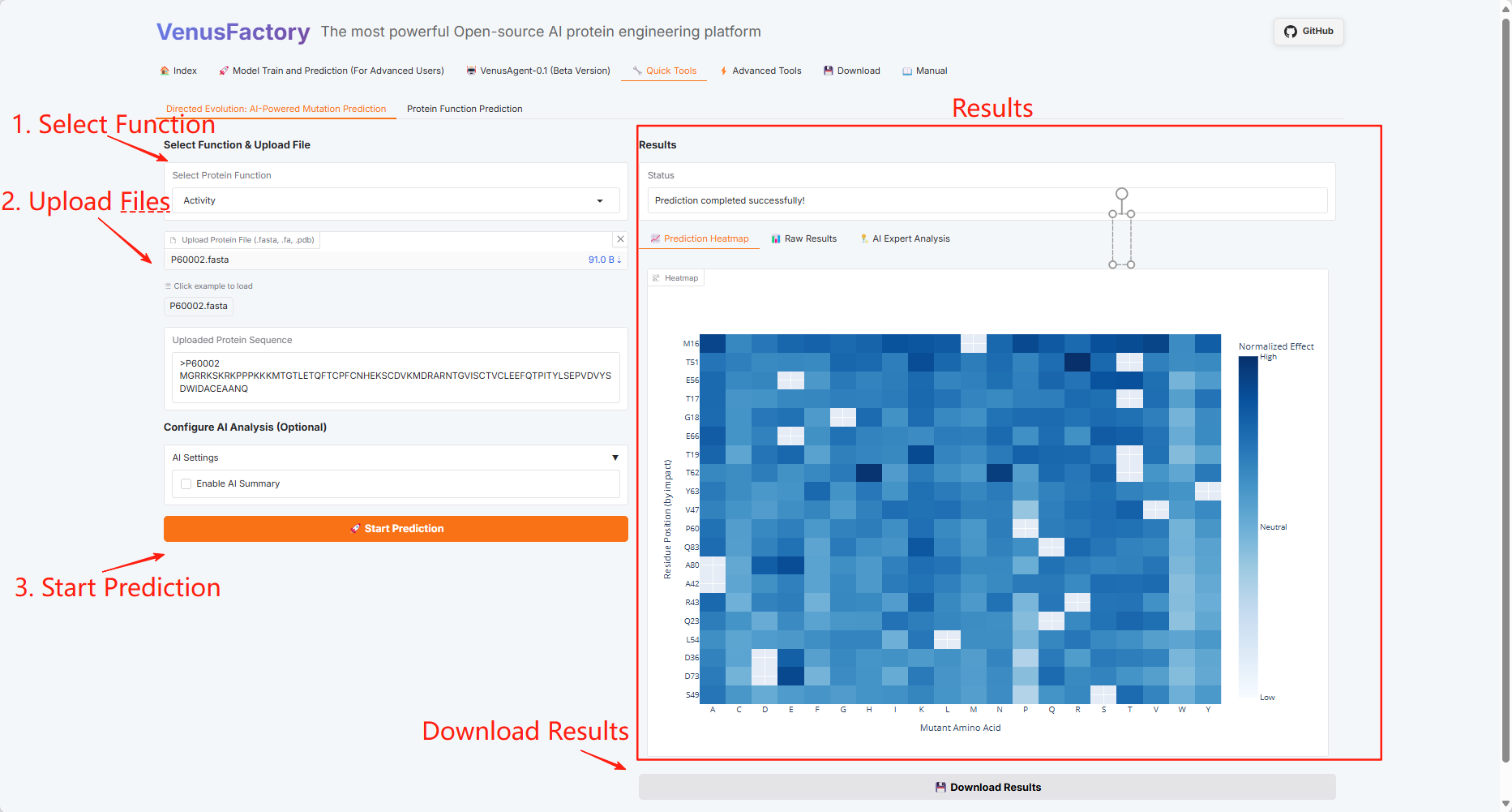
3.5 Outils rapides
Cliquez sur le module « Outils rapides », qui comprend deux fonctions : Évolution dirigée : prédiction de mutation alimentée par l'IA et prédiction de la fonction des protéines.
Évolution dirigée : prédiction des mutations par l'IA
Prédiction de la fonction des protéines
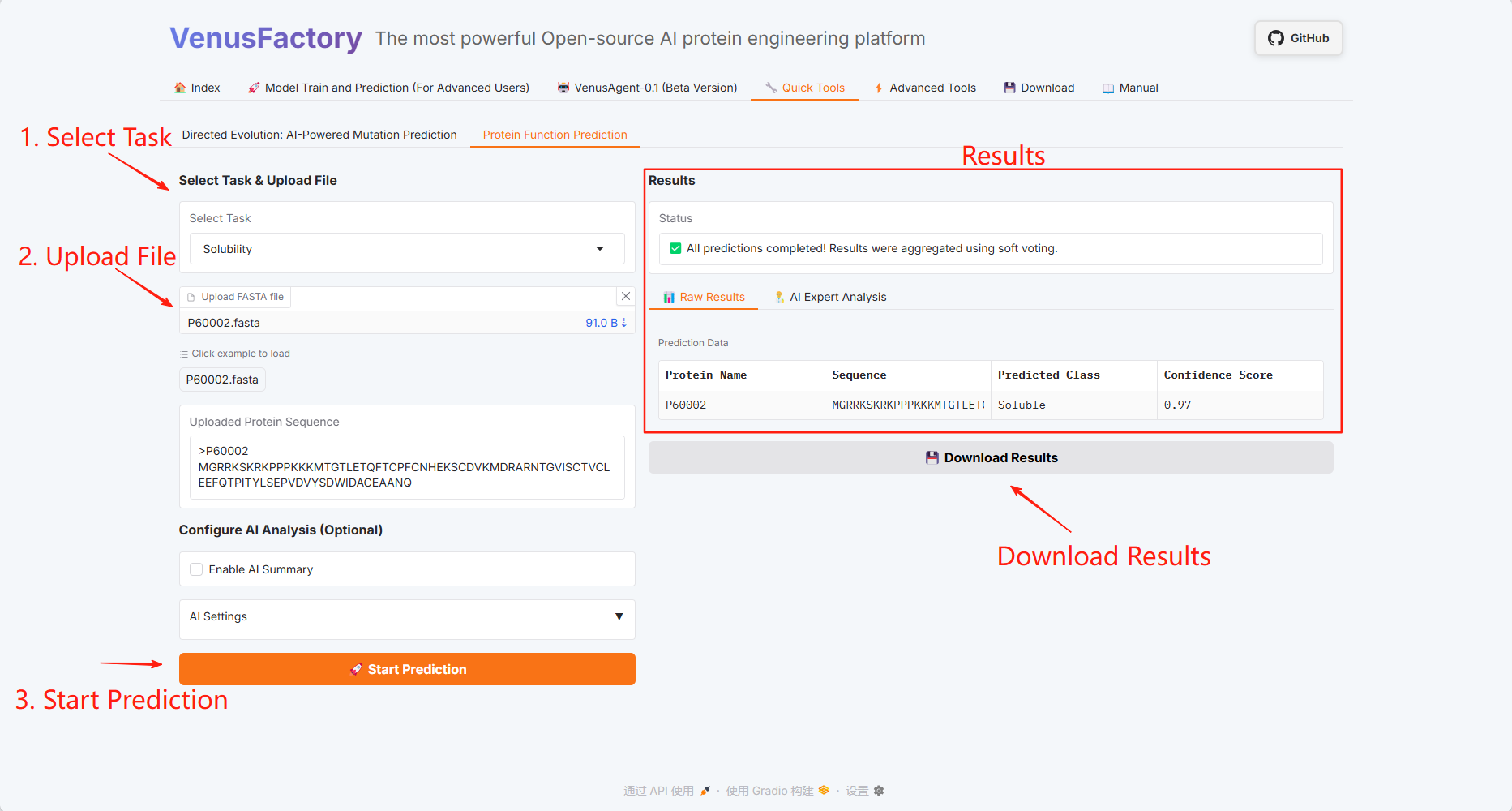
3.6 Outils avancés
Cliquez sur le module « Outils avancés », qui comprend deux fonctions : Évolution dirigée : prédiction de mutation alimentée par l'IA et prédiction de la fonction des protéines.
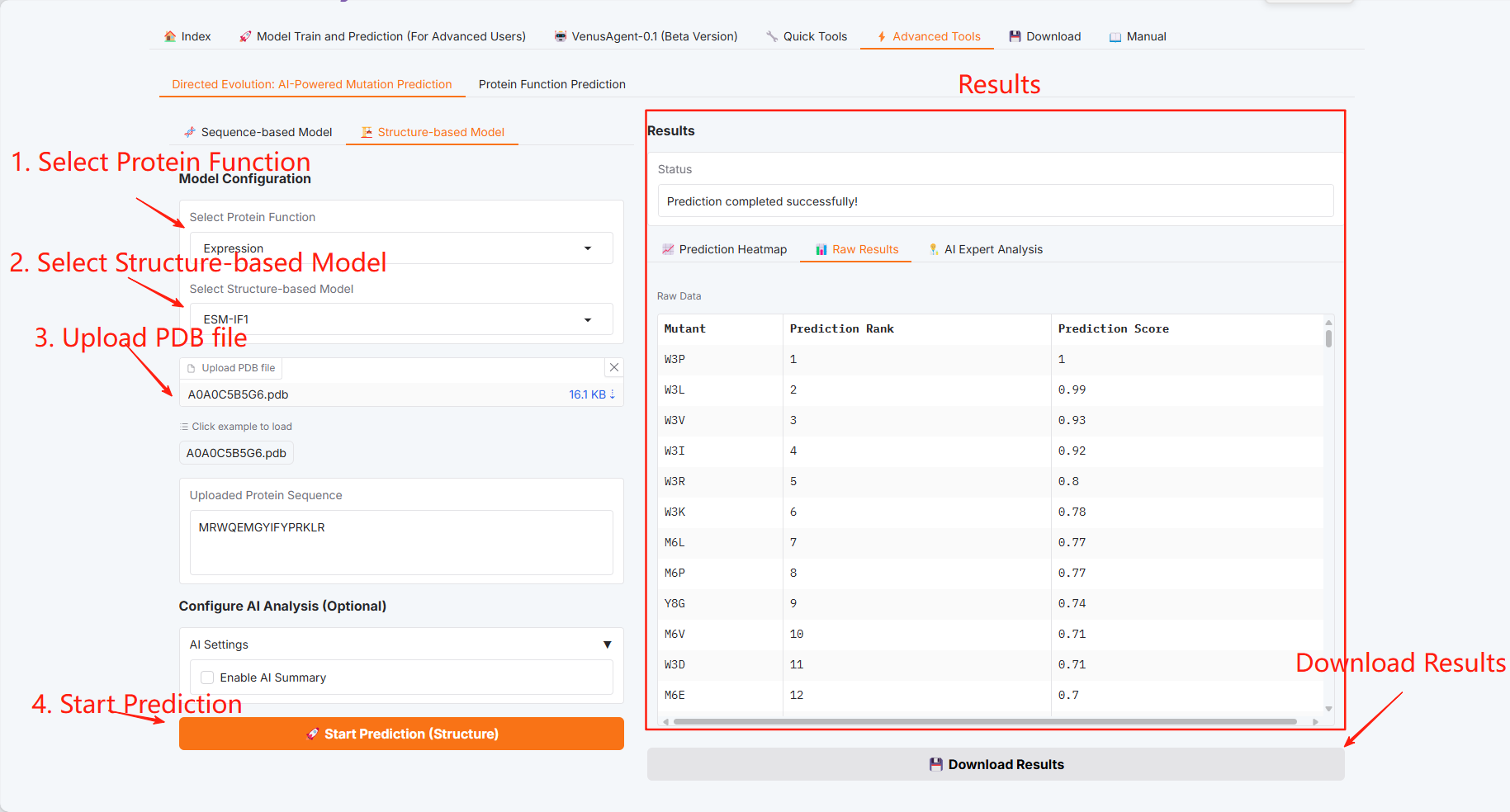
Évolution dirigée : prédiction des mutations par l'IA
Modèle basé sur les séquences
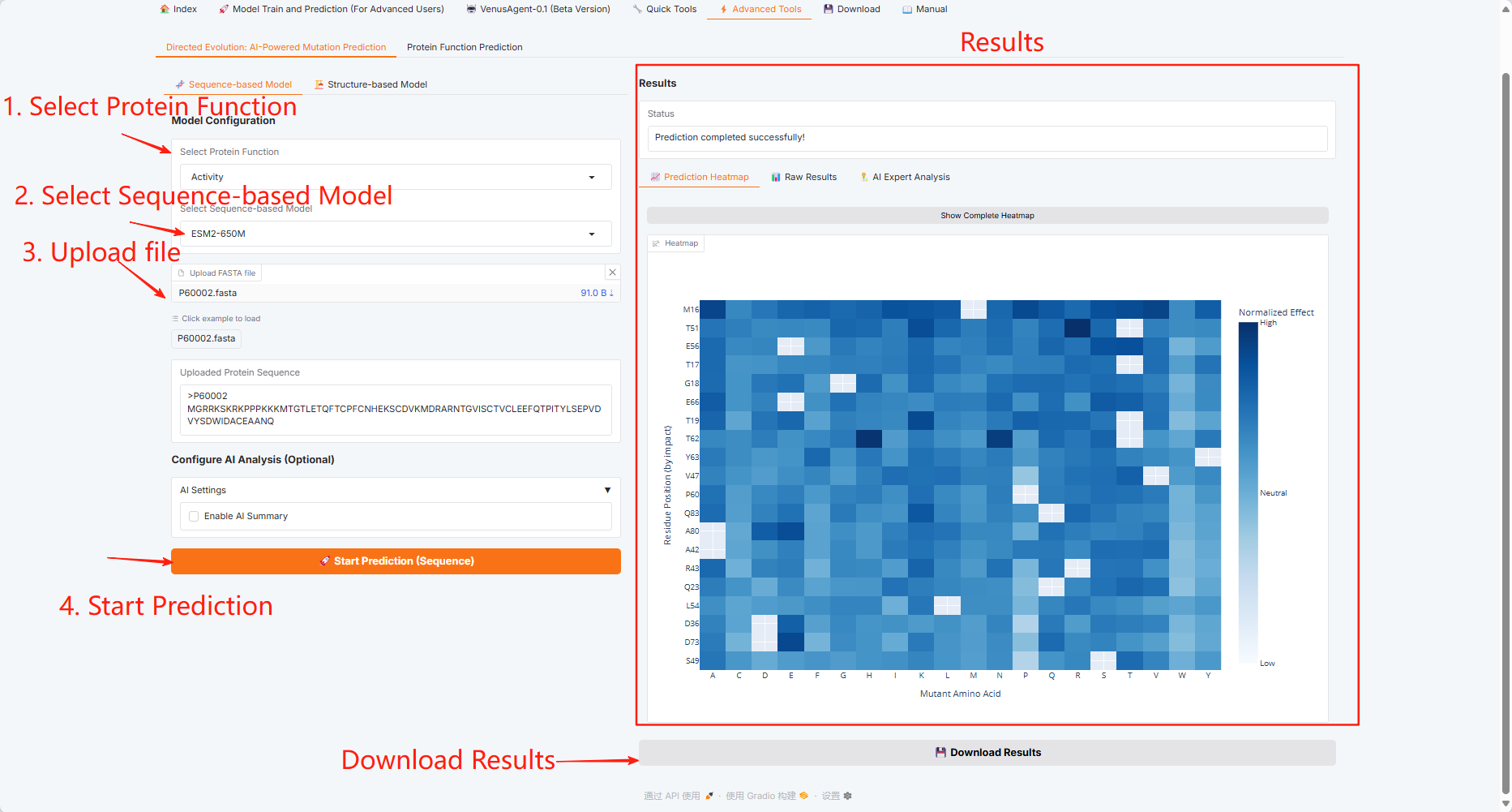
Modèle basé sur la structure
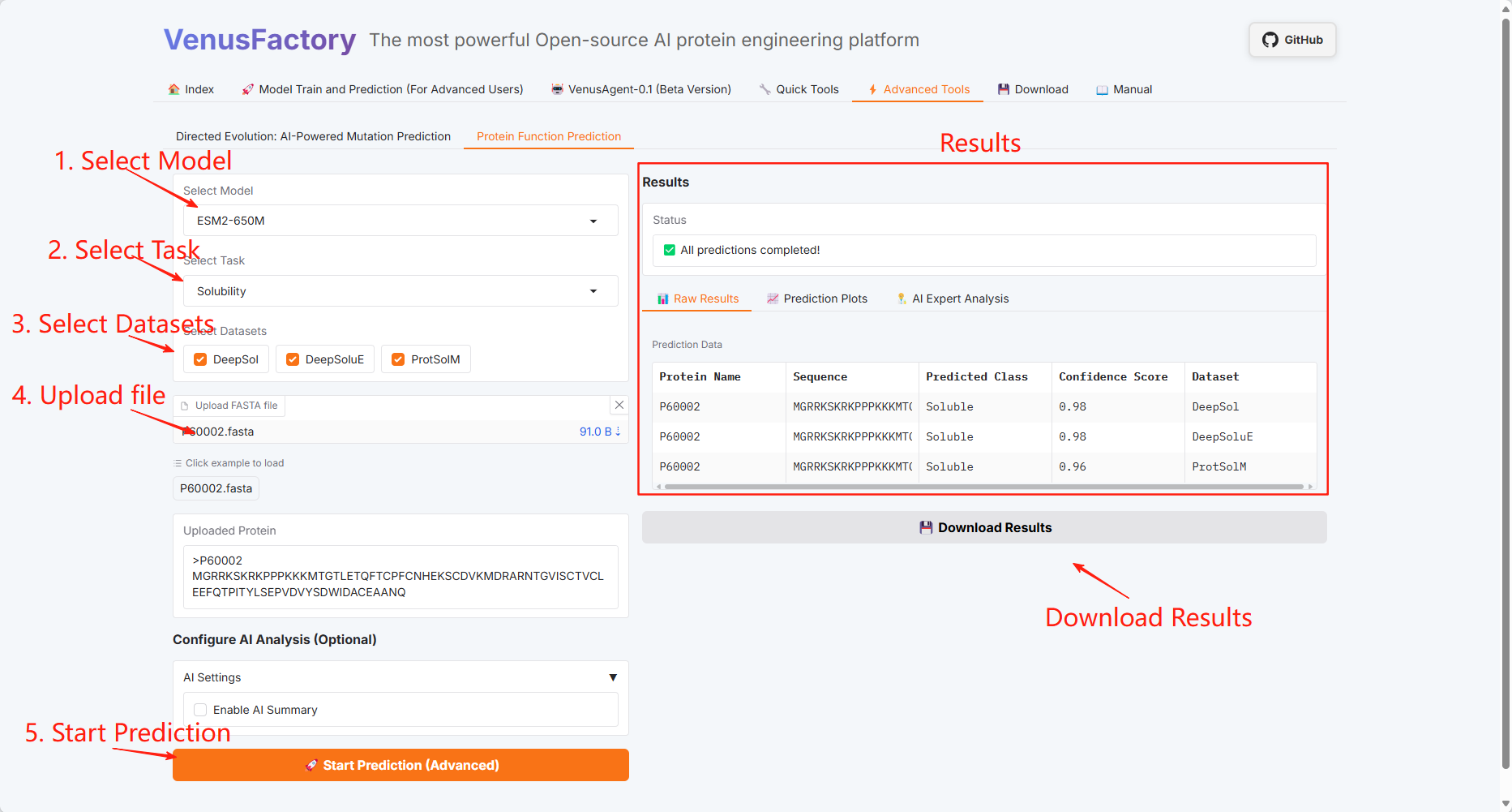
Prédiction de la fonction des protéines
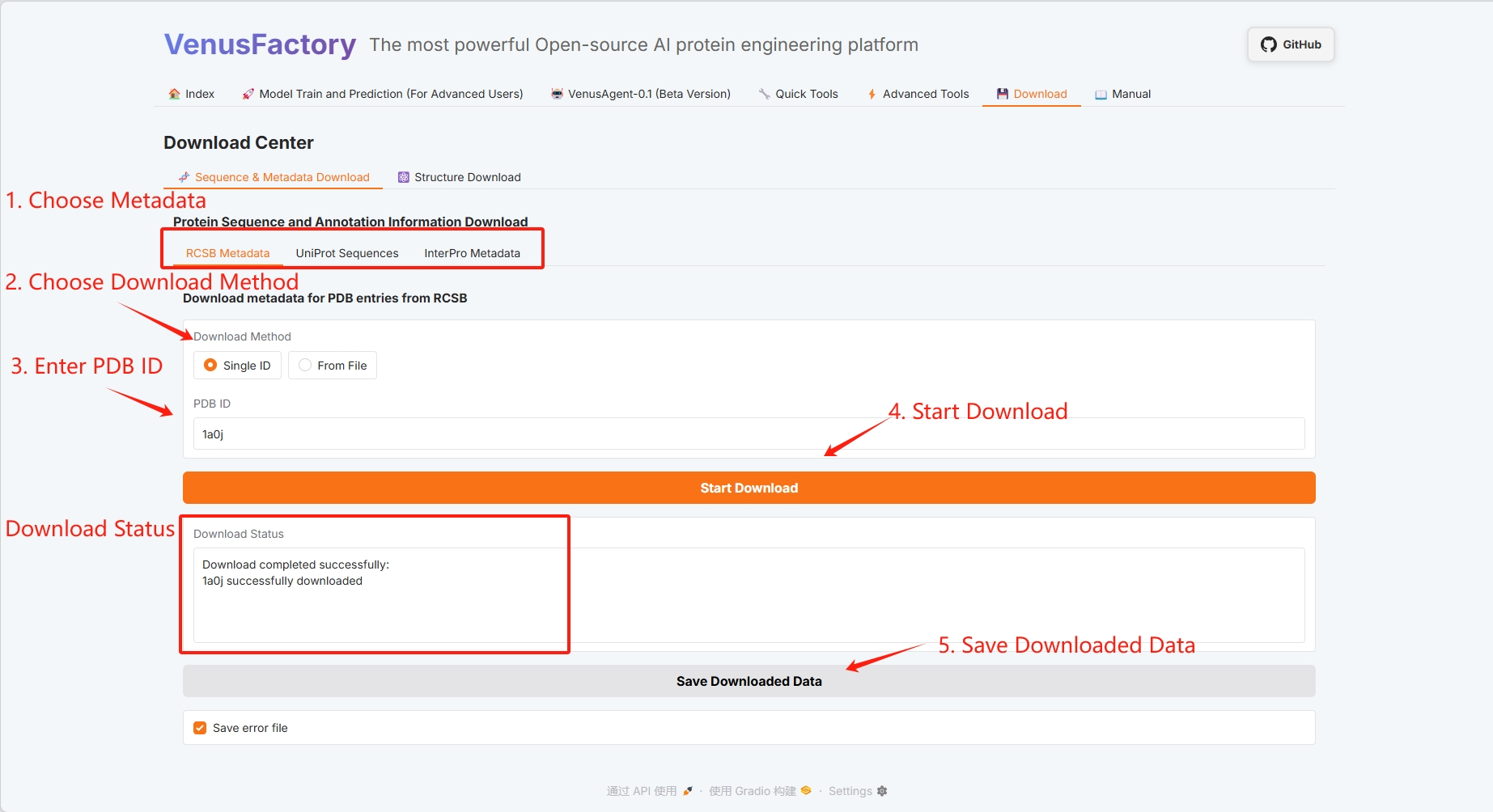
3.7 Télécharger
Cliquez sur le module Télécharger pour télécharger les données sur les protéines dans cette interface.
3. Discussion
🖌️ Si vous voyez un projet de haute qualité, veuillez laisser un message en arrière-plan pour le recommander ! De plus, nous avons également créé un groupe d’échange AI4S. Bienvenue amis pour scanner le code QR et commenter [AI4S] pour rejoindre le groupe pour discuter de divers problèmes techniques et partager les résultats de l'application↓

Informations sur la citation
Les informations de citation pour ce projet sont les suivantes :
@inproceedings{tan-etal-2025-venusfactory,
title = "{V}enus{F}actory: An Integrated System for Protein Engineering with Data Retrieval and Language Model Fine-Tuning",
author = "Tan, Yang and Liu, Chen and Gao, Jingyuan and Wu, Banghao and Li, Mingchen and Wang, Ruilin and Zhang, Lingrong and Yu, Huiqun and Fan, Guisheng and Hong, Liang and Zhou, Bingxin",
editor = "Mishra, Pushkar and Muresan, Smaranda and Yu, Tao",
booktitle = "Proceedings of the 63rd Annual Meeting of the Association for Computational Linguistics (Volume 3: System Demonstrations)",
month = jul,
year = "2025",
address = "Vienna, Austria",
publisher = "Association for Computational Linguistics",
url = "https://aclanthology.org/2025.acl-demo.23/",
doi = "10.18653/v1/2025.acl-demo.23",
pages = "230--241",
ISBN = "979-8-89176-253-4",
}Créer de l'IA avec l'IA
De l'idée au lancement — accélérez votre développement IA avec le co-codage IA gratuit, un environnement prêt à l'emploi et le meilleur prix pour les GPU.