Command Palette
Search for a command to run...
L'Université De Pennsylvanie a Découvert 386 Nouveaux Peptides Antimicrobiens À Partir De Venins d'animaux Et a Développé Un Modèle d'apprentissage Profond, APEX, Pour Sélectionner Des Candidats Antibiotiques potentiels.

La découverte de la pénicilline en 1928 a marqué le début d'une nouvelle ère dans la lutte contre les infections bactériennes. Ces antibiotiques ont considérablement amélioré la capacité de l'humanité à combattre les maladies infectieuses. Cependant, avec leur utilisation généralisée et souvent excessive dans des domaines comme la médecine et l'agriculture, une résistance bactérienne est apparue et s'aggrave à un rythme alarmant.
À l'aube du XXIe siècle, la résistance aux antibiotiques est devenue un enjeu majeur de santé publique mondiale. Selon les données de l'Organisation mondiale de la Santé, environ 5 millions de personnes meurent chaque année dans le monde, directement ou indirectement, d'infections pharmacorésistantes. La principale cause de cette crise est la croissance et la propagation rapides des agents pathogènes résistants aux antibiotiques. Parmi eux, les bactéries à Gram négatif, classées « agents pathogènes prioritaires » par l'OMS, sont particulièrement problématiques. Grâce à leur forte capacité à développer une résistance aux antibiotiques, elles continuent de dominer la « guerre antibactérienne » contre l'homme. Plus grave encore, ces dernières décennies,La recherche et le développement de nouveaux antibiotiques stagnent : les coûts élevés de la R&D et la longueur des cycles d'essais cliniques ont dissuadé de nombreuses sociétés pharmaceutiques de se lancer dans ce domaine. Le nombre de nouveaux antibiotiques a fortement diminué, bien en deçà de l'évolution des bactéries résistantes aux médicaments.
Face à ce dilemme, les scientifiques se sont intéressés à un potentiel « trésor » naturel : le venin animal. Après des millions d'années d'évolution naturelle, le venin animal a engendré une diversité moléculaire extrêmement riche, notamment des peptides et des protéines bioactifs.Il peut non seulement interagir avec une large gamme de cibles biologiques, mais également présenter une activité antibactérienne significative.En fait, l'utilisation du venin en sciences biomédicales est déjà un succès avéré. Le ziconotide (nom commercial Prialt®), extrait du venin de cône, est devenu un analgésique important pour le traitement de la douleur chronique ; le captopril, développé à partir du venin de serpent, est un antihypertenseur couramment utilisé en pratique clinique. Ces exemples apportent des éclairages précieux sur l'exploration du venin dans le domaine antimicrobien.
Dans ce contexte,Une équipe de recherche de l'Université de Pennsylvanie aux États-Unis a examiné à haut débit des peptides candidats antimicrobiens issus de protéines de venin du monde entier et a découvert 386 peptides antimicrobiens dotés de nouvelles structures à partir de venins d'animaux du monde entier.Parmi elles, la molécule 91.4%, vérifiée expérimentalement, a montré une forte activité antibactérienne, offrant une idée révolutionnaire pour la découverte d'une nouvelle génération d'antibiotiques.
Les résultats de recherche pertinents ont été publiés dans Nature Communications sous le titre « Exploration computationnelle des venins mondiaux pour la découverte d'antimicrobiens avec l'intelligence artificielle Venomics ».
Points saillants de la recherche :
* Cette étude a construit une base de données mondiale sur les venins et généré une bibliothèque candidate de peptides cryptés, qui comprenait un total de 16 123 protéines de venin et a généré plus de 40 millions de peptides cryptés de venin ;
* Cette étude a utilisé le modèle d'apprentissage profond APEX pour prédire la concentration inhibitrice minimale de chaque peptide contre 34 espèces bactériennes, en sélectionnant finalement 386 peptides candidats avec un potentiel antibactérien et une faible similitude de séquence avec les AMP connus ;
* L'équipe de recherche a synthétisé et testé 58 peptides à partir de 386 VEP, dont 91,4% (53) avaient des effets inhibiteurs significatifs sur au moins un pathogène.

Adresse du document :
https://www.nature.com/articles/s41467-025-60051-6[*](https://arxiv.org/pdf/2507.09466*)
Suivez le compte officiel et répondez « APEX » pour obtenir le PDF complet
Base de données : Collection de 16 000 protéines de venin
Afin d'identifier de nouveaux candidats antimicrobiens, l'étude a collecté des séquences de protéines de venin à partir de quatre bases de données : ConoServer, ArachnoServer, ISOB et VenomZone, ce qui a donné un total de 16 123 protéines de venin. ConoServer se concentre sur les conopeptides, ArachnoServer inclut les toxines d'araignées, ISOB est une base de données de venins de serpents indigènes du Bangladesh, et VenomZone, compilée à partir d'UniProtKB, couvre les protéines de six taxons : serpents, scorpions, araignées, escargots cônes, anémones de mer et insectes, représentés dans cette étude par UniProt.
Chacun de ces ensembles de données possède des caractéristiques et des utilisations uniques. Comme le montre la figure ci-dessous, une analyse du chevauchement des espèces entre différentes bases de données révèle qu'UniProt contient le plus grand nombre d'espèces uniques, atteignant 699, ce qui témoigne d'une plus grande diversité protéique. ConoServer et ArachnoServer, en revanche, contiennent des sous-ensembles relativement restreints d'espèces uniques, tandis qu'ISOB n'en contient aucune.L’existence de cette diversité permet aux chercheurs d’explorer les peptides antimicrobiens dans le venin sous de multiples angles, d’intégrer des informations sur les protéines provenant de différentes sources et d’augmenter la possibilité de découvrir de nouveaux peptides antimicrobiens.
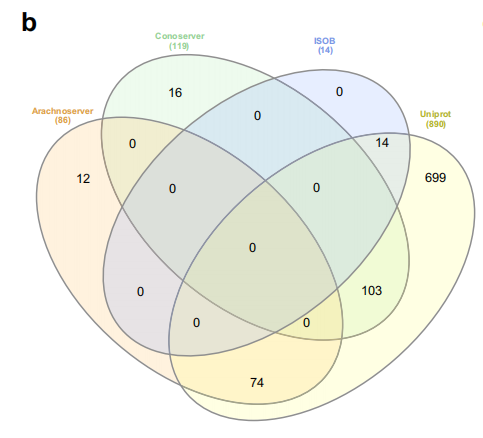
Comme le montre la figure ci-dessous, les chercheurs ont traité ces protéines de venin pour générer plus de 40 millions de peptides cryptés de venin (PEV). Le modèle d'apprentissage profond APEX a ensuite été utilisé pour prédire l'activité antimicrobienne de ces peptides, en estimant la concentration minimale inhibitrice (CMI) de chaque peptide contre les souches bactériennes, la CMI médiane étant utilisée comme mesure de l'activité antimicrobienne. Grâce à des analyses plus poussées, les chercheurs ont finalement identifié 7 379 PEV dont la CMI médiane était ≤ 32 μmol·L⁻¹. Après filtrage pour rechercher la similarité de séquence avec des peptides antimicrobiens connus, 386 candidats ont été identifiés. Ces candidats diffèrent structurellement et fonctionnellement des peptides antimicrobiens connus, offrant des cibles précieuses pour une validation expérimentale ultérieure.
Prédiction du modèle APEX : un processus d'intégration de données protéomiques de venin multi-sources et de criblage de peptides candidats antimicrobiens
Le modèle APEX est un outil d’apprentissage en profondeur qui se concentre sur l’exploitation minière potentielle et la prédiction de peptides antimicrobiens.Son principal avantage réside dans sa capacité à prédire l'activité antimicrobienne spécifique à une souche bactérienne. Après un entraînement systématique, le modèle peut prédire avec précision les concentrations minimales inhibitrices (CMI) des peptides pour 34 souches bactériennes différentes, un paramètre clé pour mesurer l'efficacité des agents antimicrobiens.
L'ensemble de données d'entraînement du modèle se compose de deux parties :Données de séquences peptidiques accumulées en interne et informations publiques sur les peptides antimicrobiens (AMP) obtenues à partir de la base de données DBAASP.Ce support de données diversifié permet à APEX d'effectuer des analyses exceptionnelles de la relation de cartographie complexe entre les séquences peptidiques et l'activité antibactérienne, posant ainsi une base algorithmique solide pour le criblage ultérieur.
En termes de conception architecturale, APEX adopte un cadre de modèle de tâche multi-objectif et une stratégie en « trois étapes » pour cribler les molécules candidates peptidiques antimicrobiennes à partir de protéines de venin.d'abord,La méthode de la fenêtre coulissante a été utilisée pour construire une bibliothèque de peptides, et des fragments d'une longueur allant de 8 à 50 acides aminés ont été sélectionnés.Deuxièmement,Le modèle APEX a été utilisé pour prédire la concentration minimale inhibitrice (CMI) de ces peptides contre différentes souches bactériennes ;enfin,Une sélection plus poussée basée sur la similarité des séquences a permis d'obtenir un groupe de peptides cryptés par le venin (VEP) dotés de nouvelles structures et d'une excellente activité.
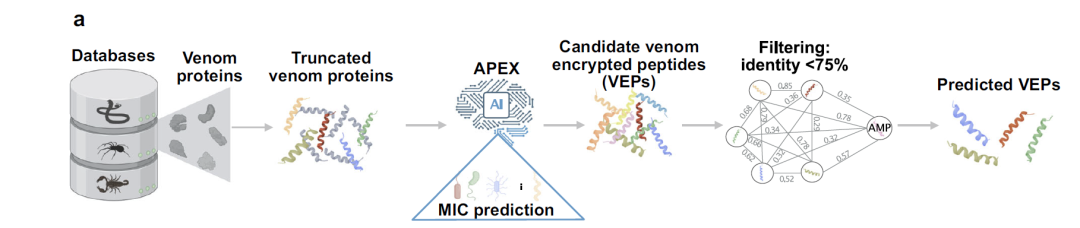
Cette conception permet l'identification précise de molécules candidates présentant une activité antimicrobienne potentielle lors du traitement de quantités massives de peptides cryptés de venin (PEV). Comparé aux méthodes de criblage expérimentales traditionnelles, APEX améliore considérablement l'efficacité de la découverte de peptides antimicrobiens tout en réduisant le recours à des analyses biochimiques gourmandes en ressources, offrant ainsi aux chercheurs une méthode de criblage plus rapide et plus économique.
Dans les applications pratiques, l'utilisation du modèle APEX nécessite une certaine préparation technique. Les chercheurs doivent configurer un environnement d'exécution adapté, notamment l'installation de Python 3.9, d'une version spécifique de PyTorch et d'une série de bibliothèques dépendantes telles que numpy, scipy et matplotlib. Une fois l'environnement configuré, les séquences peptidiques candidates sont enregistrées dans un fichier texte. En exécutant la commande spécifiée, le modèle génère un fichier CSV contenant les résultats de prédiction de la CMI. L'analyse de ces prédictions permet aux chercheurs d'identifier rapidement les peptides à fort potentiel antimicrobien pour une vérification expérimentale plus approfondie.
Vérification de l'activité antibactérienne des VEP et analyse de leur mécanisme d'action
Pour vérifier l’activité antibactérienne des peptides cryptés du venin (VEP), l’équipe de recherche a mené une série d’expériences.Dans le test d’activité antimicrobienne, 58 VEP ont été testés contre une variété d’agents pathogènes.Les résultats sont présentés dans la figure ci-dessous, 53 d'entre eux (représentant 91,4%) ont montré une activité contre au moins un pathogène.Tous les VEP de la base de données ArachnoServer ont montré une activité antibactérienne, ce qui a fortement confirmé le fort potentiel antibactérien de ce type de peptide.

Cependant, certains peptides viraux de la base de données UniProt présentaient une efficacité relativement limitée. Par exemple, UniprotKB-2 n'a montré aucune activité antibactérienne, tandis qu'UniprotKB-6 et UniprotKB-11 n'ont eu d'effet inhibiteur que sur Enterococcus faecalis. Des analyses plus poussées ont révélé que ces peptides de faible activité présentaient généralement une faible hydrophobicité et une faible charge nette, ce qui suggère queL'hydrophobicité et la charge nette jouent un rôle clé dans la promotion de l'interaction entre les peptides et les membranes bactériennes, et leurs niveaux peuvent affecter directement l'efficacité antibactérienne des VEP.
Lors de l'étude de la structure secondaire, l'équipe a observé, grâce à l'analyse du dichroïsme circulaire (CD), que les modifications structurelles des VEP dépendent fortement de l'environnement. En solution aqueuse, les VEP présentent principalement une structure désordonnée ; cependant, dans un environnement membranaire bactérien simulé (comme un système micellaire SDS) ou un milieu d'induction hélicoïdal (comme une solution mixte trifluoroéthanol/eau), ils subissent une transition conformationnelle significative, passant d'une structure désordonnée à une hélice α. Cette caractéristique structurelle est très cohérente avec le comportement des peptides antimicrobiens typiques.Cela suggère que les VEP peuvent être spécialement adaptés aux fonctions liées au biofilm, ce qui fournit également des indices importants sur leur mécanisme antibactérien.
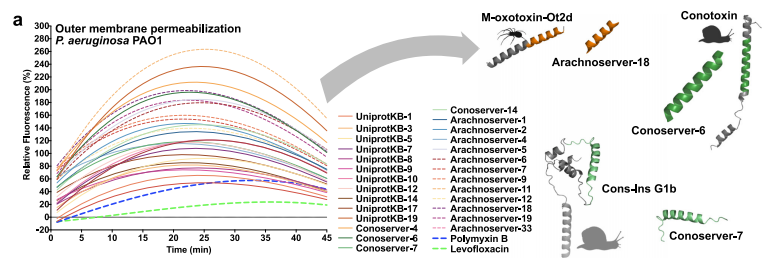
Concernant la recherche sur le mécanisme d'action, l'équipe a évalué les effets des PEV sur les membranes externes bactériennes et les membranes cytoplasmiques par des tests de fluorescence. Les résultats sont présentés dans la figure ci-dessous. 23 PEV peuvent pénétrer efficacement la membrane externe bactérienne, parmi lesquels Arachnoserver-18, ConoServer-6 et ConoServer-7, qui présentent des activités de perméabilité particulièrement remarquables. Lors de l'expérience de dépolarisation de la membrane cytoplasmique, l'effet de dépolarisation de la plupart des PEV testés était supérieur à celui du groupe témoin, mais légèrement inférieur à celui de certaines familles de peptides connues. Globalement,L'effet antibactérien des VEP s'obtient principalement par l'induction d'une dépolarisation de la membrane cytoplasmique. Ce mécanisme est cohérent avec le mode d'action de nombreux peptides antimicrobiens et peptides de cryptage connus, ce qui renforce son potentiel en tant que nouvel agent antibactérien.
Le monde universitaire et l'industrie collaborent pour explorer et développer de nouveaux médicaments antimicrobiens à faible résistance
Afin de développer de nouveaux médicaments antimicrobiens moins susceptibles d’induire une résistance aux médicaments, l’exploration collaborative entre le monde universitaire et le monde des affaires, de la recherche fondamentale à la transformation industrielle, libère constamment le potentiel d’application des peptides antimicrobiens du venin.
Dans le monde universitaire, des équipes de recherche de divers pays ont mené des recherches approfondies sur la découverte, la conception et l'analyse des mécanismes des peptides antimicrobiens issus du venin. L'équipe Mu Yuguang de l'Université technologique de Nanyang à Singapour s'est penchée sur le problème des limites des modèles traditionnels en matière de prédiction des interactions protéiques, liées à la quantité de données et aux dimensions des caractéristiques.Construit le plus grand ensemble de données d'interactions protéiques basé sur la structure à ce jour,Ils ont également développé le framework d'apprentissage profond ProAffinity-GNN. Ce modèle intègre de manière innovante des modèles de langage protéique à des réseaux de neurones graphes, couplant ainsi étroitement les informations de séquence aux caractéristiques structurelles spatiales. Non seulement il surpasse les méthodes existantes en termes de précision de prédiction d'affinité protéine-protéine, mais ses puissantes capacités de généralisation fournissent également un paradigme technique pour la prédiction efficace des interactions entre les peptides de venin et les cibles bactériennes. De plus, l'analyse du mécanisme d'action apporte un soutien théorique à la conception optimisée de peptides antimicrobiens.
L'équipe du Qilu Medical College de l'Université du Shandong a utilisé le modèle de diffusion dans le domaine de l'intelligence artificielle pour simuler le processus de « criblage dirigé par perturbation aléatoire » de l'évolution des peptides dans la nature.40 nouvelles séquences peptidiques ont été générées avec succès, dont 25 ont montré une activité antibactérienne ou antifongique claire.
Dans le monde des affaires, l'entreprise israélienne Bountica se concentre sur les problèmes de conservation des aliments et utilise la technologie de fermentation microbienne pour produire en masse des peptides antifongiques dérivés du venin. Sa gamme de produits VenomShieldUne seule partie par million suffit pour inhiber la croissance des moisissures et des levures dans le pain et les jus, prolongeant ainsi la durée de conservation à plus de deux fois celle des conservateurs traditionnels.
China Shanghai Hi-Tech Bioengineering Co., Ltd. coopère avec l'Université Fudan,Ensemble, nous avons construit trois bases de données de protéines antimicrobiennes relativement complètes dans le monde.Ces bases de données fournissent non seulement des outils efficaces pour la recherche et le développement de nouvelles enzymes antimicrobiennes biologiques et de peptides antimicrobiens, mais accélèrent également la recherche de nouvelles substances contre les micro-organismes résistants aux médicaments grâce à l'exploration systématique des données.
Cela démontre l'immense dynamisme de la recherche et de la traduction des peptides antimicrobiens issus du venin, tant en sciences fondamentales qu'en applications industrielles. Alors que les modèles d'apprentissage profond analysent de plus en plus précisément les relations séquence-fonction et que les techniques de biologie synthétique optimisent en permanence la stabilité et l'efficacité de l'expression des peptides, ces molécules, inspirées par la « sagesse de survie » de la nature, sont en passe de devenir un élément clé de la lutte contre la résistance aux antibiotiques et d'offrir de nouvelles garanties pour la santé publique mondiale.
Articles de référence :
1. https://mp.weixin.qq.com/s/lwYWWIe9-Az22jlhiZ747A
2. https://mp.weixin.qq.com/s/sGHRf-ebaSRiqGiUxMMueA
3. https://mp.weixin.qq.com/s/0mJltGuaKTUwYDGK4wD9UA
4. https://mp.weixin.qq.com/s/R6Y_-38saZwqSSow0LNhzw








