Command Palette
Search for a command to run...
Sur La Base De 8 Millions De Données Réelles, l'équipe De l'Université Cornell a Utilisé Des Réseaux Neuronaux Graphiques Pour Prédire Avec Précision La Survie Des Patients Atteints De Cancer Du Poumon Et a Découvert 3 sous-types mortels.
Il y a dix ans, les résultats de l’essai CheckMate 017 ont choqué la communauté oncologique. Le New England Journal of Medicine, le Journal of the American Medical Association et d'autres revues ont rapporté à plusieurs reprises que les données de survie des patients atteints d'un cancer du poumon épidermoïde avancé traités avec l'inhibiteur de PD-1 Nivolumab étaient significativement améliorées : la durée médiane de survie globale est passée de 6 mois avec la chimiothérapie à 9,2 mois, et le taux de survie à 18 mois était deux fois supérieur à celui du groupe de chimiothérapie. Cette étude marque le début de l’ère des inhibiteurs de points de contrôle immunitaires (ICI), mais elle expose également le problème selon lequel les patients atteints d’un cancer du poumon non à petites cellules avancé (aNSCLC) ont des réponses différentes à l’immunothérapie :Au cours de l'essai, les tumeurs de certains patients ont continué à se résorber pendant plus de 3 ans, tandis que d'autres ont connu une progression de la maladie en quelques mois. Cette hétérogénéité dans la réponse au traitement est devenue un problème à l’ère de la médecine de précision.
La complexité du cancer du poumon provient de sa grande hétérogénéité. Le cancer du poumon non à petites cellules (CPNPC) représente 80% à 85% des cancers du poumon.Environ 751 patients atteints de TP3T sont diagnostiqués à un stade avancé et le taux de survie à 5 ans n'est que de 26,41 TP3T.L’expression différentielle des biomarqueurs du microenvironnement tumoral, les différents états fonctionnels des cellules immunitaires et les diverses comorbidités des patients rendent la situation pathologique compliquée. Les patients recevant un traitement ICI peuvent en bénéficier en raison d'une expression élevée de PD-L1, mais peuvent également connaître une faible efficacité en raison d'une faible charge de mutation tumorale, et les comorbidités peuvent également affecter les options de traitement et le pronostic.
Pour relever ces défis, les plans de diagnostic et de traitement passent d’une approche « universelle » à une « stratification précise ». Dans ce processus de transformation, la médecine prédictive a progressivement émergé. Son objectif principal est d’intégrer des données multidimensionnelles, notamment des dossiers médicaux électroniques et des informations omiques, afin d’adapter le plan de traitement le plus approprié à chaque patient. Ces dernières années, avec l’accumulation continue de données biomédicales à grande échelle et le développement rapide de la technologie d’apprentissage automatique, les chercheurs ont commencé à essayer d’utiliser des méthodes d’apprentissage automatique non supervisées pour regrouper des groupes de patients présentant des caractéristiques similaires afin de prédire les réponses au traitement. Malheureusement, les méthodes traditionnelles présentent souvent des limites dans leurs applications pratiques.Il est difficile d’assurer la cohérence des résultats de survie parmi les patients au sein du groupe, ce qui limite la valeur d’application des résultats stratifiés dans la pratique clinique.
Pour résoudre les problèmes ci-dessus, l'Université Cornell et Regeneron Pharmaceuticals ont proposé le modèle de survie mixte codé par graphique (GEMS).Les relations complexes dans les dossiers médicaux électroniques des patients ont été codées via des réseaux neuronaux graphiques et combinées à des modèles d’analyse de survie pour identifier les sous-phénotypes avec des caractéristiques et des résultats de survie cohérents.L’étude a révélé qu’elle est supérieure aux méthodes traditionnelles pour prédire la survie globale (SG), en identifiant trois sous-phénotypes avec des caractéristiques cliniques et des modèles de survie différents, ouvrant une nouvelle voie à la médecine de précision pour le cancer du poumon.
Les résultats de recherche pertinents ont été publiés dans Nature Communication sous le titre « Identification de sous-phénotypes prédictifs pour les résultats cliniques à l'aide de données du monde réel et d'apprentissage automatique ».

Adresse du document :
https://doi.org/10.1038/s41467-025-59092-8
Le projet open source « awesome-ai4s » rassemble plus de 100 interprétations d'articles AI4S et fournit également des ensembles de données et des outils massifs :
https://github.com/hyperai/awesome-ai4s
Construction d'une cohorte de patients atteints d'un cancer du poumon non à petites cellules avancé à partir de l'ensemble de données réelles à grande échelle de ConcertAI
L'étude a utilisé l'ensemble de données ConcertAI Patient360™ NSCLC de la base de données US Oncology Electronic Health Record (EHR) pour construire une cohorte de patients atteints d'un cancer du poumon non à petites cellules avancé (aNSCLC) recevant un traitement de première intention (1 L) par inhibiteur de point de contrôle immunitaire (ICI).Cet ensemble de données est un ensemble de données anonymisées au niveau des patients basé aux États-Unis, extrait du réseau ConcertAI, couvrant plus de 8 millions de patients uniques.Des données provenant de plus de 900 cliniques d'oncologie et d'hématologie du cancer, représentant des patients traités dans des pratiques communautaires et universitaires dans les 50 États, ont été extraites, y compris des données sur la date et le type de récidive de la maladie, l'histologie, les informations sur le test PD-L1, la réponse tumorale, l'ECOG-PS et les comorbidités.
Comme le montre la figure ci-dessous, cette étude a sélectionné des patients atteints d’un cancer du poumon non à petites cellules (CPNPC) confirmé histologiquement de janvier 2015 à janvier 2023 (n = 17 265) pour construire une cohorte observationnelle rétrospective. Après les critères d'inclusion/exclusion et l'exclusion des patients sans dossiers de survie globale (SG) valides,4 666 patients ont été inclus dans l’étude, et les patients étaient représentés par un vecteur à 104 dimensions, avec des dimensions comprenant des informations démographiques, des tests de laboratoire et d’autres variables.
Sur la base des régions géographiques des établissements cliniques définies par le Bureau du recensement des États-Unis, les chercheurs ont divisé la cohorte en un modèle de développement (régions du Nord-Est, du Sud et de l'Ouest, n = 3 225) et une sous-cohorte de validation (région du Midwest, n = 1 441), qui avaient des caractéristiques démographiques similaires, la sous-cohorte de validation ayant une proportion plus élevée de patients blancs et de patients provenant d'institutions médicales communautaires. La période d’observation de l’étude était de 180 jours avant la date de l’indice. La survie globale (SG) a été définie comme le temps écoulé entre la date d'index et le décès, quelle qu'en soit la cause, et la survie sans progression (SSP) a été définie comme le temps écoulé entre la date d'index et le premier événement de progression dans le monde réel ou le décès, quelle qu'en soit la cause. L’objectif de l’étude est de résoudre des problèmes tels que la prédiction de la survie des patients atteints d’un cancer du poumon non à petites cellules avancé grâce à une analyse pertinente de cet ensemble de données.
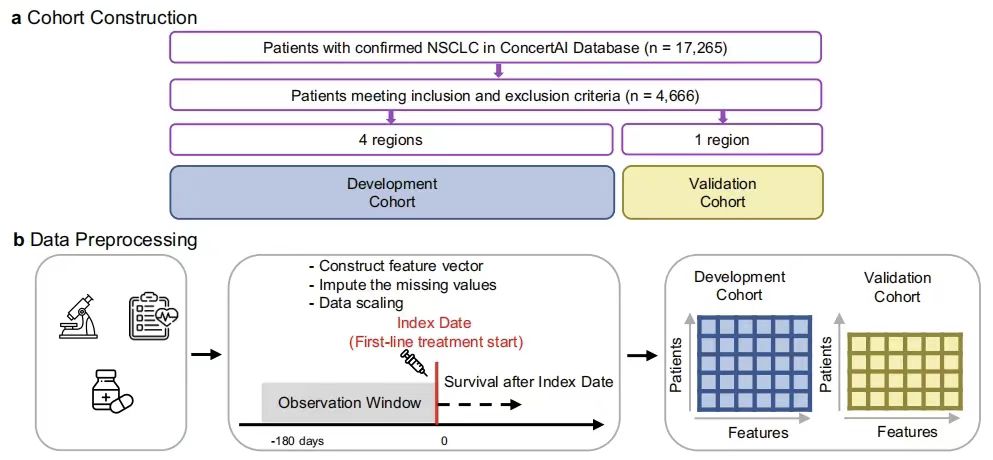
Construction du modèle GEMS : identification basée sur le GNN des sous-phénotypes de survie et validation des performances de prédiction pour le cancer du poumon non à petites cellules avancé
Dans cette étude, le modèle GEMS a été conçu pour identifier les sous-phénotypes prédictifs associés aux caractéristiques de survie globale (SG) dans le monde réel chez les patients atteints d'un cancer du poumon non à petites cellules avancé (aNSCLC).Son architecture principale comprend l'encodeur GNN, le module Cluster et le prédicteur de survie de mélange.
Parmi eux, l'encodeur GNN extrait efficacement les représentations de patients d'ordre élevé en capturant la relation de structure graphique du vecteur de caractéristiques à 104 dimensions du patient (couvrant des variables telles que les données démographiques, les tests de laboratoire et l'état des métastases) ; les représentations codées sont entrées dans le module de clustering pour générer des sous-phénotypes avec une valeur de prédiction de survie comme composants de base du modèle hybride.
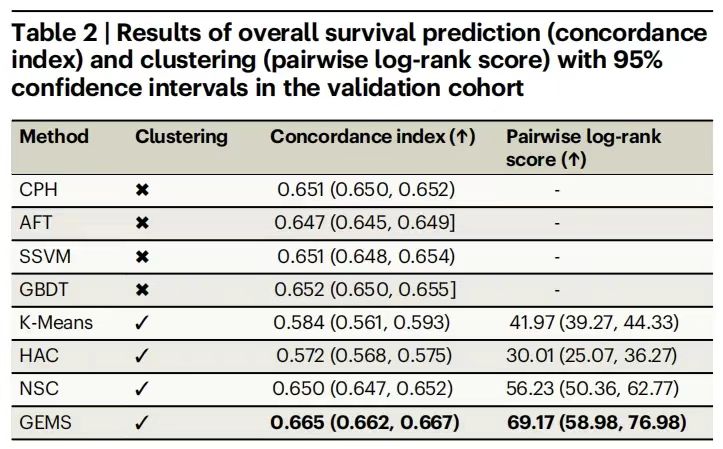
La formation du modèle a d'abord utilisé la cohorte de développement (n = 3 225) comme support de données, et a utilisé l'indice de cohérence (c-index) et le score log-rank par paires comme indicateurs d'évaluation, et les a comparés aux modèles de base traditionnels tels que la régression à risque proportionnel de Cox (CPH), l'arbre de décision à gradient boosté (GBDT), le clustering de survie neuronale (NSC) et les méthodes non supervisées telles que K-means et le clustering hiérarchique.
Les résultats expérimentaux sont présentés dans le tableau suivant.Le GEMS a obtenu de bons résultats dans la prédiction de la survie globale.L'indice c moyen a atteint 0,665 (IC 95% : 0,662-0,667), significativement plus élevé que le 0,652 du meilleur modèle de base GBDT ; le score log-rank était de 69,17 (95% IC : 58,98-76,98), dépassant de loin les 56,23 du NSC, vérifiant l'utilisation efficace des fonctionnalités des données par le cadre d'apprentissage supervisé.
Alors,Cette étude a en outre caractérisé l’impact de l’encodeur GNN sur GEMS en visualisant les représentations dérivées des patients et de leurs encodeurs GNN.L'approximation et la projection uniformes des variétés (UMAP) sont utilisées. Comme le montre la figure ci-dessous, grâce à la visualisation par projection d'approximation de collecteur uniforme (UMAP), on constate que dans l'espace de représentation des patients généré par l'encodeur GNN, les groupes de patients ayant des temps de survie totaux différents sont clairement séparés, tandis que divers types de patients sont répartis de manière mixte dans l'espace des caractéristiques d'origine, ce qui reflète intuitivement la capacité du réseau neuronal graphique à modéliser des relations de caractéristiques complexes.
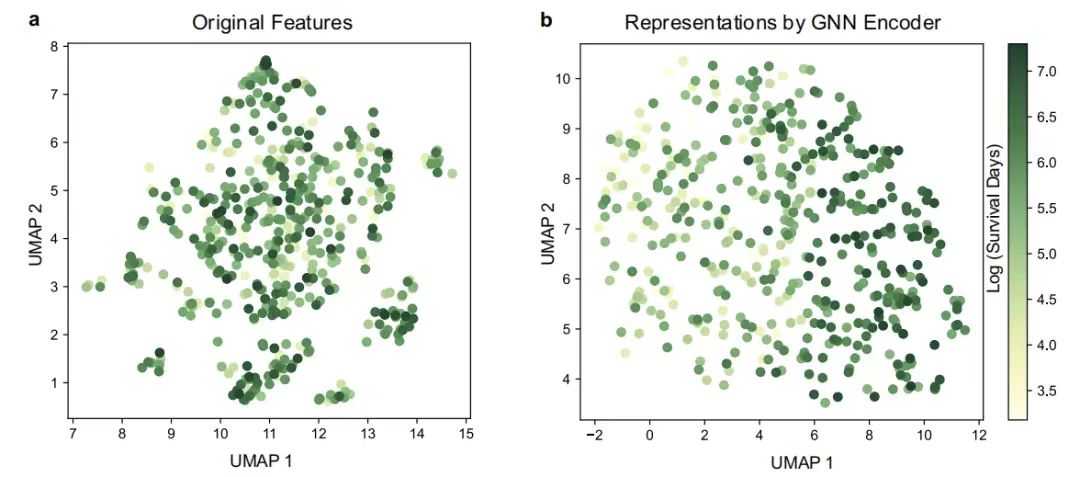
Visualisation UMAP des patients
Figure a : Visualisation UMAP des fonctionnalités d'origine ; Figure b : Visualisation UMAP des caractéristiques obtenues par l'encodeur GNN
Comme le montre la figure ci-dessous,Les chercheurs ont également utilisé le modèle pour identifier trois sous-phénotypes prédictifs présentant des différences de survie significatives :Le sous-phénotype 1 (n = 1335) était caractérisé par une proportion élevée de femmes (55,50%), des comorbidités légères et une faible charge métastatique, avec une survie globale moyenne de 688 jours et les taux d'utilisation les plus faibles d'antitussifs, de bêtabloquants et l'incidence de métastases osseuses/cérébrales/surrénaliennes. La courbe de survie du sous-phénotype 2 (n = 420) a montré une augmentation du risque à moyen terme, avec des comorbidités intermédiaires et une charge métastatique. Le sous-phénotype 3 (n = 1420) avait une proportion féminine de 35,21% et une survie globale moyenne de seulement 321 jours, caractérisée par de multiples médicaments, un taux métastatique élevé (métastases hépatiques 31,20%, métastases osseuses 51,48%) et de graves comorbidités (troubles de l'eau et des électrolytes 8,31%, anomalies rénales 21,43%), et le schéma de cooccurrence le plus complexe de métastases-comorbidités-anomalies de laboratoire.
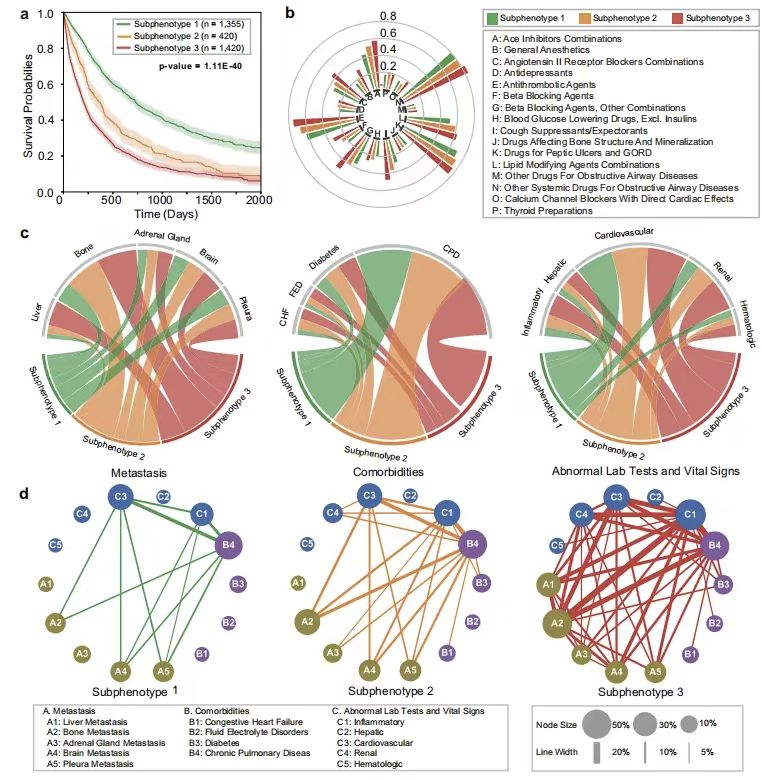
* Figure a : Courbes de Kaplan-Meier de survie globale pour chaque sous-phénotype
* Figure b : Diagramme en forme de rayon de soleil du taux d'administration du médicament de chaque sous-type
* Figure c : Diagramme d'accords des différences dans la classification des métastases (à gauche), des comorbidités (au milieu) et des caractéristiques cliniques anormales
* Figure d : L'incidence des différents sous-phénotypes
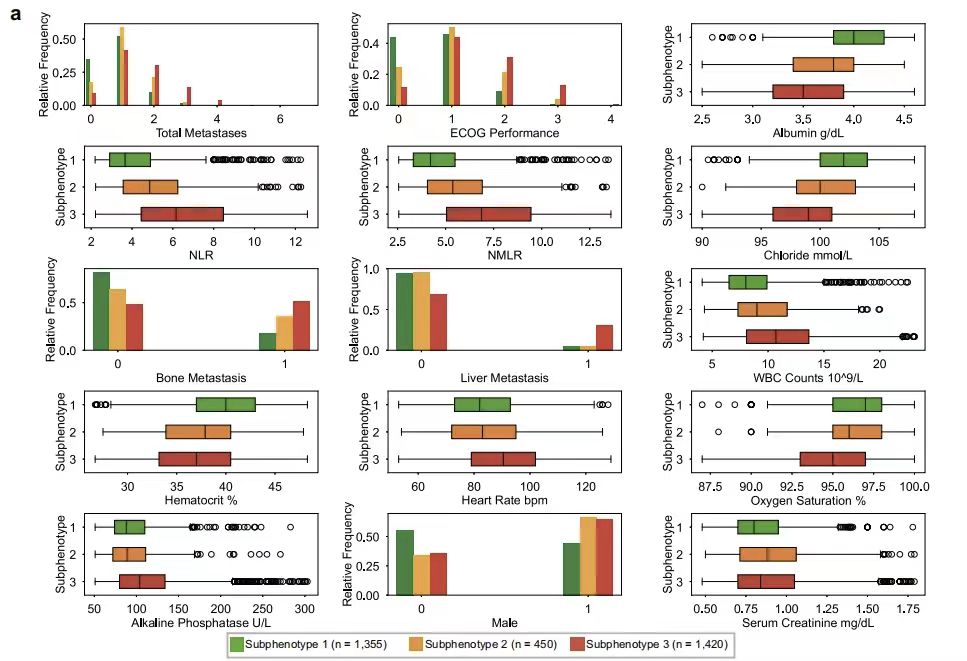
Pour mieux comprendre les différentes caractéristiques entre les différents sous-phénotypes, cette étude a testé les différences dans chaque variable entre les sous-phénotypes. Comme le montre la figure ci-dessous, l’analyse des facteurs prédictifs clés a montré que le statut de performance de l’Eastern Cooperative Oncology Group (ECOG Performance) et le nombre total de sites métastatiques (Total Metastases) sont les indicateurs clés permettant de distinguer les sous-phénotypes. En termes d'indicateurs de laboratoire, le rapport neutrophiles/lymphocytes (NLR) et le rapport neutrophiles/monocytes/lymphocytes (NMLR) sont des paramètres caractéristiques du sous-phénotype 2, tandis que le sous-phénotype 1 est associé à des niveaux d'albumine normaux (numération des globules blancs) et à un hématocrite élevé (hématocrite), et le sous-phénotype 3 est étroitement associé à des indicateurs tels qu'une augmentation de la fréquence cardiaque (fréquence cardiaque bpm), une diminution de la saturation en oxygène (saturation en oxygène) et une augmentation de la phosphatase alcaline (phosphatase alcaline).
Les résultats ci-dessus montrent queLe modèle GEMS permet non seulement une stratification précise du pronostic de survie des patients atteints d'un CPNPC acellulaire,De plus, grâce à l’analyse des caractéristiques des sous-phénotypes, il fournit une base de prise de décision clinique basée sur des données réelles pour la formulation de stratégies de traitement individualisées.
La révolution mondiale dans le diagnostic et le traitement de précision du cancer du poumon : comment l’IA et les technologies multi-omiques changent-elles le paysage de la survie ?
Dans le domaine du diagnostic et du traitement du cancer du poumon, une transformation impulsée par l’intelligence artificielle (IA) et la médecine de précision remodèle la pratique clinique. Une équipe de recherche de l’Université de Toronto au Canada a développé une technologie de test sanguin assistée par l’IA qui analyse les mutations de l’EGFR dans l’ADN tumoral circulant.La combinaison de l’apprentissage automatique avec les données cliniques améliore efficacement le taux de reconnaissance des personnes qui bénéficient d’un traitement ciblé.Il permet aux patients porteurs de mutations sensibles à l'EGFR de recevoir avec précision un traitement par inhibiteur de la tyrosine kinase (TKI) de l'EGFR, prolongeant ainsi considérablement la survie médiane sans progression.
Lien vers l'article :https://pubmed.ncbi.nlm.nih.gov/35624472/
Le système « evA.I. » de l’University College London utilise des données cliniques à 27 dimensions.Prédisez avec précision les réponses des inhibiteurs de points de contrôle immunitaire (ICI) et aidez à identifier les populations résistantes aux médicaments.Cela améliore l’efficacité de l’immunothérapie et prolonge la survie globale médiane.
Lien vers l'article :https://pmc.ncbi.nlm.nih.gov/articles/PMC10957591/
En Chine, des résultats innovants continuent d’émerger des universités et des entreprises dans la recherche sur le diagnostic de précision et le traitement du cancer du poumon non à petites cellules avancé. Par exemple,L'équipe du professeur Zhang Peng de l'Université de Tongji et l'équipe de l'Académie chinoise des sciences ont achevé la première étude internationale de cartographie génomique des protéines du cancer du poumon à petites cellules.,En intégrant les données omiques multidimensionnelles de 112 échantillons, nous avons découvert qu'une expression élevée de la protéine HMGB3 était associée à un mauvais pronostic et avons établi un modèle de prédiction des bénéfices de l'immunothérapie basé sur le statut de mutation ZFHX3, ouvrant une nouvelle voie pour un traitement de précision guidé par le typage moléculaire.
Lien vers l'article :https://doi.org/10.1016/j.cell.2023.12.004
L'École supérieure internationale de l'Université Tsinghua de Shenzhen et l'hôpital populaire de Shenzhen ont développé conjointement le système « IA + pathologie intelligente ».Après avoir étudié en profondeur plus de 3 000 cas difficiles, il peut identifier avec précision les types histologiques de cancer du poumon peu différencié avec un taux de précision de 97%.Raccourcir le cycle de prise de décision pour une thérapie ciblée. Le modèle de prédiction d'IA développé par son équipe à partir de marqueurs de glycoprotéines sanguines peut avertir du risque de cancer du poumon trois ans à l'avance, avec un taux de précision cliniquement vérifié dépassant 92%, offrant une solution non invasive pour un dépistage ultra-précoce.
Lien vers l'article :https://www.nature.com/articles/s41598-025-98731-4
Articles de référence :
1.https://mp.weixin.qq.com/s/LBcVbQUpTYRnKZ5I1KY_VA








