Command Palette
Search for a command to run...
Résultats De Sortie En 30 Minutes, l'Université Nationale De Singapour/MIT Et d'autres Construisent Un Modèle De Détection De Contamination Microbienne Basé Sur SVM
En tant que composant important des médicaments thérapeutiques avancés (ATMP), les produits de thérapie cellulaire (CTP) apportent de l’espoir aux patients atteints de maladies rares et incurables. Cependant, son processus de production est extrêmement sensible à l’invasion microbienne, et la contamination microbienne plane toujours sur cette lueur d’espoir comme une brume. Les méthodes traditionnelles de test de stérilité, telles que l'USP <71> (méthodes de test de stérilité enregistrées dans le chapitre 71 de la Pharmacopée des États-Unis), qui sont utilisées depuis un demi-siècle, semblent incapables de répondre aux nouvelles exigences de la médecine de précision : le cycle de culture de deux semaines, les étapes de prétraitement fastidieuses et les observations de turbidité qui reposent sur un jugement subjectif sont non seulement sérieusement en retard sur la courte durée de conservation des préparations cellulaires, mais peuvent également exposer les patients à des risques d'infection en raison d'une erreur de jugement.
Avec le développement fulgurant des produits de thérapie cellulaire, la détection rapide et précise de la contamination microbienne est devenue de plus en plus urgente. Une méthode de détection innovante – la spectroscopie d’absorption UV assistée par apprentissage automatique – a émergé. Il combine la technologie optique avec de puissantes méthodes d’apprentissage automatique. Cela ne nécessite pas une grande quantité de données de formation ou d’étapes d’enrichissement de la croissance. Seule une petite quantité d'échantillon est nécessaire pour produire les résultats du test en seulement 30 minutes, offrant ainsi une protection solide pour la sécurité des produits de thérapie cellulaire.
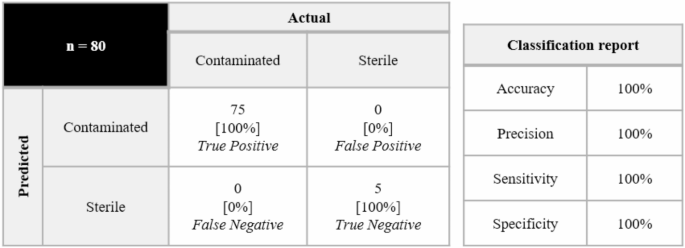
Récemment, une équipe de recherche conjointe de la Singapore-MIT Research Alliance, du Singapore A*SRL Laboratory, de l'Université nationale de Singapour et du Massachusetts Institute of Technology a proposé une méthode de détection combinant la spectroscopie d'absorption ultraviolette avec l'apprentissage automatique, qui peut compléter la détection de la contamination microbienne du surnageant de culture cellulaire en 30 minutes. Cette méthode utilise une machine à vecteur de support à une classe (SVM à une classe) pour analyser les différences caractéristiques entre la nicotinamide et la niacine dans le spectre ultraviolet, et présente un taux de vrai positif moyen de 92,7% lors du test de sept micro-organismes contaminants courants. Même après avoir exclu les échantillons de donneurs présentant un métabolisme anormal de la niacine, le taux de vrais négatifs atteint toujours 92%, dépassant de loin la précision des jugements traditionnels qui reposent sur l'expérience.
Les résultats de recherche pertinents ont été publiés dans la revue Scientific Reports de Nature sous le titre « Spectroscopie d'absorption UV assistée par apprentissage automatique pour la contamination microbienne dans les produits de thérapie cellulaire ».

Adresse du document :https://hyper.ai/en/sota/papers/s41598-024-83114-y
Suivez le compte public « HyperAI Super Neural » et répondez « Surveillance de la contamination microbienne » pour obtenir le PDF complet
Ensemble de données : Données d'échantillons de culture MSC stériles collectées à l'aide d'un spectromètre commercial
Dans cette étude, la construction et l’analyse de l’ensemble de données ont été étroitement liées à la culture de cellules stromales mésenchymateuses (MSC). Étant donné la large application de la thérapie MSC dans le traitement des lésions tissulaires aiguës, des maladies inflammatoires et des maladies dégénératives chroniques, l'équipe de recherche l'a sélectionnée comme objet de démonstration et a utilisé un spectromètre commercial pour collecter les spectres d'absorption d'échantillons de culture MSC stériles comme données de base pour la formation d'un modèle de machine à vecteurs de support à une classe (SVM à une classe). L'étude a adopté une stratégie de détection d'anomalies pour prédire l'état de contamination des échantillons de culture cellulaire en capturant les différences dans les caractéristiques spectrales, et a exploré le mécanisme potentiel du modèle SVM pour identifier la contamination sur la base de l'hypothèse de différences spectrales entre les métabolites de la niacine (NA) et de la nicotinamide (NAM).
Au cours de l'expérience, les chercheurs ont inoculé 10 UFC d'E. coli dans le système de culture MSC du donneur A et ont détecté avec succès le signal de contamination à 21 heures. Après avoir comparé les performances de détection des échantillons provenant de sept donneurs commerciaux, le donneur A a obtenu de bons résultats dans l’identification des échantillons stériles provenant d’autres donneurs et a donc été sélectionné comme source de données de formation initiales. Dans des études ultérieures, cette méthode a non seulement permis de détecter efficacement 7 types de micro-organismes avec des niveaux de contamination aussi bas que 10 UFC, mais a également vérifié la robustesse de la technologie grâce à des tests croisés entre donneurs.
Afin d'analyser en profondeur les performances du modèle SVM, la technique d'analyse en composantes principales (ACP) a été introduite dans cette étude. Les échantillons inoculés avec Pseudomonas aeruginosa ont été utilisés comme objet de recherche car ils ont montré la concentration de NA la plus élevée lors de la détection par chromatographie liquide-spectrométrie de masse (LC-MS). La distribution des ensembles de données d’entraînement et des échantillons contaminés a été visualisée via PCA. Les résultats ont montré que les échantillons contaminés et les échantillons stérilisés étaient significativement séparés dans l’espace, distinguant efficacement les états stériles et contaminés. Les vecteurs de chargement du composant principal 1 (PC 1) et du composant principal 2 (PC 2) ont été analysés plus en détail, et les spectres d'absorption UV normalisés de 100 μg/mL de NA et de NAM dans du PBS ont été combinés pour présenter intuitivement l'association entre les caractéristiques spectrales et les composants principaux.
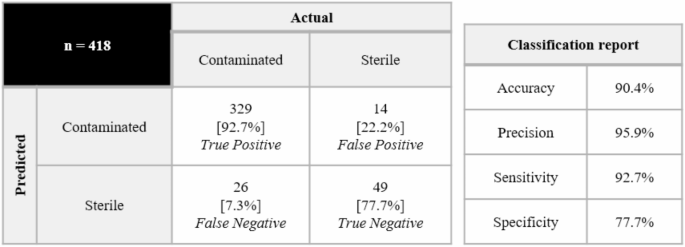
Dans la phase de vérification de la robustesse du modèle, l'étude a collecté des échantillons de milieu de culture usagé et de stérilisation PBS auprès de 6 donneurs commerciaux (donneur BG) pour construire un ensemble de données de test inter-donneurs. En formant des modèles SVM à partir de différentes sources de donateurs et en effectuant une validation croisée, il a été constaté que le modèle formé sur la base des donateurs A et B avait une précision de prédiction plus élevée. L'analyse de 418 échantillons de test a montré que le taux de vrais positifs du modèle atteignait 92,7% et que la limite de détection était maintenue de manière stable à 10 UFC ; Cependant, le taux de vrais négatifs de 77,7% indiquait qu'il y avait encore place à l'amélioration, parmi lesquels le problème de faux positifs causé par des niveaux anormaux de NA dans l'échantillon du donneur F était particulièrement important, ce qui a indiqué la direction de l'optimisation ultérieure de l'adaptabilité du modèle aux différences métaboliques entre les donneurs.
Spectroscopie d'absorption ultraviolette assistée par apprentissage automatique : la machine à vecteurs de support comme algorithme de base
Afin de permettre à l’apprentissage automatique de mieux aider à la détection de la contamination microbienne, cette étude l’a combiné de manière innovante avec la spectroscopie d’absorption ultraviolette pour proposer une méthode rapide, sensible et rentable de détection de la contamination microbienne. Cette méthode utilise la machine à vecteurs de support (SVM) comme algorithme de base et identifie avec précision la contamination microbienne en analysant les caractéristiques du spectre d'absorption ultraviolette du milieu de culture cellulaire.
En termes de construction du modèle, les chercheurs ont utilisé une machine à vecteurs de support à classe unique et ont sélectionné la fonction de base radiale (RBF) comme fonction noyau, en définissant la valeur γ à 0,002 et la valeur ν à 0,2. L'ensemble de données de formation comprenait le donneur 5 des jours 2 à 7 des passages 2, 4 et 6, et des échantillons stériles enrichis en PBS du donneur 8, qui étaient tous étiquetés 1, représentant l'état stérile. Les données d'absorbance de chaque échantillon couvraient la gamme de longueurs d'onde de 237 nm à 300 nm pour se concentrer sur les caractéristiques spectrales importantes de la niacine (NA) et de la nicotinamide (NAM) et éviter les interférences dues au bruit dans d'autres bandes. Tous les échantillons ont été centrés sur la moyenne avant l’apprentissage du modèle pour corriger les écarts spectraux causés par la dérive de l’instrument et d’autres facteurs, améliorant ainsi la précision du modèle.
En termes de validation et d'application du modèle, la spectroscopie d'absorption UV assistée par apprentissage automatique a été utilisée pour démontrer la capacité de cette méthode à détecter des polluants à faible concentration en ajoutant 10 UFC d'E. coli à la culture MSC du donneur A et en extrayant des échantillons de surnageant trois fois à des intervalles de 3 heures entre 9 et 24 heures. Les résultats sont présentés dans la figure ci-dessous. Le modèle SVM a pu prédire avec précision que l’échantillon était contaminé après 21 heures, et le temps de détection total était d’environ 21,5 heures.
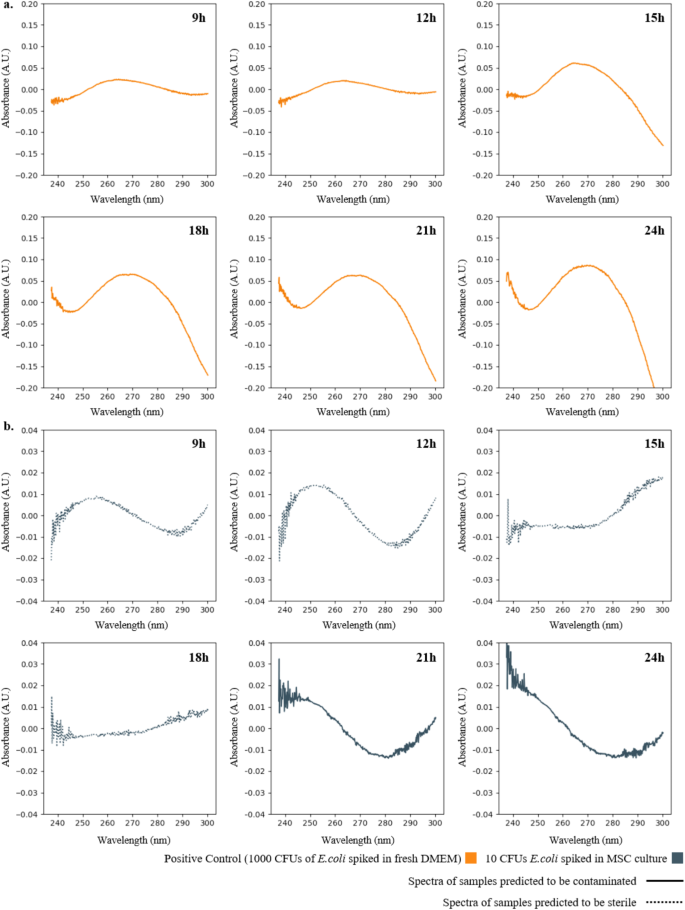
En outre, l’étude a comparé la spectroscopie d’absorption UV assistée par apprentissage automatique avec les méthodes existantes. Les résultats ont montré que la méthode de spectroscopie d'absorption UV assistée par apprentissage automatique avait un temps de détection (TTD) de 21 heures, ce qui est comparable au test USP <71> (24 heures jusqu'à ce que la turbidité soit observée) et à la méthode de calcul du rapport NA/NAM par LC-MS (18 heures). Cependant, BacT/Alert® 3D et USP <71> nécessitent tous deux des opérateurs formés pour extraire des échantillons de cultures cellulaires et les inoculer dans divers milieux d'enrichissement de croissance. Le flux de travail de la spectroscopie d'absorption UV est relativement simple et ne nécessite pas d'inoculation supplémentaire dans le milieu d'enrichissement de la croissance, ni de temps d'incubation et de préparation d'échantillon supplémentaires, éliminant ainsi les ressources et les coûts supplémentaires requis pour l'étape d'inoculation d'enrichissement de la croissance observée.
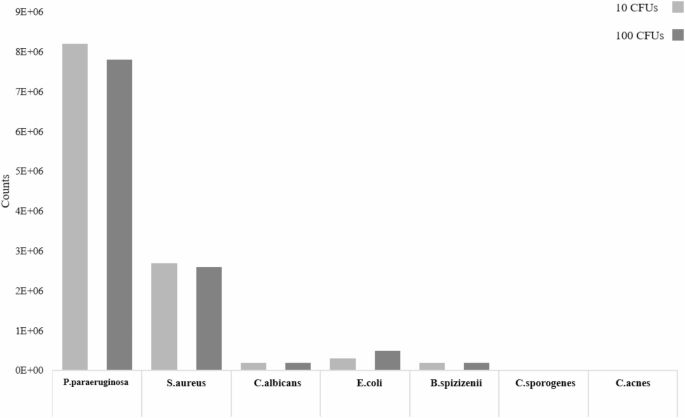
Pour déterminer si la méthode proposée de spectroscopie d'absorption UV assistée par apprentissage automatique peut être appliquée à d'autres micro-organismes, les chercheurs ont utilisé des échantillons stérilisés au PBS du donneur A et ont appliqué la méthode à la détection d'autres organismes à croissance lente tels que Staphylococcus aureus (S. aureus), Pseudomonas aeruginosa (P. paraeruginosa), Bacillus subtilis (B. spizizenii), Clostridium perfringens (C. sporogenes), des levures telles que Candida albicans (C. albicans), Escherichia coli K-12 (E. coli) et Propionibacterium acnes (C. acnes), avec une limite de détection (LoD) aussi basse que 10 UFC. En outre, l'étude a également visualisé les performances du modèle SVM grâce à l'analyse en composantes principales (ACP) et a constaté que les échantillons inoculés avec Pseudomonas aeruginosa étaient clairement distingués des échantillons stérilisés au PBS dans le graphique ACP, indiquant que le modèle peut capturer efficacement les différences dans les caractéristiques spectrales.
En termes d'étude de robustesse du modèle, considérant que les différences entre les donneurs peuvent affecter les performances du modèle, les chercheurs ont collecté des échantillons de milieu de culture usagé auprès de 6 donneurs commerciaux (donneur BG) et ont préparé des échantillons stérilisés au PBS. En formant des modèles SVM basés sur différents donneurs et en évaluant leur précision de prédiction sur des échantillons provenant d’autres donneurs, il a été constaté que les modèles des donneurs A et B avaient des taux de prédiction moyens plus élevés. Par conséquent, dans cette étude, le modèle SVM formé sur la base des donneurs A et B a ensuite été appliqué à des échantillons provenant des 6 autres donneurs. Les résultats ont montré que la spectroscopie d’absorption UV assistée par apprentissage automatique a atteint un taux de vrais positifs de 92,7%, et la limite de détection (LoD) est restée systématiquement à 10 UFC pour les sept micro-organismes testés. Cependant, le taux de vrais négatifs du modèle était de 77,7%, et les échantillons faussement positifs provenaient principalement du donneur F. L'analyse a révélé que le niveau de NA dans les échantillons du donneur F était plus élevé, ce qui suggère que le modèle doit être davantage optimisé à l'avenir pour améliorer son adaptabilité aux différences entre les donneurs.
AI4S renforce la thérapie cellulaire : la recherche scientifique, l'industrie et les politiques progressent en synergie
Ces dernières années, de la recherche scientifique au monde des affaires, des laboratoires à l’industrialisation, les produits de thérapie cellulaire (CTP) ont connu un grand succès.
Dans le domaine de la recherche fondamentale, l'équipe TJ-AI4S de l'Université Tongji de Shanghai, en Chine, a remporté le concours mondial d'algorithmes de développement de médicaments par l'IA. La stratégie d’extension des caractéristiques moléculaires qu’ils ont proposée a amélioré la capacité de généralisation du modèle et a fourni de nouvelles idées pour la construction de la bibliothèque d’empreintes moléculaires polluantes CTP. Le cadre UniBind, développé par l'Université des postes et télécommunications de Pékin et l'Université de Pékin en Chine, analyse les interactions protéiques via des réseaux neuronaux graphiques multi-échelles, fournissant une base informatique pour étudier les associations dynamiques entre les cytokines et les métabolites microbiens dans le CTP.
Lien vers l'article : https://www.nature.com/articles/s41591-023-02483-5
De plus, une équipe de l'Université ShanghaiTech en Chine a développé la plateforme CAR-Toner, qui utilise des algorithmes d'IA pour optimiser la distribution de charge des molécules CAR, améliorant ainsi avec succès l'efficacité de l'expansion cellulaire et réduisant les différences de lots. Cette innovation améliore non seulement l’efficacité de la production, mais fournit également un nouveau support technique pour la production standardisée de CTP.
Lien vers l'article : https://www.nature.com/articles/s41422-024-00936-1
L’industrie a également reçu de bonnes nouvelles. Le médicament CAR-T « Yikeda » de la société de biotechnologie chinoise Fosun Kairui a réalisé son premier approvisionnement transfrontalier en Chine en 2025. Derrière cela se cache un système de gestion logistique de la chaîne du froid piloté par l'IA qui garantit l'activité des cellules pendant le transport à très basse température. En outre, la société de biotechnologie américaine A2 Bio a réalisé une préproduction à grande échelle de médicaments CAR-T grâce au criblage par IA de cellules donneuses universelles, ce qui a considérablement réduit les coûts de production et raccourci la période d'attente pour le traitement, apportant plus d'espoir aux patients.
Les institutions de recherche étrangères ont également obtenu des résultats fructueux dans le domaine de la thérapie cellulaire basée sur l’IA. En 2025, une équipe d'ingénierie biomédicale de l'Université Duke aux États-Unis a développé la technologie PepPrCLIP, qui a conçu des peptides courts fonctionnels basés sur le modèle de langage protéique ESM-2, offrant une nouvelle stratégie pour le traitement de précision du cancer. Le modèle de prédiction d'IA construit par IBM Watson Health en collaboration avec l'Université de Stanford aux États-Unis a amélioré la précision de prédiction du syndrome de libération de cytokines (CRS) dans la thérapie CAR-T au 89% en intégrant le génome du patient, le protéome et les données cliniques multidimensionnelles, aidant ainsi à l'intervention clinique chez les patients à haut risque à l'avance. Ces avancées ont insufflé une nouvelle vitalité au développement du domaine mondial de la thérapie cellulaire.
Actuellement, AI4S a construit une chaîne complète allant de la recherche fondamentale à l’application clinique dans le domaine du CTP, mais elle doit encore être améliorée dans des aspects tels que la normalisation des données et les mécanismes de collaboration interinstitutionnelle. À l’avenir, avec un soutien politique accru et une intégration approfondie de l’industrie, du monde universitaire et de la recherche, l’AI4S devrait réaliser de plus grandes avancées dans des domaines tels que la préparation personnalisée du CTP, la traçabilité de la qualité en temps réel et l’optimisation de la logistique transfrontalière.
Articles de référence :
- https://mp.weixin.qq.com/s/VZI7pm-kO7CxNJyrn9-qQw
- https://mp.weixin.qq.com/s/0AoP5XSLOLzcTChQoIIfmw
- https://mp.weixin.qq.com/s/Avu5SbLetCFBVUwnDybfsw
- https://mp.weixin.qq.com/s/WoruStfCdYNDskAn_iFYtQ
- https://www.nsfc.gov.cn/publish/portal0/tab1128/info90687.htm








