Command Palette
Search for a command to run...
Couvrant Plus De 40 Modèles Et Ensembles De Données Grand Public, l'équipe De l'Université Jiao Tong De Shanghai a Lancé La plate-forme Unique De Conception d'ingénierie Des Protéines VenusFactory

Avec le développement rapide de l’intelligence artificielle et des méthodes basées sur les données, l’ingénierie des protéines évolue vers le stade de la conception assistée par l’IA. Les chercheurs ont plus que jamais besoin d’ensembles de données protéiques complets et de haute qualité, de modèles d’intelligence artificielle protéiques plus puissants et influents, et de plateformes d’analyse plus efficaces et standardisées, afin d’extraire avec précision des informations précieuses à partir de données biologiques massives, d’accélérer la conception et l’optimisation de nouvelles protéines et de promouvoir des percées innovantes en biomédecine, en biologie synthétique et dans d’autres domaines.
Dans ce contexte, de plus en plus de praticiens des sciences de la vie souhaitent comprendre l’IA et utiliser la technologie de l’IA pour aider à la conception de l’ingénierie des protéines. Cependant, la solution open source repensée de David Baker et la série de grands modèles ESM de Meta présentent de nombreuses difficultés d'utilisation, telles que la logique complexe du cadre de calcul de l'IA, la grande quantité de code et la nécessité d'une base de programmation informatique solide. En d’autres termes, les chercheurs en biologie et même les informaticiens non expérimentés sont toujours confrontés à un seuil d’utilisation assez élevé. À cet égard, les applications low-code conviviales sont progressivement devenues la tendance dominante dans l’utilisation des outils open source modernes. Ils peuvent aider les chercheurs à se débarrasser de la configuration complexe des modèles et de l'implémentation du code, permettant aux informaticiens et aux biologistes d'appeler ou de former des modèles d'apprentissage en profondeur de manière plus pratique et de se concentrer sur la recherche scientifique elle-même.
Afin de promouvoir l'application et le développement de l'intelligence artificielle dans le domaine de l'ingénierie des protéines, le groupe de recherche du professeur Hong Liang à l'Université Jiao Tong de Shanghai en Chine a développé VenusFactory, une plate-forme ouverte unique conçue pour l'ingénierie des protéines. Les chercheurs peuvent facilement mettre en œuvre la récupération fastidieuse de données, la formation de modèles, l'évaluation des tâches, le déploiement de modèles et d'autres fonctions via l'interaction d'interface ou la ligne de commande. Grâce à une conception sans code et basée sur les processus, la plateforme simplifie les opérations d'ingénierie d'IA complexes du passé en opérations légères à portée de main. Les chercheurs peuvent démarrer des services Web localement et appeler facilement plus de 40 modèles d'apprentissage en profondeur de protéines de pointe sans écrire de codes complexes, protégeant ainsi la confidentialité des données privées, abaissant considérablement le seuil de recherche scientifique intelligente et accélérant l'application approfondie de l'IA dans le domaine des sciences de la vie.
Le code et les données sont open source sur : https://github.com/ai4protein/VenusFactory
Actuellement, la « VenusFactory Protein Engineering Design Platform » a été lancée dans la section tutoriel du site Web HyperAI. Le tutoriel d'utilisation détaillé est joint à la fin de cet article. Les lecteurs intéressés peuvent découvrir la plateforme via le lien ci-dessous :
VenusFactory : une plateforme unifiée qui élimine les obstacles aux applications de l'IA protéique
Les données sur les protéines sont très dispersées. VenusFactory accède directement à la source des données biologiques La recherche sur les protéines de l’IA dépend fortement des données biologiques à grande échelle, et les données annotées sont distribuées dans plusieurs bases de données publiques grand public. Les scientifiques doivent souvent basculer entre plusieurs bases de données, télécharger manuellement les données et écrire des scripts pour convertir le format, ce qui entraîne une perte de temps et d’énergie sur des travaux de recherche non pratiques. VenusFactory se connecte directement aux bases de données publiques courantes, telles que RCSB PDB, UniProt, InterPro, etc. Le téléchargement multithread à grande vitesse améliore considérablement l'efficacité de la récupération des données :
- Accès unique à la séquence protéique, à la structure tridimensionnelle et à l'annotation fonctionnelle, intégrant pleinement les informations biologiques.
- La sortie au format standardisé évite les problèmes de compatibilité des données et facilite la formation directe de l'IA.
- Le mécanisme de téléchargement multithread améliore considérablement la vitesse d’acquisition des données, permettant aux scientifiques de se concentrer sur la recherche elle-même.
Le système d’évaluation des tâches d’IA protéique n’est pas unifié. VenusFactory couvre cinq tâches principales. À l’heure actuelle, le système d’évaluation des modèles d’IA protéique manque de données de référence fiables et prêtes à l’emploi, et la plupart des recherches se concentrent encore sur l’optimisation des tâches individuelles. Lorsque les chercheurs choisissent une solution, ils doivent souvent consacrer beaucoup de temps supplémentaire à des comparaisons expérimentales. VenusFactory intègre plus de 40 ensembles de données d'évaluation d'ingénierie des protéines de pointe, couvrant cinq tâches principales :
- Prédiction de la fonction des protéines:Prédire les étiquettes fonctionnelles des protéines pour aider à découvrir de nouvelles enzymes et de nouvelles cibles.
- Prédiction de la localisation subcellulaire des protéines :Prédire la localisation des protéines dans les cellules pour faciliter le diagnostic des maladies.
- Évaluation de la solubilité des protéines:Améliorez l'efficacité de l'expérience humide en préjugant de la solubilité.
- Analyse des effets des mutations protéiques:Explorez l’impact potentiel des mutations génétiques et faites progresser la médecine de précision.
- Autres tâches de prédiction:Tels que la liaison des ions métalliques, la prédiction du signal de tri des protéines, la prédiction de la température optimale, etc.
À l’aide de ces ensembles de données de référence et de ces résultats d’évaluation, les utilisateurs peuvent facilement comparer les performances de différents modèles et sélectionner et optimiser des solutions. Dans le même temps, VenusFactory fournit également la fonction de téléchargement de tous les ensembles de données, afin que les utilisateurs puissent obtenir la séquence protéique, la structure, l'étiquette et d'autres informations correspondantes en un seul clic.
Les outils informatiques d'IA protéique existants présentent des obstacles importants à leur utilisation et sont difficiles à utiliser pour les chercheurs sans formation informatique. L’utilisation des modèles actuels d’IA protéique nécessite souvent de solides compétences en programmation et des connaissances en apprentissage approfondi. Pour la plupart des biologistes, la formation, le réglage fin et l’application des modèles d’IA restent une tâche à haut niveau de compétence. VenusFactory intègre plus de 40 modèles de langage protéique (PLM) de pointe au monde, couvrant des solutions complètes de grands modèles d'IA, telles que la série Venus (ProSST, Pro-Prime, PETA, etc.), la série ESM (ESM2, ESM1b, etc.), la série Ankh (Base, Large) et la série ProtTrans (ProtBert, ProtT5), etc.
- Écosystème de modèles pré-entraînés:Appelez directement le PLM open source sans formation à partir de zéro, économisant ainsi des ressources informatiques.
- Réglage fin haute performance: Prend en charge des méthodes de pointe telles que LoRA et SES-Adapter pour adapter le modèle à des tâches biologiques spécifiques.
- Prise en charge multitâche:Qu'il s'agisse de prédiction de la solubilité des protéines ou de prédiction des propriétés mutantes, vous pouvez démarrer facilement.
- Mode ligne de commande:Adapté aux informaticiens, il peut ajuster les paramètres de manière flexible et réaliser une optimisation approfondie.
- Interface Web sans code:Convient aux biologistes, vous pouvez exécuter des tâches d'IA en quelques clics, aucune connaissance en programmation n'est requise !
Pour relever ces défis fondamentaux, VenusFactory a construit une plateforme unique d'ingénierie des protéines basée sur l'IA, offrant une solution complète allant de l'acquisition de données, de l'évaluation des tâches à l'affinage du modèle, permettant aux biologistes et aux informaticiens de faire progresser leurs recherches efficacement.
Open source et développement communautaire pour promouvoir l'innovation scientifique
L’avenir de la recherche scientifique réside dans le partage ouvert. VenusFactory utilise la licence Apache 2.0. Tous les codes, ensembles de données et pondérations de modèles sont entièrement open source. Les utilisateurs peuvent télécharger, modifier, optimiser et partager librement les derniers résultats avec les chercheurs du monde entier. Toutes les données, modèles et codes de réglage fin sont hébergés sur GitHub et Hugging Face, garantissant que les scientifiques du monde entier peuvent facilement accéder et reproduire des expériences et créer leurs propres projets de recherche en IA basés sur VenusFactory.
Pour aider les lecteurs à découvrir VenusFactory, HyperAI a lancé un didacticiel de déploiement en un clic pour la « VenusFactory Protein Engineering Design Platform ». Ce qui suit est une introduction détaillée à son utilisation.
Lien du tutoriel : https://go.hyper.ai/ZqO3h
Tutoriel sur la plateforme de conception d'ingénierie des protéines VenusFactory
Essai de démonstration
1. Connectez-vous à hyper.ai, sur la page Tutoriel, sélectionnez VenusFactory Protein Engineering Design Platform et cliquez sur Exécuter ce tutoriel en ligne.
2. Une fois la page affichée, cliquez sur « Cloner » dans le coin supérieur droit pour cloner le didacticiel dans votre propre conteneur.
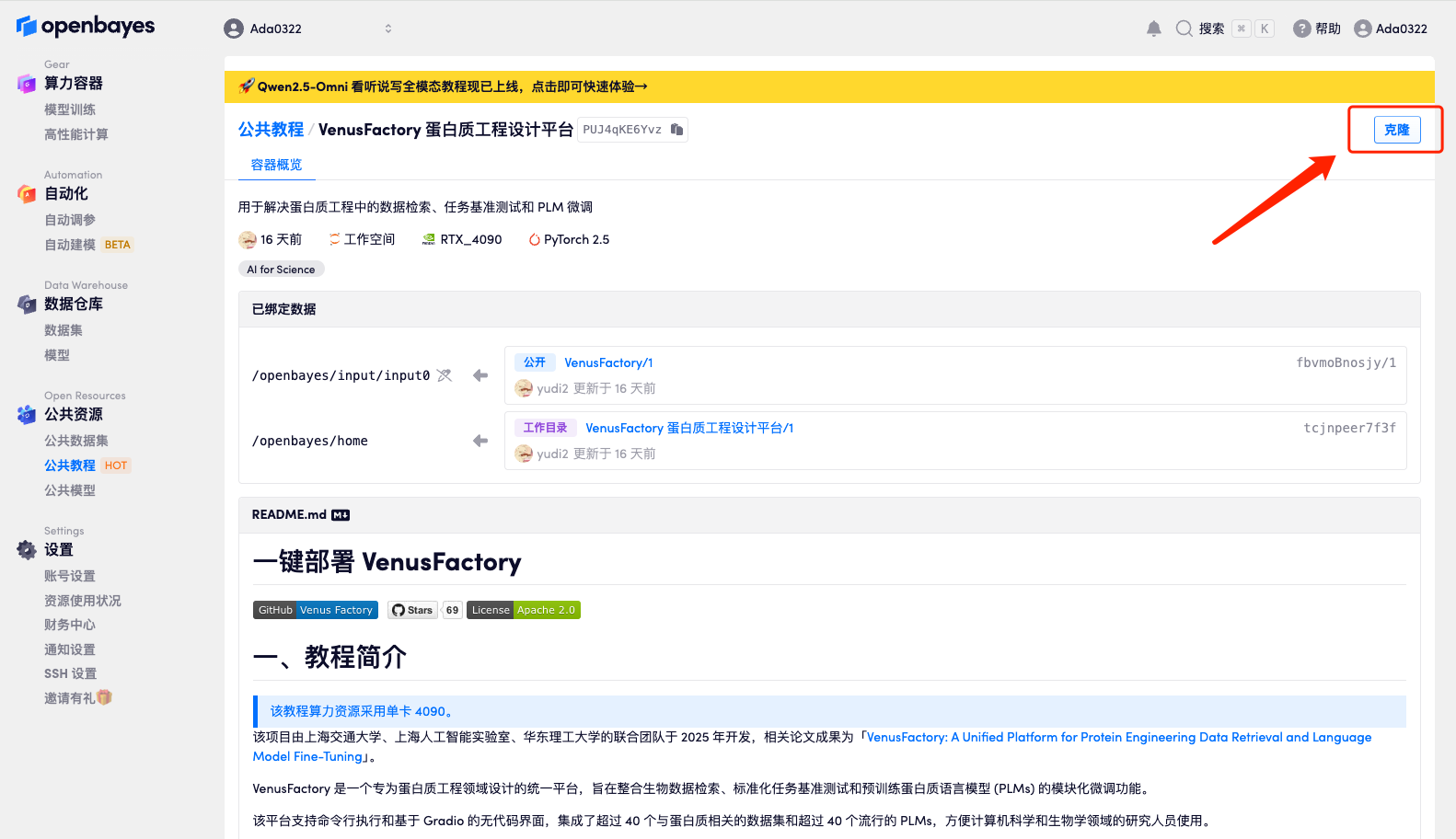
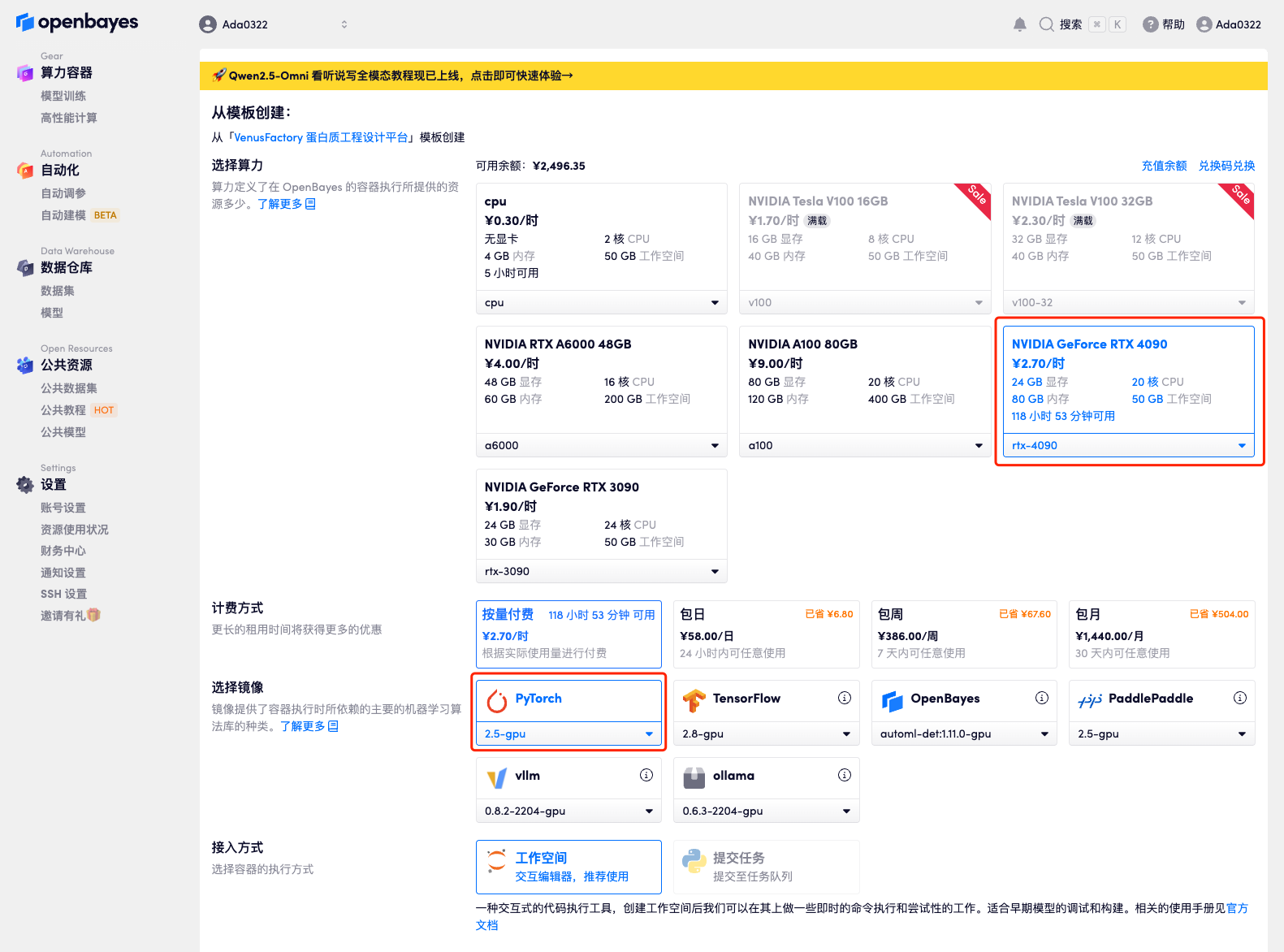
3. Sélectionnez les images NVIDIA GeForce RTX 4090 et PyTorch, puis cliquez sur Continuer. La plateforme OpenBayes propose quatre méthodes de facturation. Vous pouvez choisir « payer à l'utilisation » ou « quotidien/hebdomadaire/mensuel » selon vos besoins. Les nouveaux utilisateurs peuvent s'inscrire en utilisant le lien d'invitation ci-dessous pour obtenir 4 heures de RTX 4090 + 5 heures de temps CPU gratuit !
Lien d'invitation exclusif HyperAI (copier et ouvrir dans le navigateur) :
https://openbayes.com/console/signup?r=Ada0322_NR0n
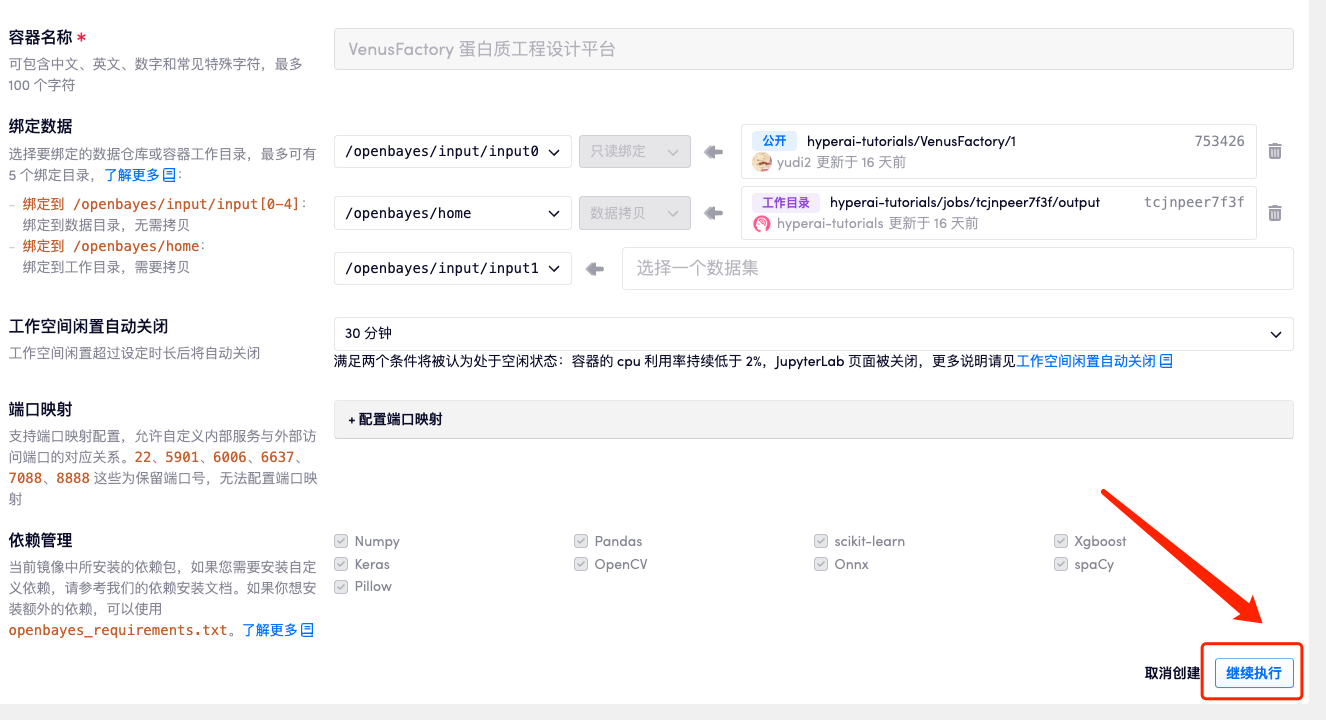
4. Attendez que les ressources soient allouées. Le premier processus de clonage prend environ 2 minutes. Lorsque le statut passe à « En cours d'exécution », cliquez sur la flèche de saut à côté de « Adresse API » pour accéder à la page de démonstration. Étant donné que le modèle est volumineux, il faut environ 3 minutes pour afficher l'interface WebUI, sinon « Bad Gateway » s'affichera. Veuillez noter que les utilisateurs doivent effectuer l'authentification par nom réel avant d'utiliser la fonction d'accès à l'adresse API.
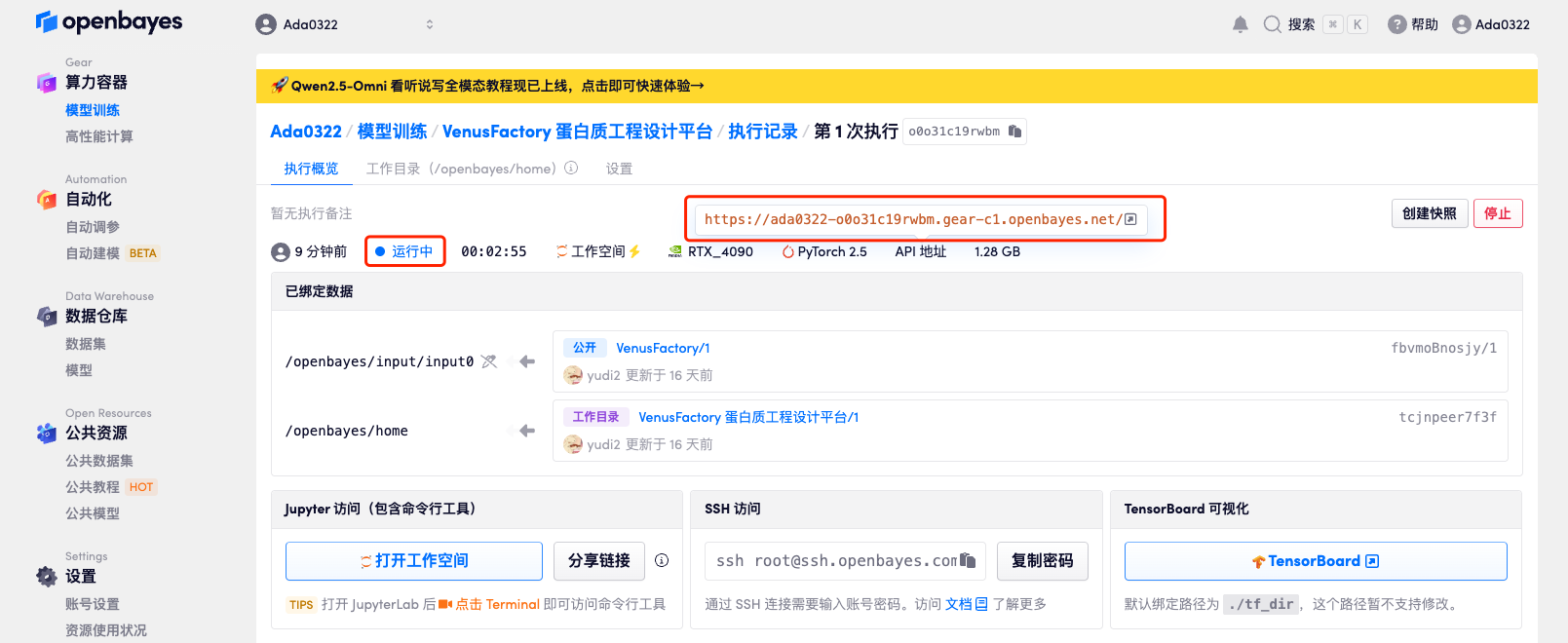
Affichage des effets
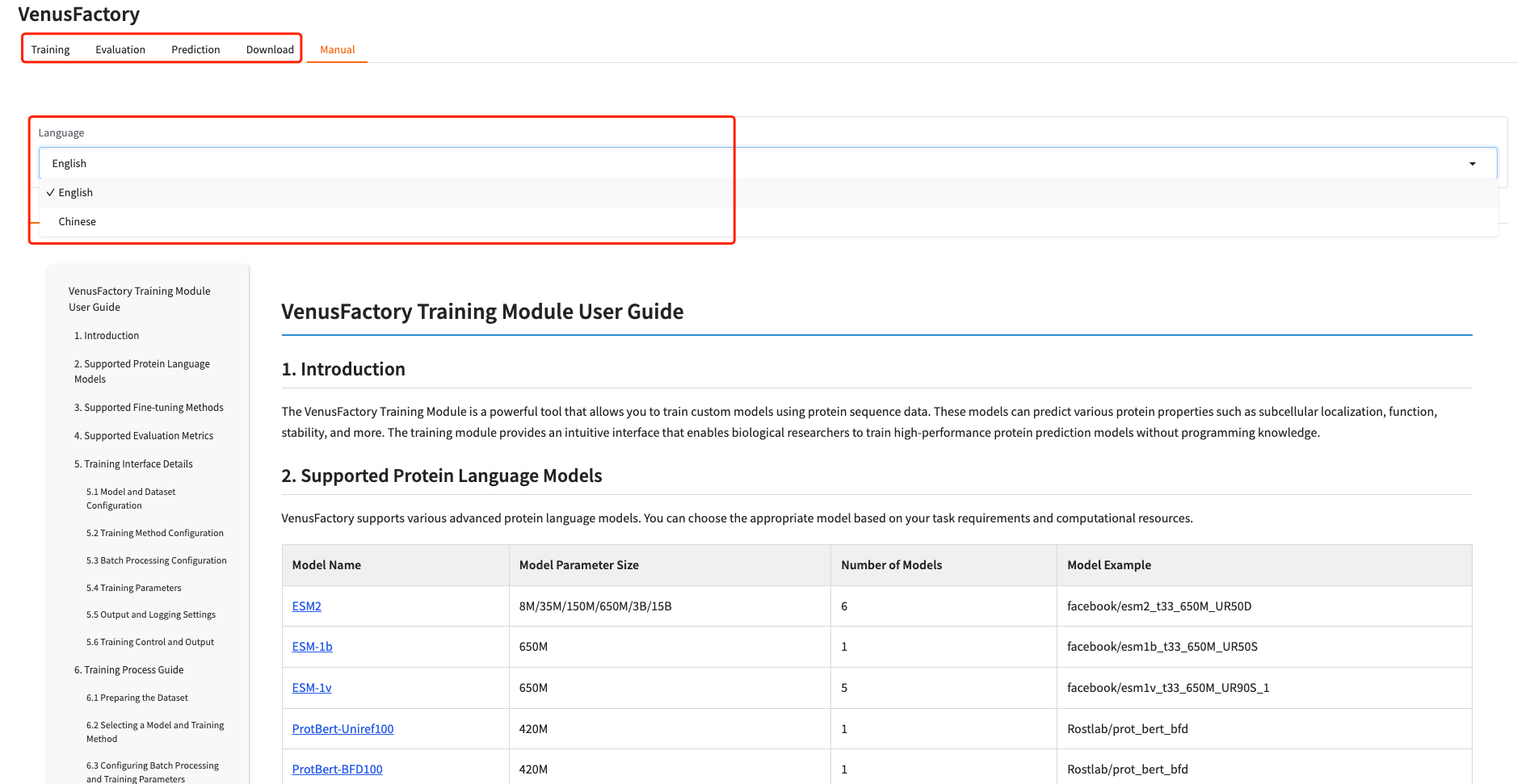
1. Ce tutoriel comprend quatre modules : Formation, Évaluation, Prédiction et Téléchargement. Cliquez sur Manuel et sélectionnez une langue pour voir les instructions détaillées pour chaque module.
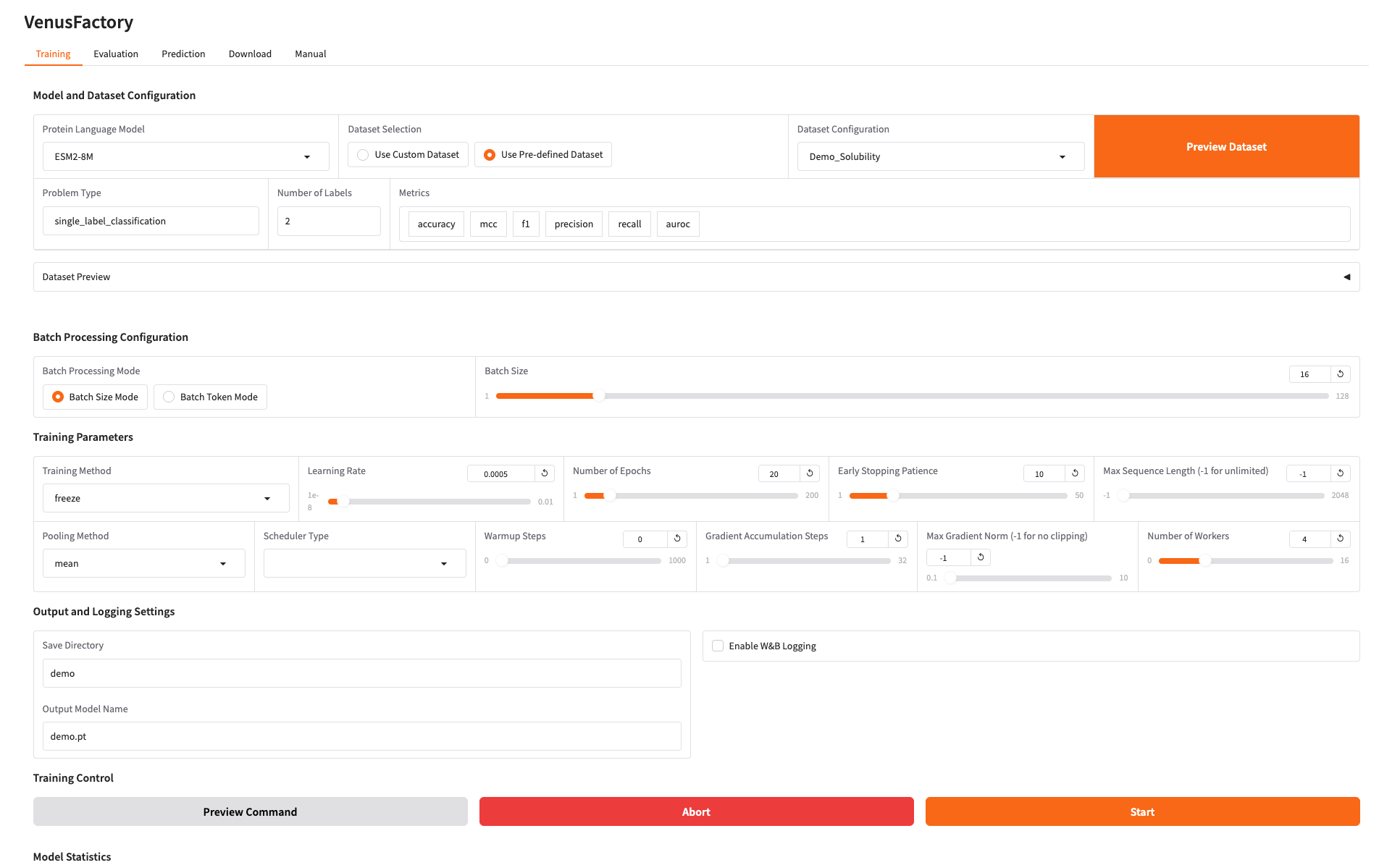
2. Module de formation
Cliquez sur le module Formation, sélectionnez le modèle que vous souhaitez former dans le modèle de langage des protéines et configurez les données de formation dans la configuration du jeu de données.
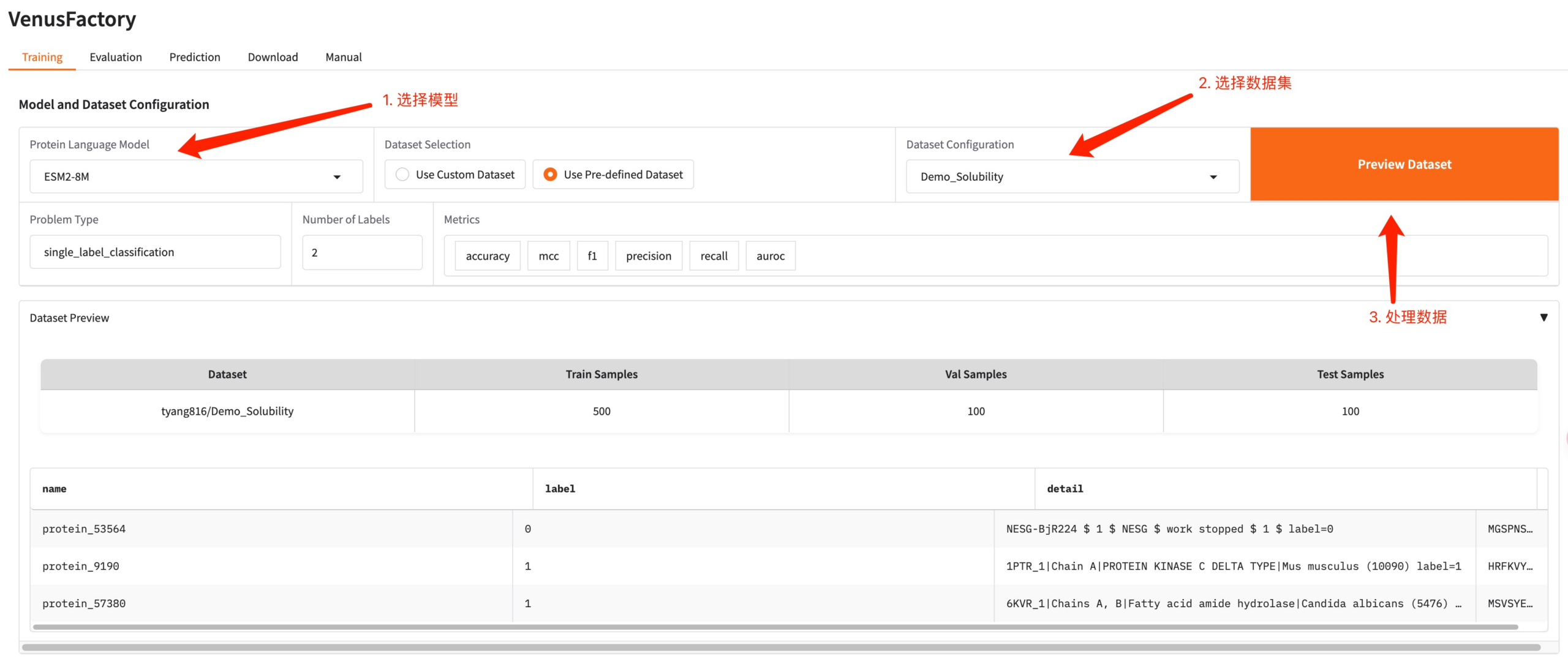
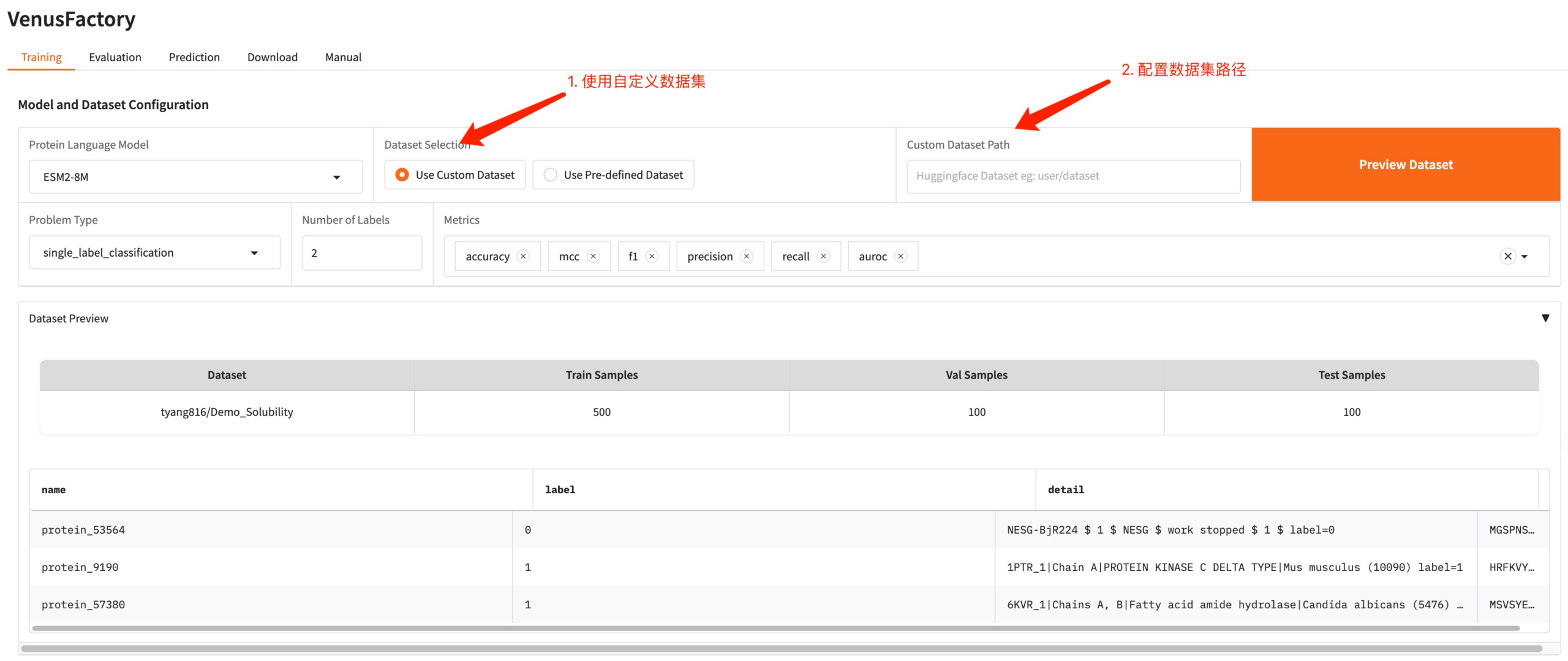
Si vous devez utiliser votre propre ensemble de données, vous pouvez utiliser la configuration Utiliser un ensemble de données personnalisé et il vous suffit de renseigner le chemin de l'ensemble de données (consultez la documentation d'utilisation manuelle pour plus de détails).
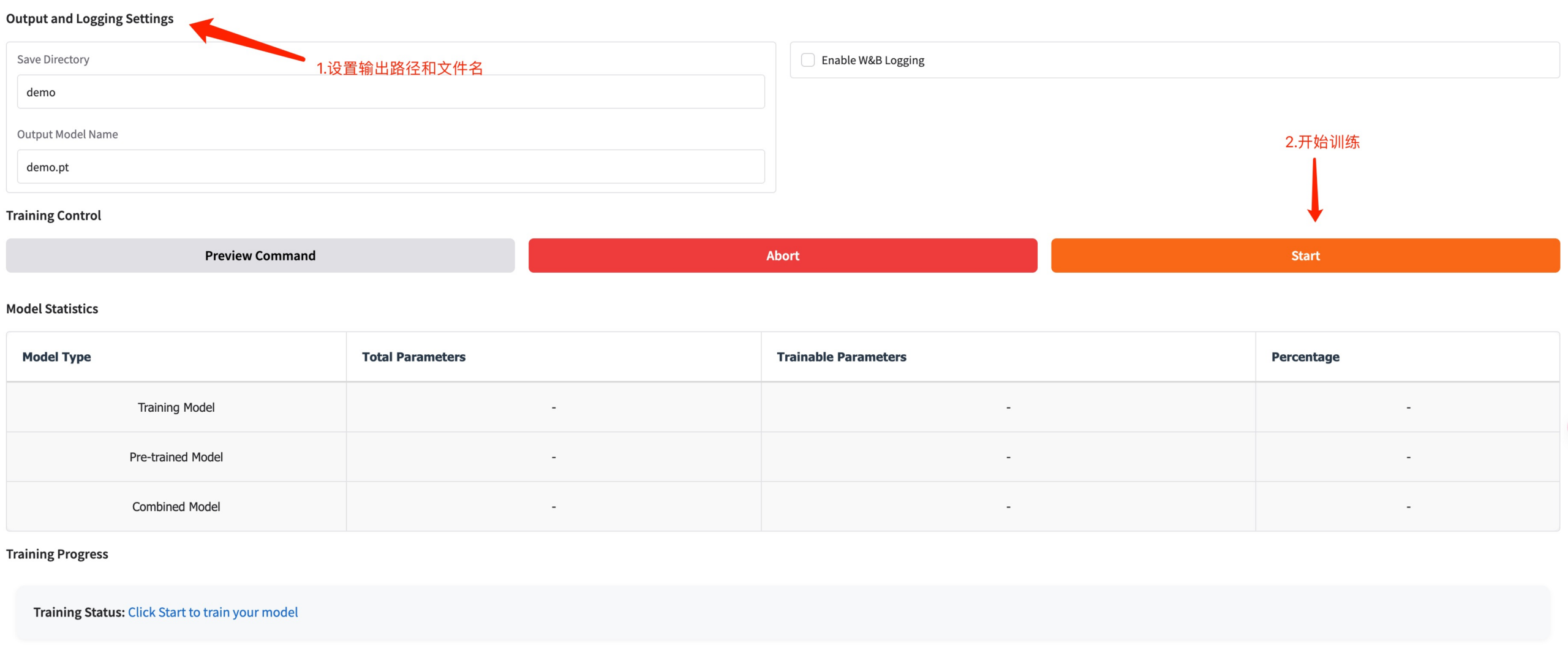
Définissez le chemin d’enregistrement du modèle de formation et cliquez sur Démarrer pour démarrer la formation.
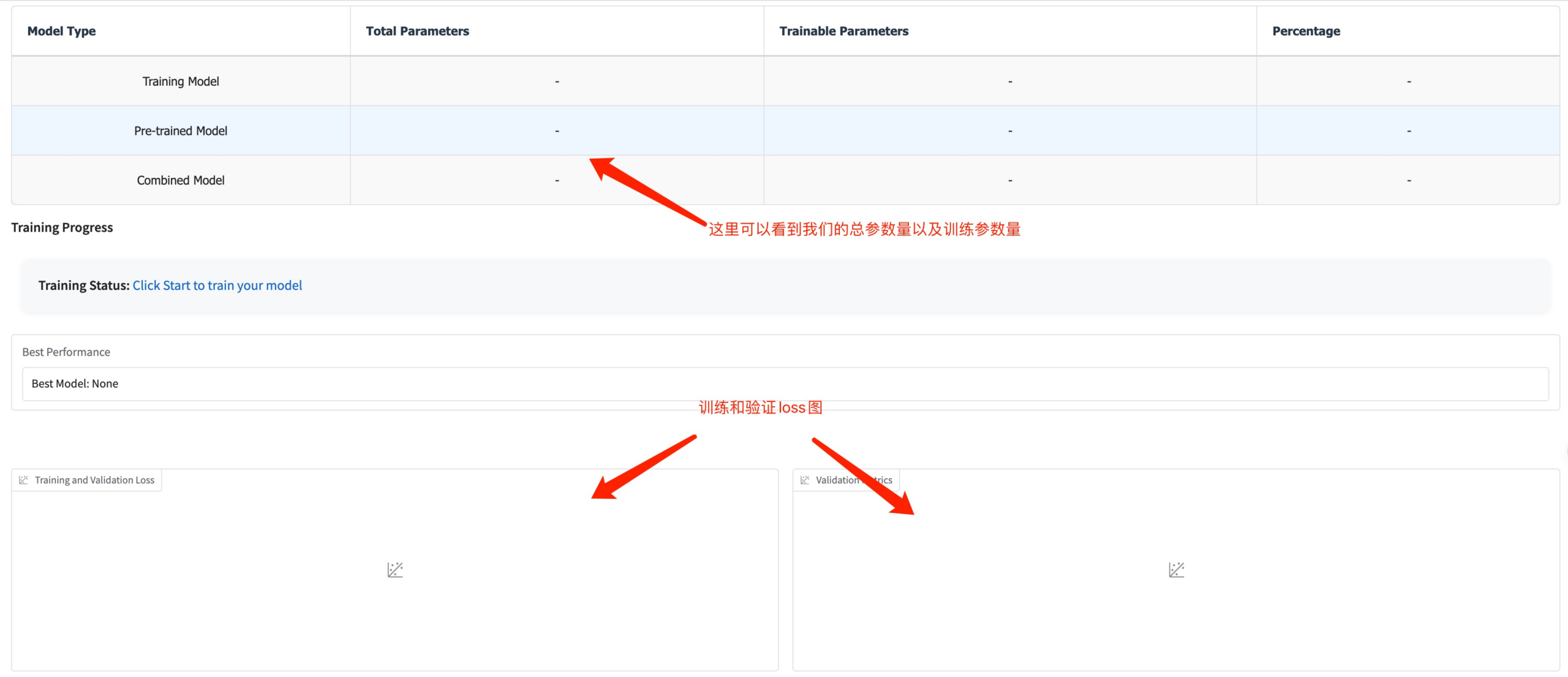
À ce stade, vous pouvez voir les paramètres d’entraînement et la courbe de perte
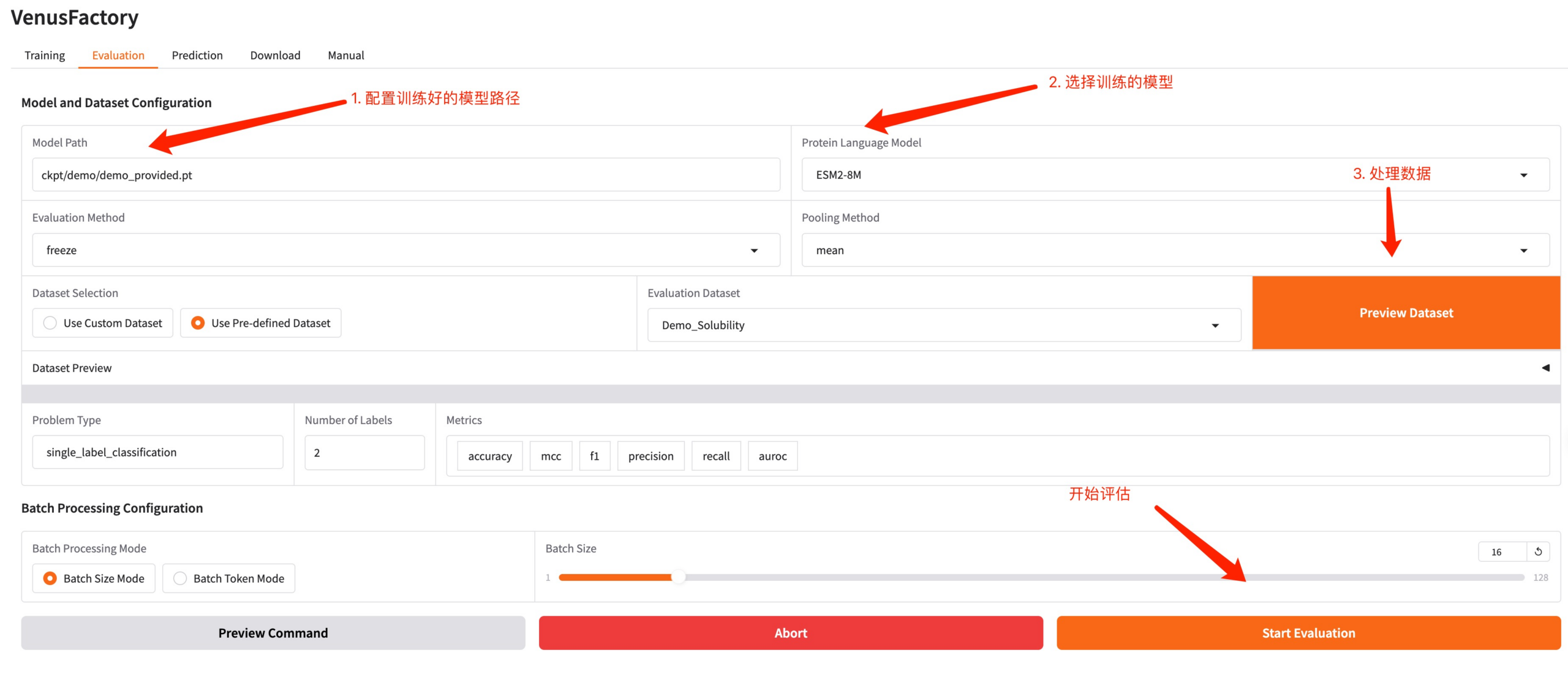
3. Module d'évaluation
Cliquez sur le module Évaluation, configurez le chemin du modèle généré par la formation et le modèle formé, traitez les données, ajustez les hyperparamètres et démarrez l'évaluation.
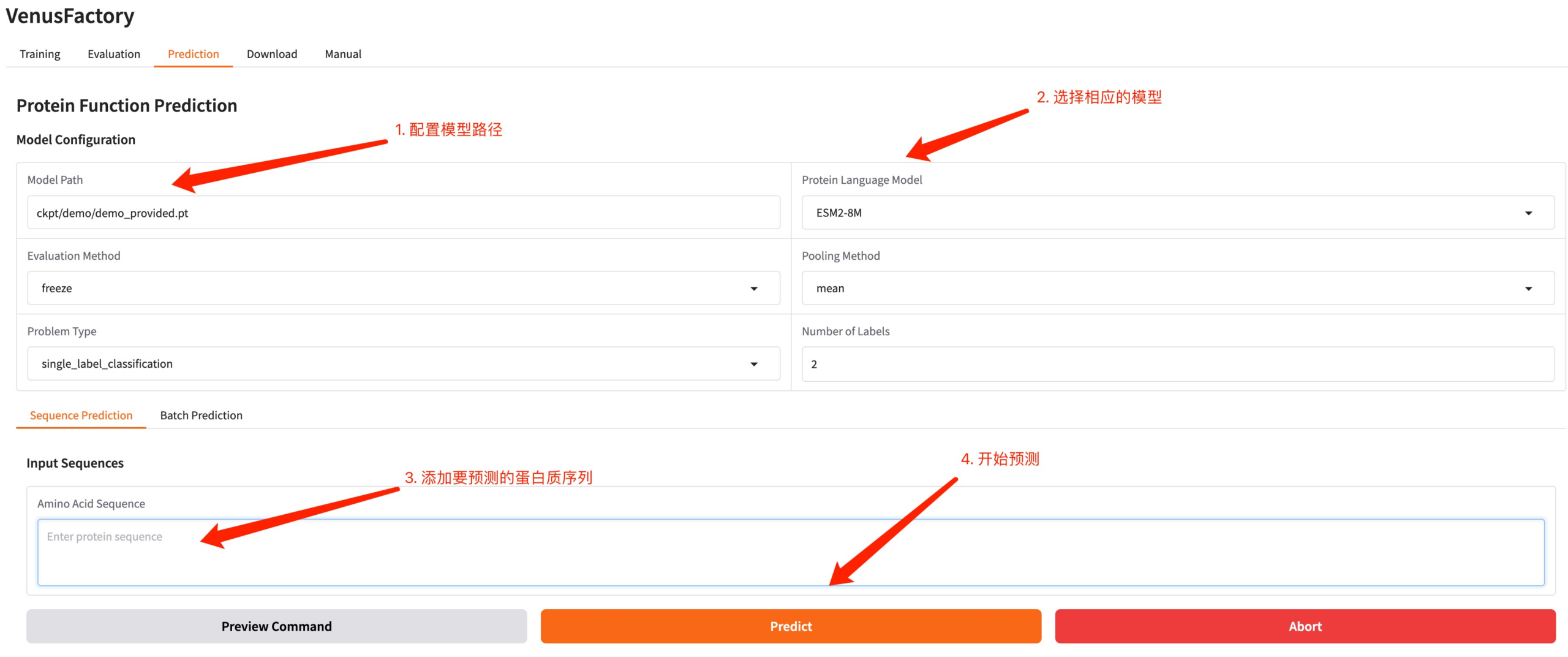
4. Module de prédiction
Cliquez sur le module Prédire, configurez le chemin du modèle généré par la formation et le modèle formé, entrez la séquence protéique que vous souhaitez prédire et cliquez sur Prédire pour effectuer une prédiction.
Exemple de séquence protéique : MKTWFGHVLQ
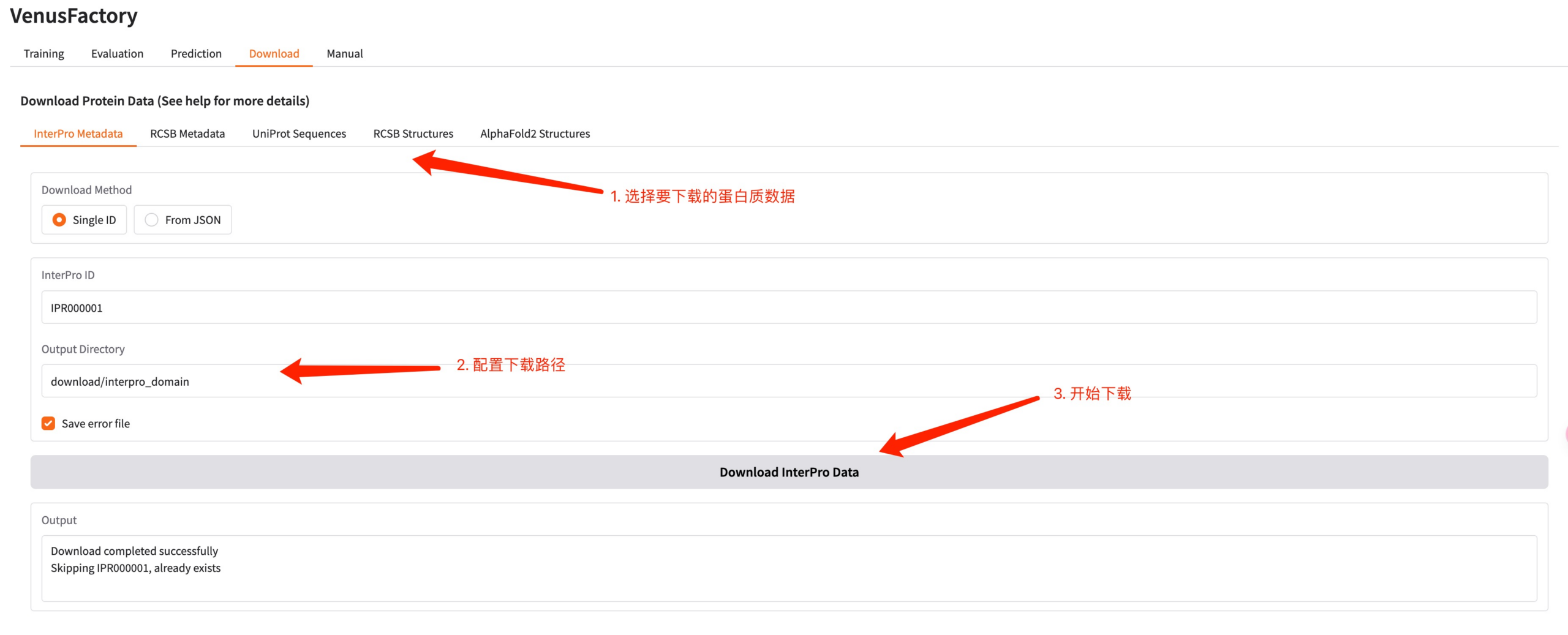
5. Télécharger le module
Cliquez sur le module Télécharger pour télécharger les données sur les protéines dans cette interface.
Ce qui précède est un tutoriel détaillé sur la façon d'utiliser la « VenusFactory Protein Engineering Design Platform ». Tout le monde est le bienvenu pour venir en faire l'expérience !








