Command Palette
Search for a command to run...
Nouvelles Réalisations De l'équipe David Baker ! La Diffusion RF Évolue Pour Obtenir Une Conception De Novo d'anticorps De Précision Au Niveau Atomique
À la fin du XIXe siècle, le bactériologiste allemand Emil Adolf von Behring a mené des recherches approfondies sur la toxine diphtérique. À cette époque, la diphtérie était comme la faux du dieu de la mort, prenant impitoyablement la vie de nombreux enfants. Dans l’expérience, Behring a injecté à des lapins des traces de toxine diphtérique, dans l’espoir d’observer leurs réactions et de trouver un moyen de combattre la toxine. Quelques jours plus tard, il fut surpris de constater que les lapins non seulement ne mouraient pas, mais développaient également une résistance aux injections ultérieures de toxine diphtérique. Des recherches plus poussées ont révélé qu’une substance capable de neutraliser la toxine diphtérique était présente dans le sérum de lapin.C’était la première fois que l’homme découvrait l’existence d’anticorps.Cette découverte accidentelle a ouvert la voie à l’application de la recherche sur les anticorps et a démontré le grand potentiel de l’utilisation de la propre puissance du corps pour combattre la maladie.
Après plus d’un siècle d’exploration approfondie, les scientifiques ont continué à faire des efforts dans le domaine de la recherche sur les anticorps, mais n’ont pas encore développé une voie efficace qui repose entièrement sur la conception informatique pour générer de nouveaux anticorps contre des épitopes spécifiques. Aujourd'hui,La découverte d’anticorps thérapeutiques repose encore principalement sur deux méthodes traditionnelles : l’immunisation animale ou le criblage aléatoire de bibliothèques.Face à des défis de plus en plus complexes en matière de maladies, les limites de ces méthodes traditionnelles sont devenues de plus en plus importantes et de nouvelles avancées sont nécessaires de toute urgence pour parvenir à une conception plus efficace et plus précise d’anticorps ciblant des cibles spécifiques, apportant ainsi un nouvel espoir à la recherche sur les anticorps.
Pour résoudre le problème de la découverte d'anticorps en s'appuyant sur des méthodes traditionnelles, l'équipe de David Baker, professeur de biochimie à l'Université de Washington, et ses collaborateurs ont combiné la conception informatique de protéines par réseau de diffusion RF affiné avec le criblage d'affichage de levure.Nous avons généré avec succès des VHH à chaîne lourde variable d'anticorps et des scFv à fragments variables à chaîne unique qui peuvent se lier à des épitopes spécifiques avec une précision au niveau atomique.La faisabilité de la conception de novo de domaines d’anticorps a été démontrée. Cette approche fournit un cadre rationnel pour la conception informatique, le criblage, l’isolement et la caractérisation d’anticorps conçus de novo, atteignant une précision au niveau atomique dans la structure et le ciblage des épitopes.
Les résultats de recherche pertinents ont été publiés sous forme de pré-impression sur bioRxiv sous le titre « Conception de novo atomiquement précise d'anticorps avec diffusion RF ».

Adresse du document :
https://doi.org/10.1101/2024.03.14.585103
Le projet open source « awesome-ai4s » rassemble plus de 200 interprétations d'articles AI4S et fournit des ensembles de données et des outils massifs :
https://github.com/hyperai/awesome-ai4s
Innovation technologique dans la recherche sur les anticorps : avancées et défis de l'IA
En tant que catégorie dominante de médicaments thérapeutiques protéiques, les anticorps sont devenus la principale méthode de traitement dans les domaines des tumeurs, des maladies auto-immunes, etc. en raison de leur haute spécificité et de leurs faibles effets secondaires. En 2025, plus de 160 médicaments à base d'anticorps ont été approuvés par les agences de réglementation du monde entier, et la taille de leur marché a augmenté à un rythme annuel moyen de 15% au cours des 10 dernières années.On s’attend à ce que ce montant atteigne 445 milliards de dollars dans les cinq prochaines années.
Cependant,La technologie traditionnelle de développement d’anticorps repose depuis longtemps sur l’immunisation animale et le criblage aléatoire de bibliothèques.Confronté à des goulots d’étranglement importants. L’immunisation animale nécessite plusieurs injections d’antigènes pour stimuler le système immunitaire de l’animal afin qu’il produise des anticorps.Le processus prend généralement de 6 à 12 mois.De plus, en raison des différences dans les réponses immunitaires individuelles des animaux, il est difficile d’obtenir des anticorps hautement efficaces contre des antigènes complexes tels que les protéines membranaires. Bien que les méthodes de criblage de bibliothèques aléatoires (telles que la technologie d'affichage de phages) puissent élargir la gamme de criblage, elles sont difficiles à stimuler les réponses immunitaires aux antigènes, tels que les protéines dépliées ou les épitopes glycosylés, et l'affinité des anticorps criblés est généralement faible.
Afin de dépasser les limites de la technologie traditionnelle, la conception informatique et l’intelligence artificielle sont progressivement devenues de nouvelles directions dans la recherche et le développement d’anticorps. Les premières études ont réussi à améliorer les performances des anticorps en intégrant des résidus dans des structures d’anticorps existantes, en optimisant la conformation de la région déterminante complémentaire (boucle CDR) et en transformant l’interface d’interaction à l’aide de l’algorithme Rosetta. Par exemple,Une étude de 2018 a augmenté l’affinité des anticorps anti-PD-1 de 20 fois grâce à la conception informatique.
Ces dernières années, la technologie d’apprentissage profond a encore favorisé la génération de séquences d’anticorps. En 2023, une équipe de l’Université de Stanford a utilisé des réseaux neuronaux pour concevoir des anticorps neutralisants à large spectre contre les nouveaux variants du coronavirus, et leur activité in vitro était trois fois supérieure à celle des méthodes traditionnelles. En 2024,Le modèle de diffusion RF développé par l'équipe de David Baker permet la conception de novo de protéines de liaison sans avoir besoin d'une structure de squelette prédéfinie.Cette technologie simule le processus physique de repliement des protéines pour générer une interface de liaison hautement complémentaire à la forme de l'épitope cible, et a permis de concevoir avec succès de nouveaux inhibiteurs contre l'hémagglutinine du virus de la grippe. Cependant, cette étude n'est applicable qu'aux épitopes avec des structures secondaires régulières, telles que les hélices α et les replis β, tandis que les anticorps s'appuient généralement sur des structures cycliques complexes (telles que CDR-H3) pour réaliser la liaison, ce qui rend son application directe dans la conception d'anticorps encore difficile.
La conception d'anticorps structurellement précis à partir de zéro, c'est-à-dire sans homologie avec des anticorps connus, reste un problème non résolu, avec des défis majeurs tels que des simulations conformationnelles dynamiques insuffisantes, un manque de données de haute qualité et de longs cycles de vérification expérimentale.
Le processus de liaison des anticorps implique des changements conformationnels dans les boucles CDR flexibles, et les algorithmes existants ont du mal à simuler avec précision de telles interactions dynamiques ; dans le même temps, la rareté des données structurelles sur les complexes anticorps-antigène restreint la capacité de généralisation des modèles d’apprentissage profond. Bien que la conception informatique puisse réduire considérablement le temps initial de R&D, la purification de l'expression et les tests d'activité nécessitent encore plusieurs semaines, ce qui constitue un goulot d'étranglement dans la boucle fermée de la technologie.Les avancées futures pourraient s’appuyer sur le développement d’algorithmes hybrides pour intégrer des modèles physiques à des outils d’IA génératifs.Les données de séquençage de cellules uniques et de microscopie cryoélectronique sont intégrées sur la base de la construction d'une base de données à plusieurs échelles, et l'itération en temps réel est réalisée grâce à l'automatisation robotique basée sur le modèle de R&D « en boucle fermée sèche et humide ».
De l’immunisation animale à la conception informatique, l’innovation de la technologie des anticorps constitue non seulement un changement de paradigme dans le domaine de la biomédecine, mais reflète également le potentiel du transfrontalier multidisciplinaire. Avec le développement de l’IA et de la biologie synthétique, la vision de concevoir des anticorps entièrement à partir de zéro peut progressivement se concrétiser, ouvrant un nouveau chapitre pour la médecine de précision.
Il convient de noter que le domaine de la conception informatique d’anticorps a récemment réalisé des avancées clés. En mars 2025, l'équipe de David Baker a développé un nouvel outil de génération de protéines IA appelé RFdiffusion (appelé : nouveau RFdiffusion).Une nouvelle version du modèle spécifiquement optimisée pour les régions variables des anticorps telles que les boucles CDR a été publiée.L'équipe avait réussi à générer des fragments d'anticorps fonctionnels à chaîne courte tels que des nanocorps dans une étude en mars 2024, mais en raison de la complexité de la structure de l'anticorps, la version de l'époque (appelée : diffusion RF ordinaire) présentait encore des limites dans la conception de structures d'anticorps plus complexes.
Après plus d'un an d'itération d'algorithme, le nouveau RFdiffusion est désormais capable de générer un fragment variable à chaîne unique scFv plus complet, plus proche des anticorps humains naturels, en introduisant une formation de base de données de structures complexes anticorps-antigène. Cette avancée marque leL’IA a permis de concevoir de manière coordonnée des chaînes lourdes et légères d’anticorps avec des domaines de liaison à l’antigène complets sans avoir besoin de modèles.Apporte un nouvel espoir à la conception d’anticorps.
Nouvelle diffusion RF : conception d'anticorps de novo avec une précision au niveau atomique
Pour adapter la diffusion RF à la conception des anticorps, l'équipe de recherche a effectué quelques ajustements précis sur cette base. Comme le montre la figure ci-dessous, pendant le processus de formation, il utilise le cadre AlphaFold2/RF2 pour représenter l'épine dorsale de la protéine et ajoute du bruit au cadre de la protéine à travers une série de « pas de temps (T) » jusqu'à ce que le cadre devienne méconnaissable. À chaque pas de temps, RFdiffusion prédit la structure débruitée, optimisée en minimisant l'erreur quadratique moyenne mse entre la vraie structure X₀ et la structure prédite pX₀. Après cette méthode de formation unique, La diffusion RF est capable de générer progressivement de nouvelles structures protéiques à partir de distributions aléatoires de résidus au moment de l'inférence.
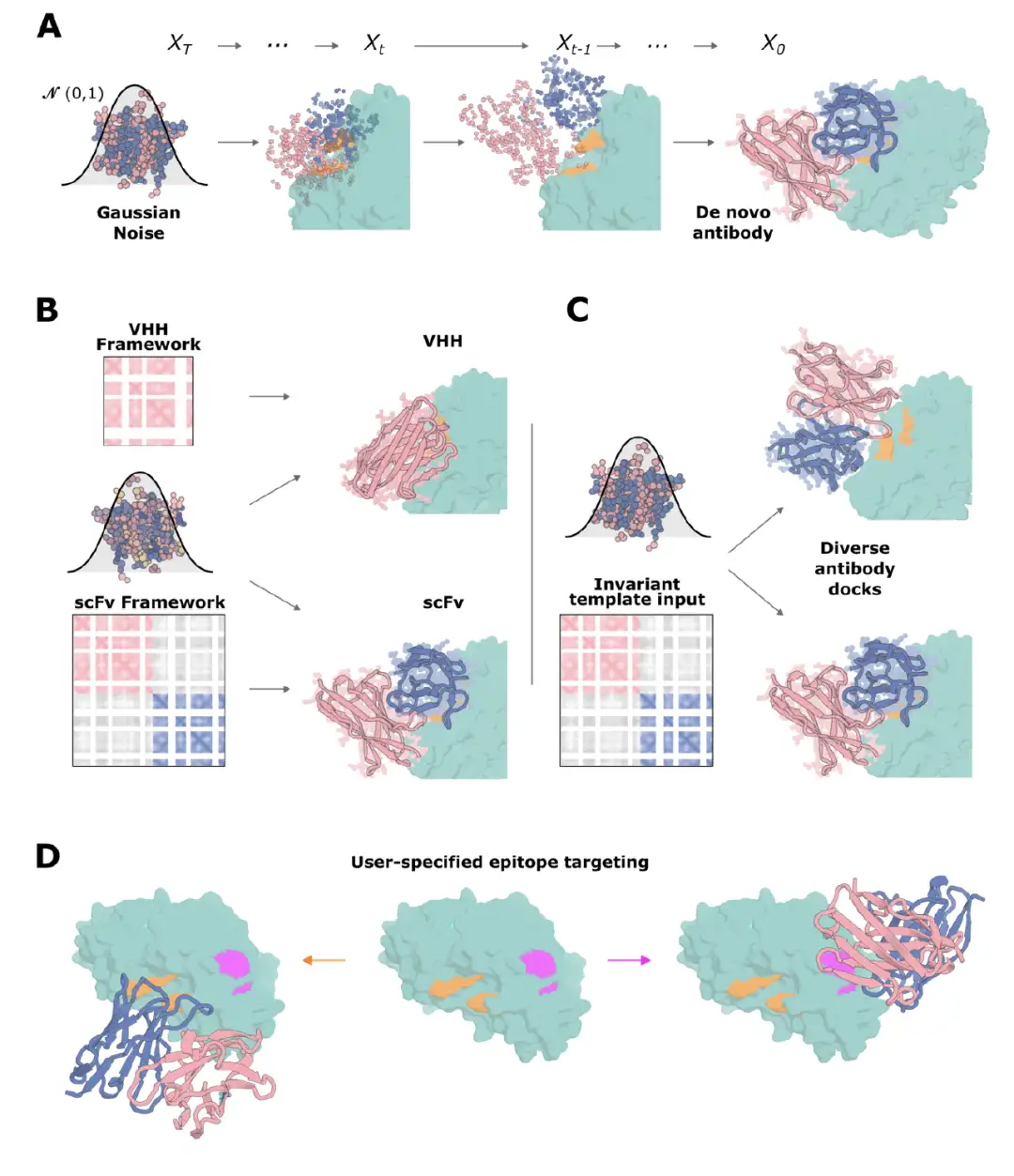
Par la suite, l’étude a appliqué la nouvelle RFdiffusion pour concevoir des VHH d’anticorps à domaine unique. Les VHH sont conçus sur la base des domaines variables des anticorps à chaîne lourde de camélidés, et leur taille plus petite rend les gènes codant les conceptions plus faciles et moins coûteux à assembler que les fragments variables à chaîne unique scFv ou les fragments de liaison à l'antigène Fab.
Bien que les VHH ne possèdent que trois boucles CDR, soit moins que les six des anticorps traditionnels, la surface d'interaction moyenne des VHH est très similaire à celle des anticorps traditionnels.Cela suggère que l’approche de conception des VHH est également applicable à la conception des anticorps.
Pour concevoir des VHH, les chercheurs ont sélectionné un cadre VHH chimérique largement utilisé comme base, ciblant une série de cibles liées à la maladie, notamment la toxine B de Clostridium difficile (TcdB), l'hémagglutinine HA de la grippe H1, etc., et ont réalisé une conception de séquence de boucle CDR dans le contexte cible via ProteinMPNN, puis ont été criblés à l'aide du réseau RoseTTAFold2 affiné. Dans la conception de l'HA de la grippe, afin de rendre les conditions de conception expérimentale cohérentes avec les paramètres de calcul, les chercheurs ont utilisé des produits HA monomères produits commercialement et exprimés dans des cellules d'insectes pour des mesures d'affinité.
Les résultats montrent queLa diffusion RF permet la conception de VHH qui interagissent spécifiquement avec les épitopes cibles.Les liants à plus forte affinité pour le site III du VRS, l'HA de la grippe, le RBD et le TcdB sont respectivement présentés dans la figure ci-dessous. Les boucles CDR sont significativement différentes de celles des VHH naturels, ce qui indique que la conception dépassait le cadre de l'ensemble de données de formation. Pour TcdB, l'épitope cible est l'interface Frizzled-7 et il n'y a pas d'anticorps ou de VHH ciblant ce site dans le PDB. De plus, le TcdB VHH a neutralisé la toxicité du TcdB dans les cellules CSPG4 knockout.
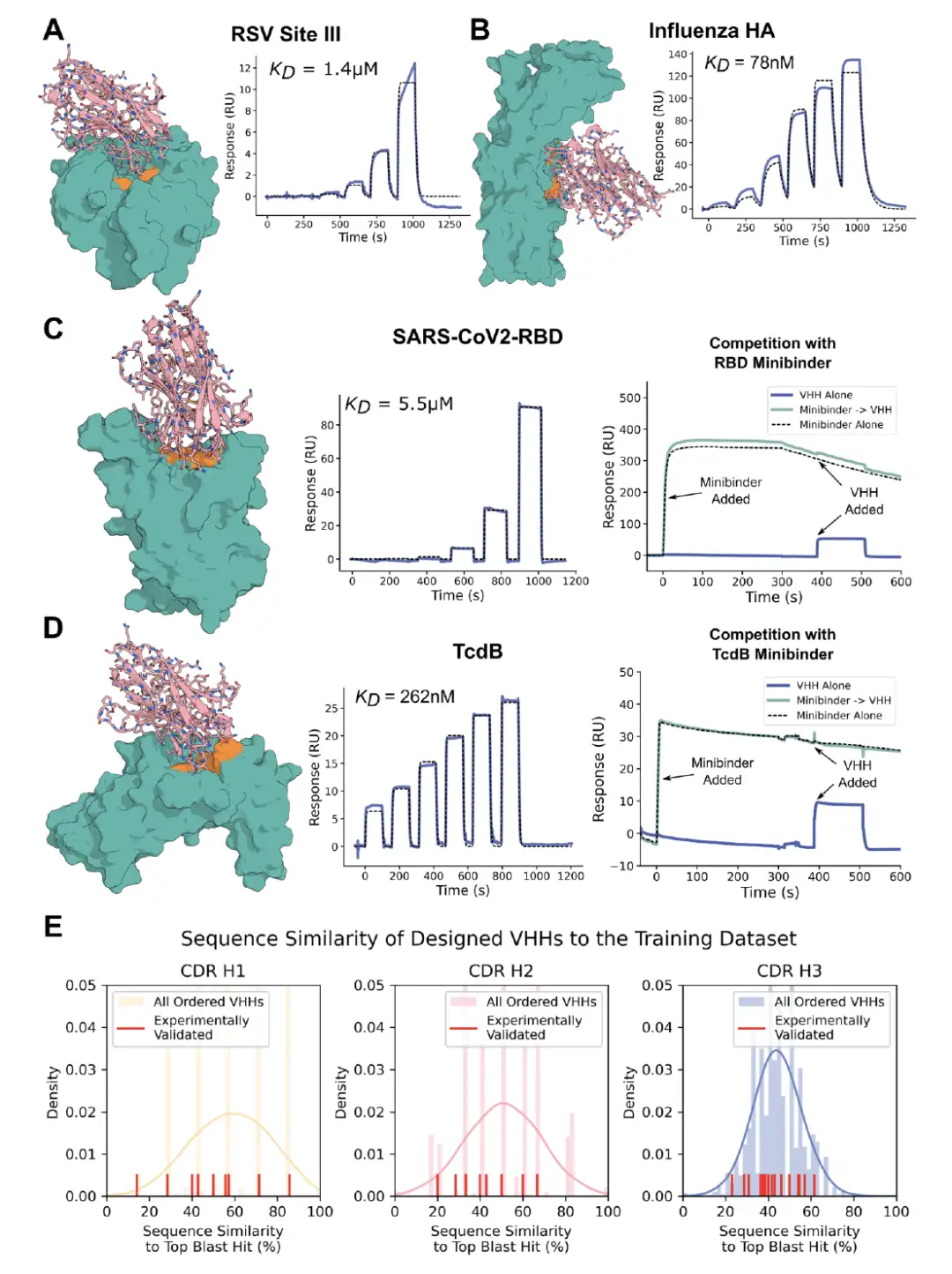
* Figure AC : Résultats des tests de la conception VHH ciblant le locus RSV III, l'HA de la grippe, le RBD et le TcdB
* Figure E : Différence entre la conception VHH et l'ensemble de données d'entraînement
Les chercheurs ont également exploré la capacité de la diffusion RF à concevoir des chaînes lourdes et légères dans un format scFv à fragment variable à chaîne unique. Contrairement aux VHH, la conception du scFv est plus compliquée et nécessite la construction des six CDR et de leurs modèles d'amarrage sur les chaînes lourdes et légères. Cependant, la synthèse génétique du scFv est confrontée à des défis. D’une part, la séquence scFv est longue et difficile à assembler à l’aide de paires d’oligonucléotides conventionnelles. D’autre part, les scFv présentent une forte homologie de séquence, ce qui rend l’appariement spécifique difficile.
À cette fin, les chercheurs ont développé un protocole d’assemblage par étapes pour parvenir à la construction de chaînes lourdes et légères.Ils peuvent être associés spécifiquement par modèle de conception ou combinés et mélangés dans des sous-ensembles de conceptions avec des modes de liaison ciblés similaires.Les résultats expérimentaux ont montré que les nouveaux scFv générés en combinant des paires de chaînes lourdes et légères de conceptions différentes étaient capables de se lier à l'épitope cible avec une fréquence similaire à celle de la conception originale. De plus, dans les ensembles conçus avec la même orientation de liaison, les CDR des chaînes lourdes et légères interagissent avec différentes régions et peuvent être combinés sans perte de précision structurelle, alors que l'appariement aléatoire produit rarement des liants prédits.
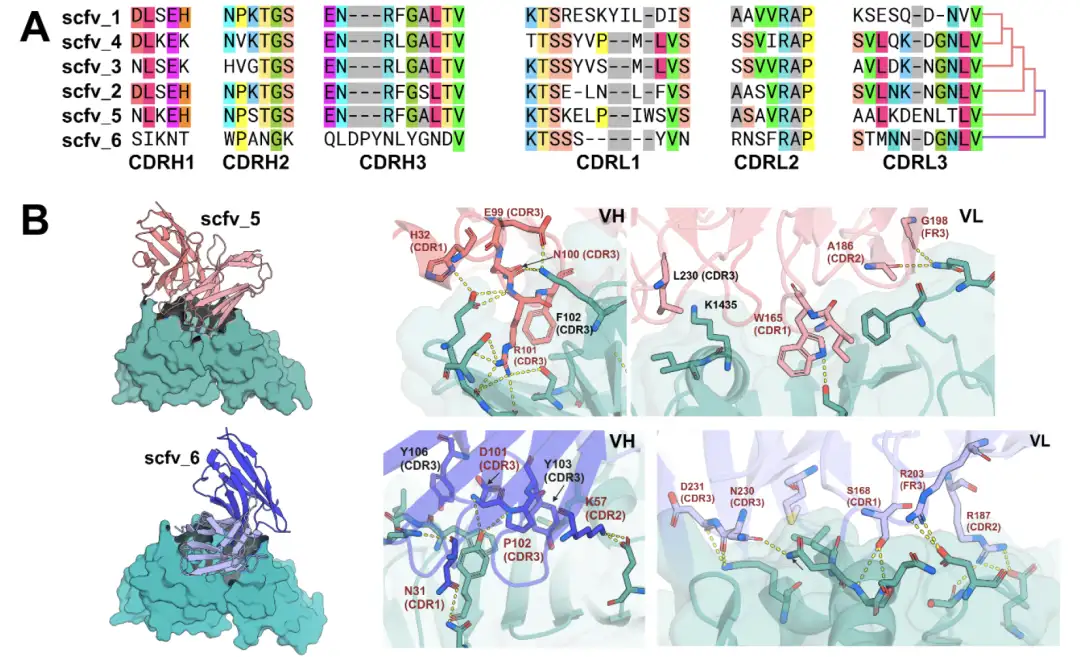
* Figure A : Alignement de séquences multiples de 6 scFv se liant à TcdB
* Figure B : Structure prédite par AlphaFold3 de scFv5 et scFv6 en complexe avec le domaine de liaison au récepteur TcdB
David Baker : L'évolution des anticorps et de la diffusion RF
Cette étude n’est en fait que la pointe de l’iceberg des résultats de recherche de David Baker. En fait, à la pointe de la biologie computationnelle, l’équipe de David Baker a réalisé une série de percées allant du ciblage des virus au traitement des maladies grâce à la conception de protéines basée sur l’intelligence artificielle.

Parmi eux, le modèle de diffusion RF est devenu une base importante pour changer le paradigme du développement de médicaments à base d’anticorps. En 2021, l'équipe de David Baker a développé RosettaFold basé sur le framework AlphaFold2.Ses capacités principales se limitent à la prédiction des structures tridimensionnelles de protéines connues.
* Adresse open source de RoseTTAFold :
https://github.com/RosettaCommons/RoseTTAFold
L’équipe de recherche a rapidement compris que la véritable révolution réside dans la « génération » plutôt que dans la « reproduction ». Ils ont donc combiné le modèle de diffusion et l’algorithme de repliement des protéines et ont lancé la première génération de RFdiffusion en 2023. Ce modèle revient à obtenir la clé de l’ingénierie inverse : la méthode traditionnelle nécessite de déduire la structure à partir de la séquence d’acides aminés.La diffusion RF peut générer de manière inverse un nouveau squelette protéique en fonction des exigences fonctionnelles cibles.Lors des premiers tests, il a réussi à concevoir un nanocorps qui se lie à l'hémagglutinine de la grippe, mais la région de la boucle CDR de l'anticorps qu'il a généré présentait encore des écarts conformationnels, et la microscopie cryoélectronique a montré un écart quadratique moyen de 1,2 Å dans l'interface de liaison.
* Adresse du papier :
https://www.science.org/doi/10.1126/science.abj8754
Cette limitation a conduit à l’introduction d’une mise à niveau clé en 2024 : un système de retenue dynamique.L'équipe de recherche a ajouté au modèle des contraintes de paramètres physiques et chimiques du site de liaison antigène-anticorps, de sorte que le processus de génération prenne non seulement en compte la stabilité structurelle, mais simule également les interactions dynamiques entre les molécules.
La diffusion RF améliorée a donné de très bons résultats dans la conception du nouvel anticorps à base de protéine de pointe du coronavirus : la structure annulaire flexible qu'elle a construite a verrouillé avec précision les épitopes conservés à la surface du virus, ce qui a été vérifié par des expériences.Son affinité de liaison atteint 0,8 nM, ce qui est 15 fois plus fort que les anticorps naturels.Ce qui est encore plus remarquable, c'est que ce modèle commence à défier des cibles « non médicamenteuses » : l'anticorps miniature conçu pour le récepteur IL-23 ne contient que 58 acides aminés, mais peut rester actif à une température élevée de 80°C et dans un environnement de pepsine, réalisant pour la première fois l'administration orale d'anticorps.
* Adresse du papier :
https://www.biorxiv.org/content/10.1101/2024.03.14.585103v2
En 2025, RFdiffusion entre dans la phase de fusion multimodale. L'équipe a intégré des données de séquençage de cellules uniques et des bibliothèques de structures de cryomicroscopie électronique, permettant au modèle de personnaliser directement les anticorps personnalisés en fonction des caractéristiques du répertoire immunitaire du patient. Dans le dernier cas, un patient atteint d’un cancer du poumon résistant aux médicaments s’est vu injecter ses néoantigènes tumoraux dans le système.La diffusion RF a généré 12 anticorps candidats en 36 heures, dont trois ont montré des effets significatifs de destruction des tumeurs dans les modèles organoïdes.Le modèle ne se limite plus à la conception d'anticorps : il explore la synthèse d'éléments protéiques inter-espèces, comme la fusion des canaux ioniques mécanosensibles des bactéries de barytoscopie des grands fonds avec des anticorps humains pour créer des médicaments intelligents capables de détecter les changements de pH du microenvironnement tumoral.
* Adresse du papier :
https://www.nature.com/articles/s41586-024-08393-x
On peut constater que RFdiffusion se transforme d’une « imprimante 3D de protéines » en un « architecte des fonctions vitales », redéfinissant les limites de la biologie synthétique. Ce qui est encore plus intéressant, c’est que cette évolution est loin d’être terminée et que les innovations dans la technologie des anticorps poussent le domaine biomédical vers de nouveaux sommets.








