Command Palette
Search for a command to run...
Oxford/Amazon/Westlake University/Tencent Et d'autres Ont Proposé Un Modèle Médical Multimodal, Multidomaine Et Multilingue M³FM, Qui Peut Être Utilisé Pour Le Diagnostic Clinique À Échantillon Zéro

Je crois que de nombreux amis qui aiment les films Marvel ont été émerveillés par cette scène. Dans le film « Iron Man 2 », le majordome de l'intelligence artificielle Jarvis a collecté des échantillons de sang de Stark et a utilisé des algorithmes d'apprentissage profond pour modulariser rapidement les données de l'échantillon. Il a analysé avec précision et rapidité la teneur en palladium du corps de Stark, et a même donné des suggestions inter-domaines lors de la publication d'un rapport, telles que « les éléments existants ne peuvent pas remplacer le palladium métallique, et de nouveaux éléments doivent être synthétisés ».Bien qu’il ne s’agisse que de quelques dizaines de secondes de séquences, elles démontrent parfaitement l’automatisation, l’intelligence et les fonctionnalités basées sur les processus des soins de santé intelligents.
Cependant, dans la vie réelle, pour obtenir le même résultat, le personnel médical doit passer par des processus complexes tels que des prélèvements et des tests sanguins, des analyses d’images, des comparaisons de données, la publication de rapports et la classification des maladies. Et ce n’est que d’un point de vue macro. Si nous le décomposons, la situation sera encore pire. Prenons l’exemple des images médicales, le type le plus courant dans le diagnostic clinique. Les images médicales peuvent décrire les résultats cliniques et fournir une base pour un diagnostic plus approfondi de la maladie. Cependant, lorsqu'il s'agit de décrire avec précision, concision, intégralité et cohérence un rapport sur des images médicales en langage naturel, de nombreux personnels médicaux trouvent cela fastidieux et ennuyeux.Les statistiques montrent que même pour les médecins expérimentés, il faut généralement en moyenne 5 minutes ou plus pour rédiger un rapport.
Heureusement, même si la science-fiction n’a pas encore complètement éclairé la réalité, une lueur d’espoir est déjà apparue à travers les fissures de l’obscurité. À l’intersection de l’intelligence artificielle et de la santé médicale, de plus en plus de chercheurs scientifiques ont mené des recherches approfondies et développé des méthodes de génération automatique de rapports. Ces méthodes génèrent automatiquement des brouillons de rapports que le personnel médical peut examiner, modifier et consulter. D’une part, ils peuvent résoudre efficacement les tâches chronophages et exigeantes en main-d’œuvre du personnel médical et, d’autre part, ils peuvent réduire la probabilité d’erreurs humaines grâce à l’automatisation.
Récemment, npj Digital Medicine, une revue de la revue universitaire de renommée internationale Nature Portfolio, a publié une étude intitulée « Un modèle de fondation médicale multimodal, multidomaine et multilingue pour un diagnostic clinique zéro coup ».Il mentionne un modèle de fondation médicale multimodal (image et texte), multidomaine (CT et CXR) et multilingue (chinois et anglais) M³FM (Multimodal Multidomain Multilingual Foundation Model), qui peut être utilisé pour le diagnostic clinique à échantillon zéro et soutenir la déclaration et la classification des maladies.Les chercheurs ont démontré l’efficacité de cette méthode sur neuf ensembles de données de référence pour deux maladies infectieuses et 14 maladies non transmissibles, surpassant les méthodes précédentes.
L’étude dispose d’une luxueuse liste d’auteurs. Outre les équipes de l'Université d'Oxford, de l'Université de Rochester, d'Amazon et d'autres institutions, il comprend également le Dr Zheng Yefeng du Laboratoire d'intelligence artificielle médicale de l'Université Westlake et le Dr Wu Xian, directeur du Centre de recherche Tianyan du Tencent Youtu Lab.

Adresse du document :
https://www.nature.com/articles/s41746-024-01339-7
Le projet open source « awesome-ai4s » rassemble plus de 200 interprétations d'articles AI4S et fournit des ensembles de données et des outils massifs :
https://github.com/hyperai/awesome-ai4s
La perte de données reste un problème pour les méthodes existantes
L'imagerie médicale est la base des rapports d'imagerie médicale et de la classification des maladies, et joue un rôle important dans le diagnostic clinique ultérieur. Par conséquent, la recherche sur les méthodes d’automatisation associées est naturellement devenue l’un des axes de recherche dans le domaine de la recherche scientifique. Cependant, malgré les résultats fructueux des recherches, de nombreuses lacunes subsistent encore d’un point de vue pratique.Parmi eux, la rareté, voire l’absence totale de données, constitue un défi majeur.
d'une part,La génération de rapports sur les maladies est similaire à la tâche de génération de langage basée sur l'image, où l'objectif est de générer un texte descriptif pour décrire l'image d'entrée. Les méthodes de base traditionnelles s’appuient souvent largement sur de grandes quantités de données de formation médicale de haute qualité annotées par des cliniciens, dont la collecte est coûteuse et prend du temps, en particulier pour les maladies rares et les langues autres que l’anglais.
En particulier, pour les maladies nouvelles ou rares, ces maladies manquent généralement de données efficaces suffisantes pour une formation aux premiers stades. Par exemple, la nouvelle pneumonie à coronavirus qui a commencé à faire rage dans le monde fin 2019 disposait de données limitées qui pouvaient être collectées à ses débuts, ce qui a fait que le temps de formation du système a largement dépassé la durée des premières vagues de l’épidémie. Selon le « Rapport d'observation des tendances de l'industrie des maladies rares en Chine 2024 », il existe plus de 7 000 maladies rares connues dans le monde. Des données probantes conservatrices estiment que la prévalence des maladies rares dans la population est d’environ 3,5% à 5,9%, et que le nombre de personnes touchées par des maladies rares dans le monde est d’environ 260 millions à 450 millions. Une maladie aussi vaste et atypique rend sans aucun doute les problèmes mentionnés ci-dessus plus difficiles à résoudre.
De plus, le système de santé mondial implique différentes régions, différentes populations et différentes langues. Pour les langues autres que l’anglais, les données étiquetées pertinentes sont généralement très rares, voire totalement absentes. Par conséquent, les données étiquetées limitées constituent sans aucun doute un défi majeur pour les systèmes de formation en langues non anglaises utilisant les méthodes existantes. Dans le même temps, cela rend également les méthodes existantes plus difficiles à traiter avec des langues peu courantes, ce qui affecte davantage l’objectif d’équité de l’IA et ne parvient pas à bénéficier pleinement aux groupes sous-représentés.
d'autre part,Afin de classer efficacement les maladies, les modèles avancés actuels s'inspirent principalement du succès de CLIP, tels que BioViL, REFERS, MedKLIP et MRM, tous développés pour mieux comprendre les données médicales multimodales. Lors de leur mise en œuvre, ces méthodes exploitent l'apprentissage contrastif et pré-entraînent les modèles CLIP à l'aide de données médicales, mais comme la plupart des modèles sont spécifiques à la radiographie thoracique (CXR), ils ne parviennent généralement pas à gérer les images et textes médicaux multi-domaines et multilingues dans un cadre unique. Dans le même temps, les travaux précédents n’ont pas non plus réussi à réaliser des rapports de maladie zéro coup dans différents domaines du langage et des images.
* Le modèle CLIP est un modèle de pré-formation d'image de langage contrastif développé par OpenAI - une méthode efficace pour apprendre à partir de la supervision du langage naturel. CLIP apprend principalement l'association entre les images et le texte grâce à l'apprentissage contrastif et s'entraîne au préalable sur des paires image-texte à grande échelle, afin que le modèle puisse comprendre et associer des informations provenant de différentes modalités.
Dans ce contexte, il est urgent de développer un modèle capable de réaliser un diagnostic clinique multimodal, multi-domaines et multilingue avec peu ou pas d’échantillons.Les innovations spécifiques proposées dans cette étude sont les suivantes :
* Le M³FM proposé est la première tentative de réalisation d'un diagnostic clinique multimodal, multidomaine et multilingue à zéro coup, où les données étiquetées pour la formation sont rares, voire totalement absentes ;
* M³FM valide son efficacité sur 9 jeux de données, dont deux domaines de données d'imagerie médicale, à savoir CXR et CT ; deux langues différentes, à savoir le chinois et l'anglais ; deux tâches de diagnostic clinique, à savoir la déclaration des maladies et la classification des maladies ; et de multiples maladies, dont 2 maladies infectieuses et 14 maladies non infectieuses.
M³FM : Deux modules majeurs, vérifiés par plusieurs ensembles de données
Dans cette étude, l'idée clé du M³FM proposé est de pré-entraîner le modèle sur des données médicales publiques à travers des modalités, des domaines et des langues afin d'acquérir des connaissances approfondies, puis d'exploiter ces connaissances pour accomplir des tâches en aval sans avoir besoin de données étiquetées. Les principaux composants du framework M³FM comprennent 2 modules principaux,C'est-à-dire MultiMedCLIP et MultiMedLM. Comme le montre la figure suivante :
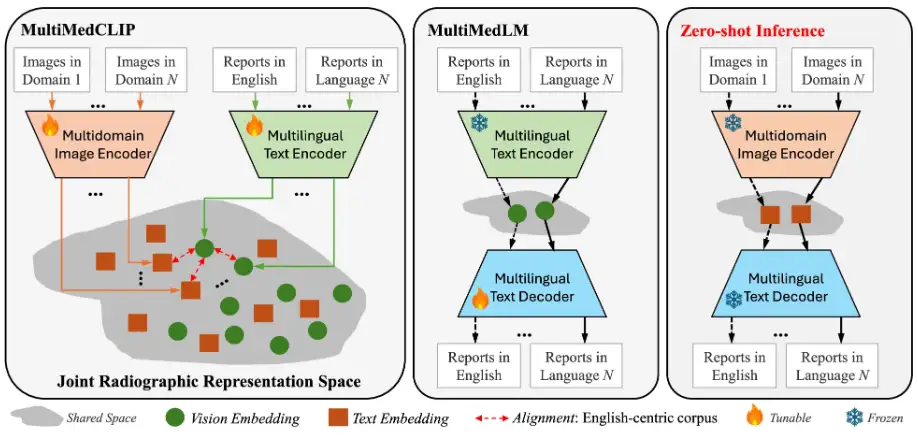
Le processus est que MultiMedCLIP aligne et relie différentes langues et images dans un espace latent commun partagé.Ensuite, MultiMedLM reconstruit le texte en fonction de la représentation du texte dans l'espace latent partagé, et enfin M³FM génère des rapports multilingues directement en fonction de la représentation visuelle des images d'entrée provenant de différents domaines dans l'espace latent unifié.
Plus précisément, MultiMedCLIP est un module d'apprentissage des représentations conjointes, qui introduit un encodeur visuel multi-domaines et un encodeur de texte multilingue, dans le but de créer un espace latent partagé pour aligner les représentations visuelles et textuelles de différents domaines d'imagerie médicale et de différentes langues. Inspirés par la méthode d'apprentissage contrastif, les chercheurs ont utilisé la perte InfoNCE (Info Noise Contrastive Estimation) et la perte MSE (erreur quadratique moyenne) comme objectifs de formation pour maximiser la similarité entre les paires d'échantillons positifs et minimiser la similarité entre les paires d'échantillons négatifs, obtenant ainsi un alignement entre les représentations visuelles dans différents domaines et les représentations textuelles dans différentes langues, posant ainsi une base solide pour le raisonnement zéro-shot en aval.
MultiMedLM est un module de génération de rapports multilingues.Un décodeur de texte multilingue est introduit, qui vise à apprendre à générer le rapport médical final sur la base des représentations extraites par MultiMedCLIP. Cette partie est formée en reconstruisant le texte d'entrée, qui peut être du texte chinois ou du texte anglais, et utilise la perte de génération de langage naturel - perte XE (entropie croisée) comme cible de formation. Il convient de mentionner que l’introduction de la formation à la reconstruction peut être considérée comme une formation non supervisée, qui ne nécessite que des données en texte brut non étiquetées pour la formation. Par conséquent, il n’est pas nécessaire de former les données d’annotation des tâches sur les tâches en aval. De plus, afin de garantir la stabilité de la formation MultiMedLM, l'équipe de recherche a également introduit des pertes aléatoires et du bruit gaussien.
L'optimiseur AdamW a été utilisé dans l'expérience, avec le taux d'apprentissage fixé à 1e-4 et la taille du lot fixée à 32. Les expériences ont été menées sur PyTorch et V100 GPU, en utilisant un entraînement de précision mixte.
En termes d’ensembles de données,La pré-formation a été réalisée sur les ensembles de données MIMC-CXR et COVID-19-CT-CXR, où MIMC-CXR se compose de 377 110 images CXR et de 227 835 rapports de radiologie en anglais, le plus grand ensemble de données publié à ce jour ; COVID-19-CT-CXR comprend 1 000 images CT/CXR et les rapports anglais correspondants. De plus, les chercheurs ont extrait la moitié du corpus anglais des deux ensembles de données et ont utilisé Google Translator pour constituer une équipe de formation chinois-anglais. Les résultats ont montré que cette méthode peut améliorer les résultats de la traduction automatique de texte.
Au cours de la phase d'évaluation, les ensembles de données utilisés comprenaient IU-Xray, COVID-19 CT, COV-CTR, Shenzhen Tuberculosis Dataset, COVID-CXR, NIH ChestX-ray, CheXpert, RSNA Pneumonia et SIIM-ACR Emphysema, permettant une évaluation complète des performances du modèle.
* IU-Radiographie :7 470 images CXR et 3 955 rapports de radiologie en anglais ont été inclus. L'ensemble de données est divisé aléatoirement en 80% – 10% – 10% pour la formation, la validation et les tests.
* Test COVID-19 :Il contient 1 104 images CT et 368 rapports de radiologie chinois. De même, l’ensemble de données est divisé aléatoirement en 80% – 10% – 10% pour la formation, la validation et les tests.
*COV-CTR :Contient 726 images CT COVID-19, liées à des rapports en chinois et en anglais.
* Ensemble de données sur la tuberculose à Shenzhen :Contient 662 images CXR, et les ensembles de formation, de validation et de test sont divisés en 7:1:2.
* COVID-CXR :Contenant plus de 900 images CXR, l'ensemble de données est divisé aléatoirement en 80% – 10% – 10% pour la formation, la validation et les tests.
* Radiographie thoracique du NIH :Contient 112 120 images CXR, chaque image est étiquetée avec l'occurrence de 14 maladies courantes liées aux radiations, et le rapport entre les ensembles d'entraînement, de validation et de test est de 7:1:2.
* CheXpert :Contient plus de 220 000 images diagnostiques CXR. Après prétraitement, nous avons obtenu 218 414 images dans l’ensemble d’entraînement, 5 000 images dans l’ensemble de validation et 234 images dans l’ensemble de test.
* Pneumonie RSNA :Il se compose d'environ 30 000 images radiologiques, avec des ratios d'entraînement, de validation et de test de 85% - 5% - 10%.
* Emphysème SIIM-ACR :Il comprend 12 047 images CXR, avec un rapport entre les ensembles d'entraînement, de validation et de test de 70% – 15% – 15%.
Les expériences montrent que M³FM a des performances supérieures, surpassant les méthodes avancées précédentes.Comme le montre la figure ci-dessous. Comme le montrent les résultats de la déclaration des maladies, les méthodes précédentes ne sont pas en mesure de gérer la tâche de déclaration des maladies dans le cadre du zéro coup, tandis que M³FM est capable d'effectuer simultanément des rapports de maladies multilingues et multidomaines dans un seul cadre. Dans le cadre de quelques prises de vue, lorsqu'il est formé avec les données étiquetées en aval de 10%, M³FM obtient des résultats de pointe, surpassant même l'approche entièrement supervisée R2Gen sur la génération de rapports CT vers chinois par les scores CIDEr de 1,5% et ROUGE-L de 1,2%.Cela démontre que M³FM peut générer des rapports multilingues précis et valides même lorsque les données étiquetées sont rares, et sera donc particulièrement utile pour les maladies rares ou émergentes.
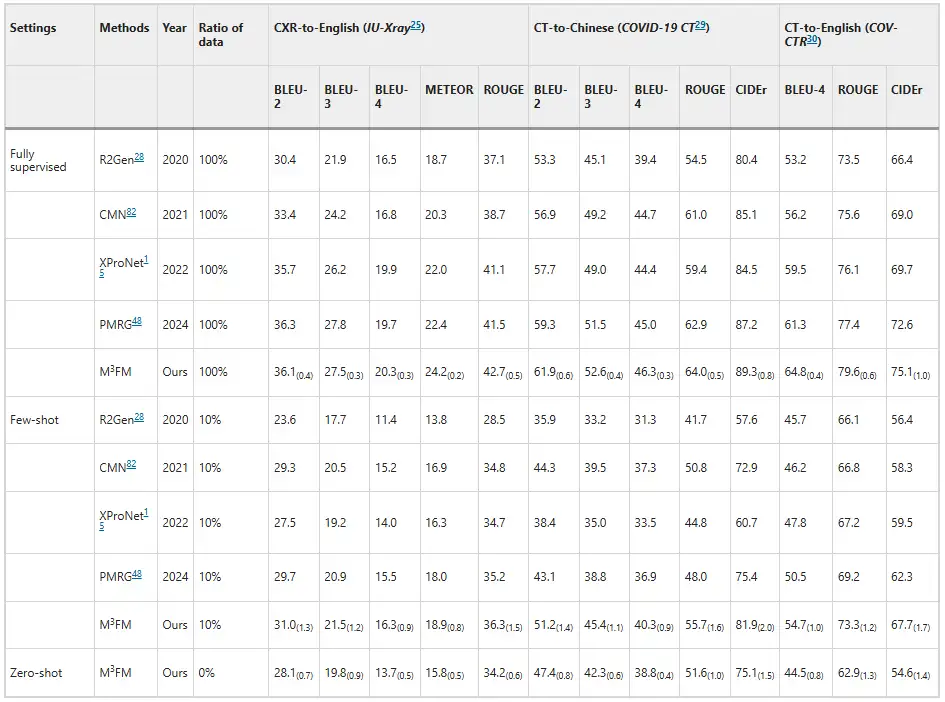
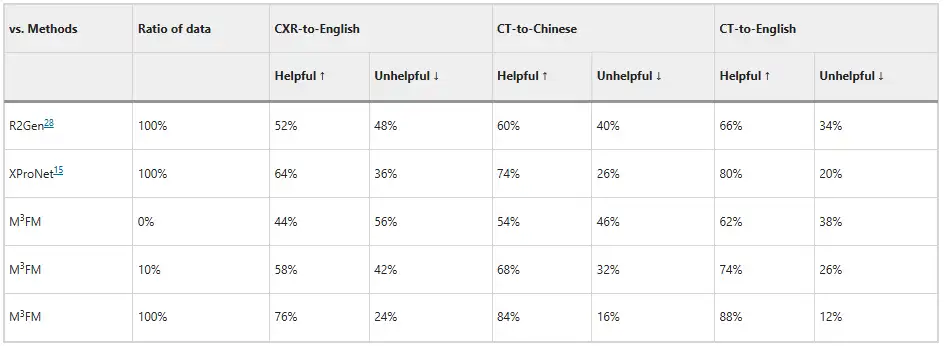
De plus, les chercheurs ont invité deux cliniciens à évaluer le modèle, et les résultats sont présentés dans la figure ci-dessous. Sans aucune formation de données étiquetées, M³FM peut générer des rapports multilingues et multi-domaines idéaux ; lorsque seulement 10% de données étiquetées sont utilisées pour la formation, M³FM peut être 6%, 8% et 8% plus élevé que la méthode entièrement supervisée R2Gen dans les tâches CXR vers l'anglais, CT vers le chinois et CT vers l'anglais respectivement ; en utilisant des données de formation complètes, M³FM peut améliorer R2Gen de plus de 20% dans trois tâches, et de 12%, 10% et 8% de plus que XProNet respectivement.Cela démontre le potentiel de M³FM pour libérer les cliniciens de la tâche fastidieuse et laborieuse de rédaction de rapports.
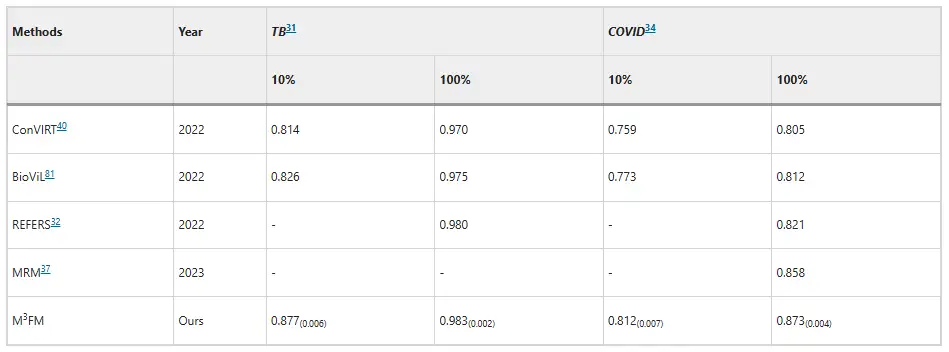
En termes de classification des maladies, M³FM a montré une supériorité dans le diagnostic des maladies infectieuses.Sur l'ensemble de données Shenzhen Tuberculosis et l'ensemble de données COVID – CXR, lors de l'utilisation de 10% de données de formation, les scores AUC de M³FM sont respectivement 5,1% et 3,9% supérieurs aux meilleurs résultats existants. Lorsque les données de formation ont été utilisées dans leur intégralité, M³FM a obtenu les meilleurs résultats dans deux maladies infectieuses. En termes de maladies non transmissibles, l'ensemble de données provient de la radiographie thoracique du NIH, et M³FM a obtenu des résultats comparables avec la méthode entièrement supervisée Model Genesis avec seulement 1% d'étiquettes de formation. Au 10%, M³FM a surpassé les méthodes de base MRM et REFERS dans le diagnostic de multiples maladies, ce qui a également confirmé l'efficacité et la capacité de généralisation de M³FM dans le diagnostic des maladies.
L'IA est à la pointe des soins de santé intelligents, et l'équipe de Zheng Yefeng prend les devants
Auparavant, de nombreux laboratoires se sont penchés sur cette question et les modèles qu’ils ont proposés présentent des orientations et des avantages différents.
Par exemple, pour la génération automatique de rapports, l'École des sciences et technologies de l'information de l'Université maritime de Dalian a publié un article de recherche intitulé « DACG : modèle de guidage à double attention et contexte pour la génération de rapports de radiologie » dans le forum professionnel Medical Image Analysis dans le domaine de l'analyse d'images médicales et biologiques. L'article propose un modèle de double attention et de guidage contextuel (DACG) pour la génération automatique de rapports de radiologie, qui peut atténuer le biais des données visuelles et textuelles et favoriser la génération de textes longs.
Adresse du document :
https://www.sciencedirect.com/science/article/abs/pii/S1361841524003025
Il existe également des modèles conçus pour plusieurs langues. Par exemple, l'équipe du professeur Wang Yanfeng et du professeur Xie Weidi de l'Université Jiao Tong de Shanghai a créé un corpus médical multilingue MMedC contenant 25,5 milliards de jetons, a développé une norme d'évaluation de questions-réponses médicales multilingues MMedBench couvrant 6 langues et a construit un modèle de base 8B MMed-Llama 3, qui a surpassé les modèles open source existants dans plusieurs tests de référence et est plus adapté aux scénarios d'application médicale. Les résultats de recherche pertinents ont été publiés dans Nature Communications sous le titre « Vers la construction d'un modèle de langage multilingue pour la médecine ».
CliquezVérifierRapport détaillé : Le test de référence dans le domaine médical surpasse Llama 3 et se rapproche de GPT-4. L'équipe de l'Université Jiaotong de Shanghai a publié un modèle médical multilingue couvrant 6 langues
En comparaison, les performances exceptionnelles de M³FM en matière de multimodalité, de multidomaine, de multilinguisme et d’autres aspects apporteront sans aucun doute une nouvelle vitalité à l’intersection de l’intelligence artificielle et des soins de santé.Bien sûr, lorsque nous parlons de cette recherche, nous devons mentionner le Dr Zheng Yefeng, l'un des auteurs de cet article.
En fait, cet article peut être considéré comme un résultat fraîchement produit, et il peut également être considéré comme le signe d’un nouveau départ pour le Dr Zheng Yefeng. Le 29 juillet 2024, Zheng Yefeng, membre de l'IEEE, membre de l'AIMBE et scientifique en intelligence artificielle médicale, a rejoint l'Université Westlake à temps plein, a été embauché comme professeur à l'École d'ingénierie et a fondé le Laboratoire d'intelligence artificielle médicale. Les axes de recherche du laboratoire comprennent l'analyse d'images médicales, la compréhension du langage naturel médical, la bioinformatique, etc. Cet article est l'une des réalisations importantes de la première année du laboratoire.
En plus de cette réalisation, le laboratoire a également publié plusieurs articles dans le domaine de la santé médicale, comme l'étude intitulée « Unlocking the Potential of Weakly Labeled Data: A Co-Evolutionary Learning Framework for Abnormality Detection and Report Generation », qui introduit un cadre collaboratif de détection d'anomalies et de génération de rapports (CoE-DG) qui utilise des données entièrement étiquetées et faiblement étiquetées pour promouvoir le développement mutuel des deux tâches de détection d'anomalies CXR et de génération de rapports. Cette recherche a été publiée dans IEEE Transactions on Medical Imaging.
Bien entendu, le laboratoire dispose également de résultats de recherche sur les grands modèles de langage actuellement populaires, comme l'étude intitulée « Atténuer les hallucinations des grands modèles de langage dans l'extraction d'informations médicales via le décodage contrastif », publiée dans EMNLP 2024. Cet article apporte une solution au phénomène selon lequel les LLM sont sujets aux « hallucinations » dans les scénarios médicaux, et propose un « décodage contrastif alternatif » (ALCD). Cette méthode peut réduire considérablement l’occurrence d’erreurs en séparant les capacités de reconnaissance et de classification du modèle et en ajustant dynamiquement les poids des deux pendant le processus de prédiction. La technologie est performante dans de nombreuses tâches médicales.
Aujourd’hui, ces réalisations sont encore au stade du laboratoire ou ont le potentiel d’être mises en œuvre, mais à terme, l’IA propulsera le domaine de la santé vers l’intelligence, l’intelligence et l’automatisation. Comme l'a déclaré le Dr Zheng Yefeng : « L'intelligence artificielle médicale est un domaine en plein essor. J'estime que dans 10 à 15 ans, l'intelligence artificielle aura la même précision que les diagnostics et les traitements des médecins et pourra être largement utilisée. »
Références :
1.https://www.nature.com/articles/s41746-024-01339-7
2.https://mp.weixin.qq.com/s/pMNXAvzgGRpPwqVtCWjXbA
3.https://mp.weixin.qq.com/s/6hw6EJY6slAIRbGGN9XY9g








