Command Palette
Search for a command to run...
Pour l'analyse d'images De Pathologie À Très Grande Échelle ! L'Université Des Sciences Et Technologies De Huazhong Propose Un Modèle De Segmentation d'images Médicales Pour Améliorer La Précision Du Diagnostic Du Syndrome De Sjögren
Bouche sèche, yeux secs, peau sèche, accompagnés de douleurs musculaires inexpliquées et de fatigue générale au quotidien. Si vous ressentez les symptômes ci-dessus, en plus de prendre en compte le temps sec en hiver, vous devez également être attentif à une maladie courante mais souvent négligée : le syndrome de Sjögren (SS).
Le syndrome de Sjögren est une maladie auto-immune caractérisée par une forte infiltration lymphocytaire des glandes exocrines.Environ 5 millions de personnes dans notre pays souffrent de cette maladie. Aux premiers stades de la maladie, les glandes exocrines (glandes salivaires, glandes lacrymales, etc.) sont détruites par des lymphocytes fortement infiltrés, ce qui entraîne leur altération fonctionnelle. Les patients souffrent souvent de sécheresse buccale et oculaire et peuvent également présenter des symptômes tels que des douleurs dans les deux articulations de l’épaule. Dans le même temps, la maladie affectera également d’autres organes importants, tels que les poumons, le foie, les reins, et affectera même la fertilité.

La détection et le diagnostic précoces du syndrome de Sjögren sont cruciaux, et la sialadénite lymphocytaire focale (FLS) est l'un des critères importants pour le diagnostic du syndrome de Sjögren. En obtenant des coupes pathologiques des glandes salivaires mineures du patient et en effectuant un examen microscopique, selon les critères diagnostiques existants,Si plus de 50 agrégats de lymphocytes étaient trouvés par 4 mm2 d’échantillon de tissu, il s’agissait d’une lésion typique.
Cependant, une image de scan pathologique complète peut atteindre 100 000*100 000 pixels, soit environ 1 milliard de pixels. Les médecins doivent examiner attentivement l’image entière et déterminer le nombre de foyers d’agrégation lymphocytaire. Cela prend non seulement du temps, mais repose souvent aussi sur l’expérience et le jugement subjectif des médecins professionnels, ce qui augmente le risque d’erreur de diagnostic ou de diagnostic manqué.
Pour relever les défis ci-dessus,Le professeur Tu Wei et le professeur Lu Feng de l'Université des sciences et technologies de Huazhong ont proposé le modèle de segmentation d'images médicales M2CF-Net utilisant la technologie de vision par ordinateur bien connue dans les domaines de la conduite autonome et de la reconnaissance faciale.En intégrant des technologies de reconnaissance d'images multi-résolution et multi-échelle, le modèle M2CF-Net peut non seulement « voir » des différences subtiles dans les images pathologiques, mais également localiser et compter avec précision les biomarqueurs clés - les foyers d'agrégation lymphocytaire, aidant les médecins à établir des diagnostics plus rapides et plus précis.
Les résultats de la recherche ont été publiés lors de la conférence internationale IEEE 2023 sur l'intelligence artificielle médicale (MedAI) sous le titre « M2CF-Net : un réseau de fusion croisée multi-résolution et multi-échelle pour la segmentation des lésions pathologiques de la sialadénite lymphocytaire focale ».
Points saillants de la recherche :
* Résolution du problème de difficulté d'identification de minuscules amas de lymphocytes dans des images de pathologie tissulaire à très grande échelle
* En intégrant la multi-résolution et la multi-échelle, M2CF-Net surpasse les trois autres modèles de segmentation sémantique d'images médicales classiques
* M2CF-Net fonctionne bien dans le traitement d'images avec des limites floues, de petits objets et des textures complexes. Ses images segmentées ont des formes plus complexes et sont très cohérentes avec la vérité fondamentale annotée par l'homme

Adresse du document :
https://doi.ieeecomputersociety.org/10.1109/MedAI59581.2023.00063
Le projet open source « awesome-ai4s » rassemble plus de 100 interprétations d'articles AI4S et fournit des ensembles de données et des outils massifs :
https://github.com/hyperai/awesome-ai4s
Ensemble de données : Données cliniques de l'hôpital Tongji
Cette étude utilise un ensemble de données de sections pathologiques de glandes salivaires mineures de l'hôpital Tongji.Parmi eux, des glandes salivaires mineures ont été retirées de patients atteints du syndrome de Sjögren primaire.
*Les glandes salivaires mineures sont réparties sous la muqueuse de la cavité buccale et du pharynx humains. Leur fonction est de sécréter de la salive, de maintenir la cavité buccale humide, de faciliter la digestion et de protéger les tissus buccaux contre les infections.
En colorant des sections pathologiques de glandes salivaires mineures, les médecins peuvent observer la structure claire des cellules au microscope. Plus précisément, les chercheurs ont examiné toutes les lames pour assurer la qualité et confirmer la présence d’une pharyngite lymphocytaire focale, caractérisée par des amas de plus de 50 lymphocytes par 4 millimètres carrés entourant la glande. S'il y a une lésion, elle est marquée.
L'ensemble de données final comprend 203 échantillons, dont 171 échantillons positifs (qui répondent aux caractéristiques de la lésion) et 32 échantillons négatifs (qui ne répondent pas aux caractéristiques de la lésion).Les chercheurs ont divisé ces échantillons en ensembles d’entraînement, ensembles de validation et ensembles de test dans une certaine proportion, qui ont été utilisés respectivement pour l’entraînement du modèle, l’ajustement et l’évaluation des performances. Au cours du processus réel, les chercheurs ont prétraité les données, ce qui a non seulement réduit la quantité de calculs mais a également amélioré la capacité de généralisation du modèle.
Conception d'un pipeline de traitement d'images à grande échelle pour optimiser la première étape de la formation du modèle
L'objectif de cette étude était d'extraire la zone de lésion de la sialadénite lymphocytaire focale (FLS) à partir de coupes de tissus de glandes salivaires mineures avec une résolution de 100 000 * 100 000. Cependant, il est impossible d'introduire directement des images gigapixels dans les réseaux neuronaux pour la formation, principalement parce que ces images sont trop grandes et que les ressources de calcul, le temps de formation, les cadres existants, etc. sont insuffisants pour les prendre en charge.
Les chercheurs ont donc conçu un pipeline pour le traitement d’images de pathologie à très grande échelle.Le pipeline comprend principalement trois étapes : l'extraction des régions d'intérêt (ROI), la normalisation des taches et le patching d'image (WSl Patching). Comme le montre la figure suivante :
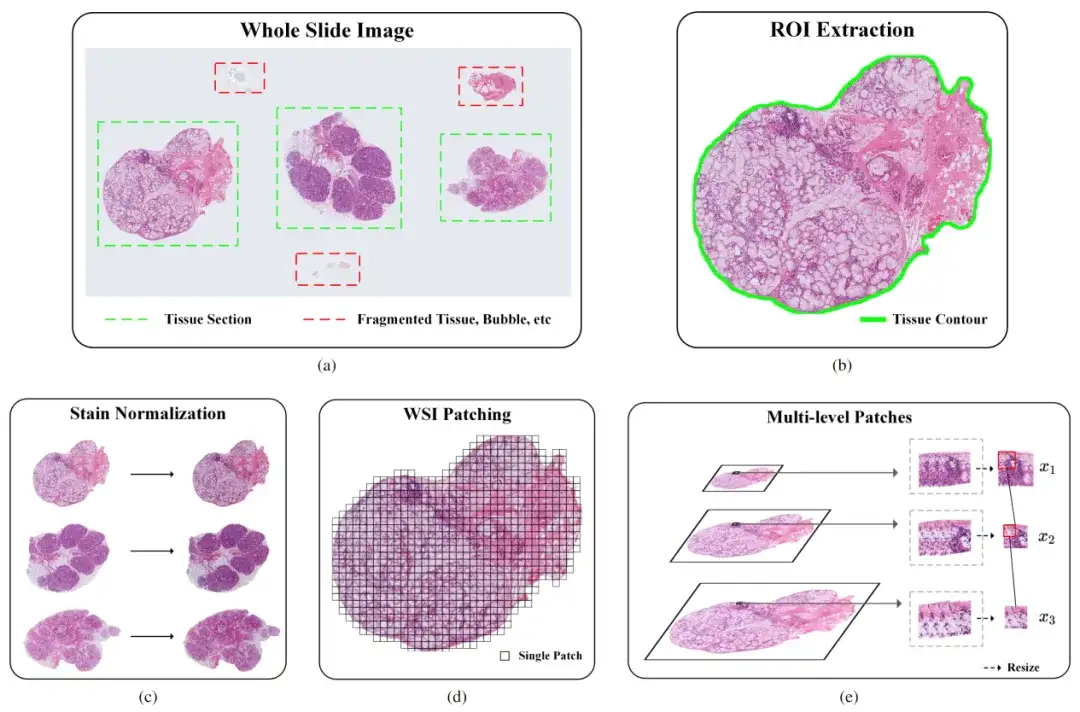
Partie 1 : Extraction du retour sur investissement
Pour améliorer la précision de l’identification de régions tissulaires spécifiques dans les images pathologiques, les chercheurs ont initialement utilisé un classificateur basé sur un réseau neuronal convolutif (CNN). Cependant, le classificateur a rencontré des difficultés dans la gestion de caractéristiques complexes telles que les bulles, les tissus fragmentés et les artefacts, ce qui a fait que ses performances ne répondent pas aux attentes. Pour résoudre ce problème, l’équipe de recherche a pris les mesures suivantes :
* Annotation manuelle : Une partie des échantillons a été annotée en détail et le modèle de classification a été réentraîné après l'enrichissement de l'ensemble de données.
* Augmentation des données : utilisez des techniques telles que la rotation, la mise à l'échelle et la translation pour augmenter la diversité des données de formation, améliorant ainsi la précision du classificateur.
Partie II : Normalisation de la coloration
L’objectif principal de la normalisation de la coloration des images de pathologie est de garantir que les images provenant de différentes sources présentent une couleur et un contraste visuels cohérents. Plus précisément, en raison de l'influence de facteurs tels que la concentration du colorant, la valeur du pH, la température et le temps, des problèmes tels qu'une coloration inégale ou une intensité incohérente se produisent souvent dans le processus de coloration réel, entraînant des effets visuels différents pour le même type de tissu. Cette différence peut affecter la précision des modèles de vision par ordinateur.
Pour résoudre ce problème, les chercheurs ont utilisé l’algorithme Vahadane. L'algorithme obtient l'effet de normalisation des couleurs en ajustant les caractéristiques de couleur de l'image source pour la rendre similaire à l'image cible. Plus précisément, il calcule la transformation de la matrice de couleurs entre l'image source et l'image cible pour obtenir la transformation de couleur de l'image source.
Partie III : Segmentation d'images
Il s’agit de résoudre le problème selon lequel la taille de l’image est toujours trop grande après l’extraction du ROI et la normalisation de la coloration, ce qui fait que l’échantillon ne peut pas être saisi dans le modèle d’apprentissage en profondeur pour la formation. Les chercheurs ont adopté une méthode de formation basée sur des patchs pour diviser l'image en petits blocs avec des zones qui se chevauchent, ce qui a non seulement amélioré l'efficacité de la formation du modèle, mais a également conservé les informations d'origine.
Afin d'analyser les caractéristiques détaillées des petits lymphocytes à proximité de canaux plus grands, il est nécessaire de capturer les caractéristiques au niveau des tissus sur un champ de vision plus large. Cependant, afin de garantir l’exactitude des résultats de segmentation, il est nécessaire de capturer les caractéristiques au niveau des cellules dans un champ de vision plus petit. Il est particulièrement important de trouver un équilibre entre les deux.
À cette fin, les chercheurs ont envisagé une méthode de segmentation d’image multi-résolution, qui consiste principalement à sous-échantillonner l’image d’origine plusieurs fois et à extraire des blocs d’image de même taille à partir de ces images sous-échantillonnées. Ces patchs découpés à partir d'images avec différents grossissements d'échantillonnage ont différentes tailles de champ de vision, qui peuvent capturer à la fois des caractéristiques au niveau des tissus et des caractéristiques au niveau des cellules.
Modèle de fusion multi-résolution et multi-échelle, amélioration efficace des performances
Le modèle M2CF-Net sélectionné par les chercheurs comprend un encodeur multi-branches et un décodeur en cascade basé sur la fusion.L'encodeur sous-échantillonne les caractéristiques des patchs de différentes résolutions à différentes échelles, tandis que le décodeur utilise un bloc de fusion en cascade pour fusionner les cartes de caractéristiques générées par les encodeurs multi-branches.
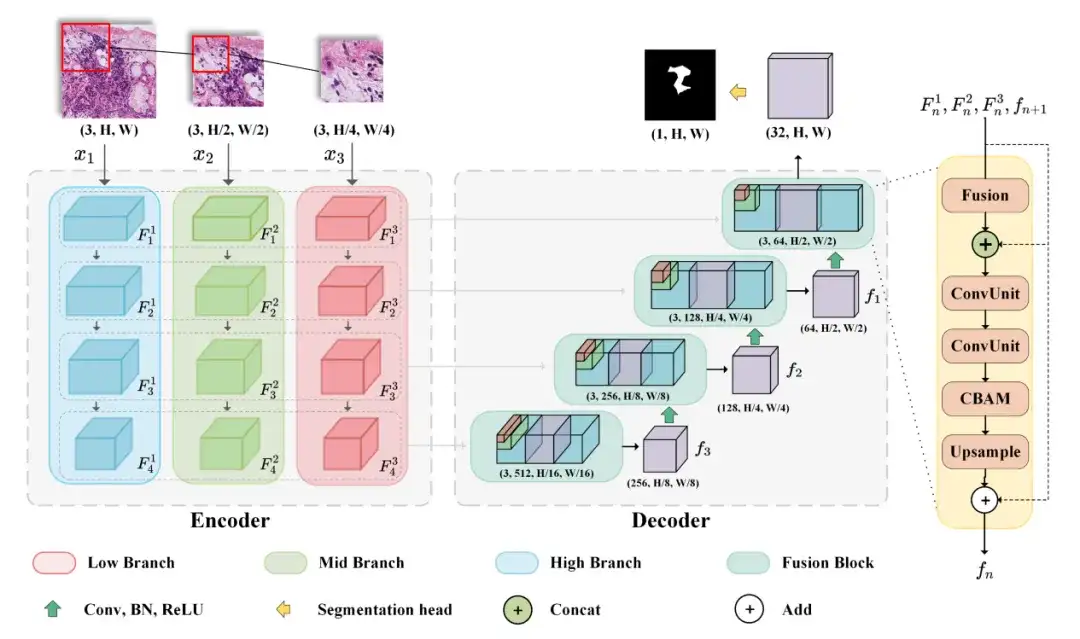
Plus précisément, afin d'obtenir simultanément des caractéristiques au niveau tissulaire et au niveau cellulaire, les chercheurs ont conçu un réseau multi-branches, qui est un modèle d'architecture encodeur-décodeur typique pouvant accepter des images de différentes résolutions en entrée. L'encodeur comprend trois branches d'entrée, qui acceptent des images de résolution de différentes tailles et génèrent des combinaisons de cartes de caractéristiques de différents champs de vision pendant le processus d'encodage. Le décodeur peut combiner les cartes de caractéristiques générées par l'encodeur à l'aide du bloc de fusion en cascade pour générer la carte de prédiction finale.
Dans ce processus, les chercheurs ont également utilisé des mécanismes d’attention spatiale et d’attention canalaire pour améliorer les capacités de représentation des caractéristiques d’entrée. Enfin, BCEDice Loss est utilisé comme fonction de perte du modèle. Cette fonction de perte pèse la perte d'entropie croisée binaire et la perte de dés, ce qui peut guider efficacement la direction d'optimisation du modèle.
Conclusion expérimentale : M2CF-Net surpasse les trois autres modèles de segmentation sémantique d'images médicales classiques
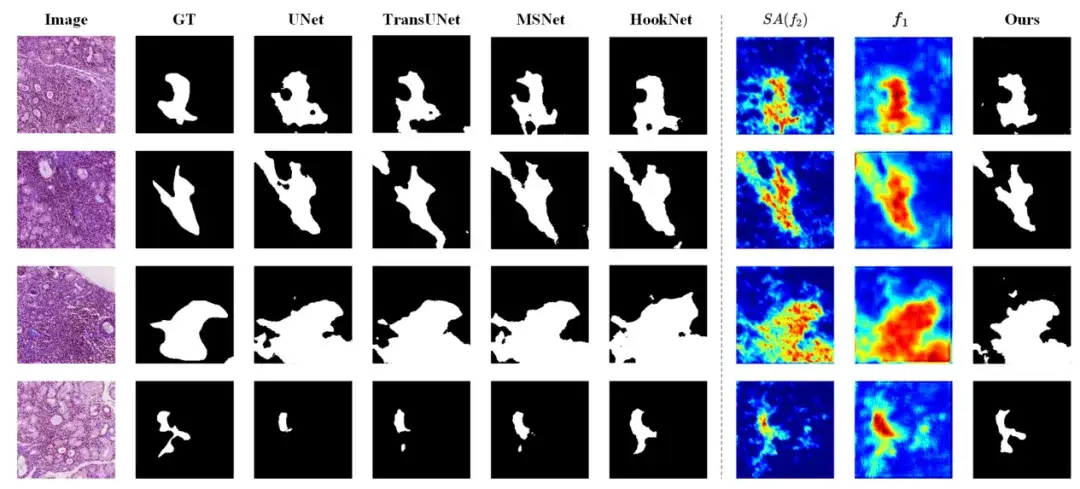
Les chercheurs ont comparé leur modèle proposé (M2CF-Net) avec quatre autres modèles populaires de segmentation sémantique d'images médicales : UNet, MSNet, HookNet et TransUNet. Les résultats ont montré que le modèle M2CF-Net présente davantage d’avantages dans l’utilisation de fonctionnalités multi-résolutions et multi-échelles.
* UNet : utilise une structure encodeur-décodeur pour capturer des fonctionnalités multi-échelles pour une segmentation précise
* MSNet : introduit un réseau de soustraction multi-échelle pour améliorer l'extraction de fonctionnalités et améliorer la précision de la segmentation
* HookNet : Hook est ajouté pour capturer et utiliser des fonctionnalités multi-résolution, améliorer la structure U-Net et gérer efficacement la segmentation d'images de différentes tailles dans les images médicales
* TransUNet : Basé sur Transformer, il améliore la précision de la segmentation en introduisant un mécanisme d'auto-attention
Comme le montre la figure ci-dessous, les chercheurs ont constaté que M2CF-Net a atteint le Dice le plus élevé de 69.40%, et son nombre de paramètres n'était que la moitié de celui de TransUNet, qui s'est classé troisième. Il a surpassé UNet et MSNet, qui avaient moins de paramètres, de 38,9% et 22,5%, respectivement.Il peut capturer et fusionner efficacement des caractéristiques de différentes échelles dans les images.

Plus précisément, le nombre de paramètres (Params) de M2CF-Net est inférieur à celui de TransUNet et HookNet, mais supérieur à celui de UNet et MSNet. Cela est dû au fait que TransUNet est basé sur l'architecture Transformer, qui possède plus de paramètres que CNN, et le décodeur à branche unique rend le nombre de paramètres de M2CF-Net inférieur à celui de HookNet. Cependant, la structure du codeur multi-branches dans M2CF-Net conduit à un nombre de paramètres plus élevé par rapport au réseau d'entrée à branche unique.
De plus, après une analyse approfondie,L’étude a révélé que M2CF-Net fonctionne bien dans le traitement d’images avec des limites floues, de petits objets et des textures complexes. Comme le montre la figure ci-dessous, les résultats de segmentation de M2CF-Net ont des formes plus complexes, qui sont cohérentes avec la vérité fondamentale annotée par l’homme.
La technologie de vision par ordinateur révolutionne la segmentation des images médicales
L’analyse d’images médicales est essentielle au diagnostic des maladies. La technologie informatique peut être utilisée pour segmenter avec précision les images médicales et identifier efficacement les zones de lésion, les organes humains et les sites d’infection, améliorant ainsi l’efficacité du diagnostic. Ces dernières années, grâce aux progrès des technologies avancées telles que l'apprentissage en profondeur, la technologie de segmentation des images médicales passe rapidement d'une opération manuelle à un traitement automatisé, et les systèmes d'IA spécialement formés sont désormais devenus un outil auxiliaire indispensable pour les professionnels de la santé.
Professeur Tu Wei, directeur adjoint du département de rhumatologie et d'immunologie, hôpital Tongji, faculté de médecine de Tongji, université des sciences et technologies de Huazhong,Il a plus de 20 ans d'expérience dans le diagnostic et le traitement des maladies rhumatismales et immunologiques et possède une vaste expérience diagnostique du syndrome de Sjögren. Dans cette étude, le professeur Tu Wei a analysé en profondeur le processus de diagnostic pathologique du syndrome de Sjögren, a souligné les points clés qui sont faciles à confondre et a démontré les résultats du diagnostic dans différentes situations à travers des cas réels. Après avoir maîtrisé la méthode de diagnostic pathologique du syndrome de Sjögren,L'équipe du professeur Lu Feng a proposé d'utiliser la technologie de segmentation d'image en vision par ordinateur pour relever les défis diagnostiques.Les deux parties ont utilisé une technologie d’IA avancée pour ouvrir une nouvelle voie pour le diagnostic du syndrome de Sjögren.
Page d'accueil personnelle du professeur Tu Wei :
https://www.tjh.com.cn/MedicalService/outpatient_doctor.html?codenum=101110
Page d'accueil personnelle du professeur Lu Feng :
http://faculty.hust.edu.cn/lufeng2/zh_CN/index.htm
Outre les chercheurs mentionnés ci-dessus, de nombreux scientifiques se consacrent à la recherche de pointe à l’intersection de l’imagerie médicale et de l’IA.
Par exemple, une équipe du Laboratoire d'informatique et d'intelligence artificielle du Massachusetts Institute of Technology (MIT CSAIL), en collaboration avec des chercheurs du Massachusetts General Hospital et de la Harvard Medical School, a proposé un modèle général, ScribblePrompt, pour la segmentation interactive d'images biomédicales.Cet outil de segmentation basé sur un réseau neuronal prend non seulement en charge les annotateurs utilisant différentes méthodes d'annotation telles que les gribouillis, les clics et les cadres de délimitation pour effectuer des tâches de segmentation d'images biomédicales flexibles, mais fonctionne également bien pour les étiquettes et les types d'images non formés.
Je crois qu’à mesure que des technologies plus avancées sont développées et appliquées dans la pratique clinique, de nombreuses branches médicales telles que l’oncologie et la neurologie en bénéficieront, et le domaine de l’analyse d’images médicales ouvrira de meilleures perspectives de développement.








