Command Palette
Search for a command to run...
Partagez Votre Expérience De Soumission À NeurIPS 2024 ! L'équipe De l'Université Du Zhejiang Utilise Le Modèle DePLM Pour Aider À l'optimisation Des Protéines, Et Le Premier Auteur De l'article Présente La Démonstration En Ligne

Harry Shum, membre étranger de l'Académie nationale d'ingénierie des États-Unis, a déclaré un jour : « S'il y a une chose que nous devons faire aujourd'hui, c'est l'IA au service de la science. Difficile d'imaginer quelque chose de plus important aujourd'hui, et l'attribution du prix Nobel cette année en est la meilleure preuve. »
Dans le passé, les scientifiques s’appuyaient sur une organisation manuelle des données et sur des hypothèses basées sur des théories thématiques. Désormais, grâce à l’aide de l’IA, la recherche est menée directement sur la base de données massives. L’IA pour la science a non seulement amélioré l’efficacité de la recherche scientifique, mais a également changé l’ensemble du paradigme de la recherche scientifique, ce qui est particulièrement évident dans le domaine de la recherche sur les protéines.
Dans le cinquième épisode de Meet AI4S, HyperAI a eu la chance d'inviter Wang Zeyuan, un doctorant du Knowledge Engine Laboratory de l'Université du Zhejiang,Il a donné une introduction détaillée à un article de l'équipe sélectionnée pour NeurIPS 2024, intitulé « Utiliser le processus de débruitage par diffusion pour aider les grands modèles à optimiser les protéines ». « DePLM : débruitage des modèles de langage protéique pour l'optimisation des propriétés ».
En tant que conférence de premier plan dans le domaine de l'IA, NeurIPS est connue comme l'une des conférences universitaires sur l'IA les plus difficiles, les plus pointues et les plus influentes. Cette année, la conférence a reçu un total de 15 671 soumissions d'articles valides, soit une augmentation de 27% par rapport à l'année dernière, mais le taux d'acceptation final n'était que de 25,8%. Les articles sélectionnés sont d’une grande valeur pédagogique.Lors de cette séance de partage, le Dr Wang Zeyuan a présenté en détail le concept de conception, les conclusions expérimentales, le mode de fonctionnement de démonstration et les perspectives d'avenir du modèle de langage protéique de débruitage DePLM. Il a également partagé son expérience en matière de soumission d’articles à des conférences de haut niveau, espérant que cela sera utile à tout le monde.
Plus précisément, le Dr Wang a déclaré que lorsque nous soumettons des articles, nous pouvons commencer par la sélection du sujet, les points innovants, la rédaction de l'article et la gestion des examens interdisciplinaires.
Tout d’abord, en termes de choix du sujet,Vous pouvez lire un large éventail d’articles de conférence de premier plan pour comprendre les orientations de recherche les plus significatives qui préoccupent actuellement la communauté. Par exemple, lors de la préparation de l'article DePLM, le Dr Wang a découvert que l'ingénierie des protéines, en particulier les tâches de prédiction des protéines, était un sujet brûlant lors des conférences ICLR et NeurIPS de l'année dernière.
Deuxièmement, en termes d’innovation,Il estime qu’il est important de cultiver la capacité de découvrir les problèmes. Dans le domaine de l’IA pour la science, nous devons d’abord avoir une compréhension approfondie des connaissances dans le domaine de la science et les comparer avec le contenu du domaine de l’IA pour découvrir les domaines vides qui n’ont pas encore été explorés par l’IA.
En termes de rédaction d'essais,Il a déclaré que l’écriture doit être logiquement claire et détaillée pour garantir que l’article soit facile à comprendre. Il est également nécessaire de communiquer davantage avec les tuteurs et les camarades de classe pour éviter de tomber dans ses propres schémas de pensée fixes.
Enfin, considérant que les articles sur l’IA pour la science peuvent être examinés par des évaluateurs issus de deux horizons différents, l’un se concentrant davantage sur la technologie de l’IA et l’autre sur les applications scientifiques,Il est donc nécessaire de clarifier le positionnement central du document lors de la rédaction.C'est-à-dire que cet article s'adresse à la communauté de l'IA ou à la communauté scientifique, et un cadre logique est construit en conséquence pour garantir que le contenu est étroitement lié au sujet.
Selon lui, la tendance actuelle de la recherche sur les modèles à grande échelle a changé. Nous sommes passés d’une simple approche de copie à une compréhension approfondie des modèles à grande échelle.Dans le passé, nous laissions les grands modèles s’adapter à diverses tâches en aval, mais maintenant nous nous préoccupons davantage de la manière dont les tâches en aval peuvent mieux coopérer avec l’étape de pré-formation des grands modèles. Plus l’adéquation entre les deux est élevée, meilleures sont les performances du modèle.
Par exemple, pour prédire les paysages adaptatifs, les méthodes traditionnelles de réglage fin simples sont peu performantes en termes de capacité de généralisation. Nous devons avoir une compréhension plus approfondie des grands modèles et des paradigmes d’apprentissage non supervisé pour identifier leurs lacunes et les améliorer. En outre, nous devons également prêter attention aux défauts des grands modèles eux-mêmes, par exemple en explorant les moyens d’éliminer les biais du modèle afin d’optimiser ses performances.
Modèle open source et testable
Aujourd'hui, j'aimerais partager un article que nous avons publié à NeurIPS 2024, qui explore comment utiliser le modèle de débruitage par diffusion pour aider à l'optimisation de grands modèles de langage pour les protéines.Dans cet article, nous proposons un nouveau modèle de langage protéique débruité (DePLM)Le cœur de cette approche est de considérer les informations évolutives capturées par le modèle de langage protéique comme un mélange d’informations pertinentes et non pertinentes pour les caractéristiques cibles, où les informations non pertinentes sont considérées comme du « bruit » et éliminées. Nous constatons que la procédure de débruitage basée sur le classement proposée peut améliorer considérablement les performances d’optimisation des protéines tout en maintenant de fortes capacités de généralisation.
Actuellement, DePLM est open source. En raison de l'environnement de configuration complexe du modèle,Nous avons lancé « DePLM : Optimisation des protéines avec des modèles de langage débruités (petits échantillons) » dans la section tutoriel du site officiel HyperAI.Afin de vous aider à mieux comprendre et reproduire notre travail, je vais vous expliquer comment fonctionne le modèle sous plusieurs aspects, notamment comment fonctionne le modèle DePLM, quels sont ses fichiers de configuration associés, comment affiner les étapes de diffusion du modèle et comment exécuter le modèle DePLM avec votre propre ensemble de données.
Adresse open source DePLM :
https://github.com/HICAI-ZJU/DePLM
Adresse du tutoriel DePLM :
Contexte : Maximiser l'utilisation des informations évolutives et minimiser l'introduction de signaux de biais dans les données
L'objet de recherche de cet article est la protéine, qui est une macromolécule biologique composée de 20 acides aminés en série. Il remplit des fonctions telles que la catalyse, le métabolisme et la réplication de l'ADN dans le corps et est également le principal exécuteur des activités vitales. Les biologistes divisent généralement sa structure en 4 niveaux. Le premier niveau décrit comment les protéines sont composées ; le deuxième niveau décrit la structure locale des protéines, comme les hélices α et les replis β communs ; le troisième niveau décrit la structure tridimensionnelle globale des protéines ; et le quatrième niveau considère les interactions entre les protéines.
Actuellement, la plupart des recherches sur l’IA et les protéines peuvent être attribuées à la recherche sur le traitement du langage naturel, car il existe des similitudes entre les deux. Par exemple, nous pouvons comparer la structure quaternaire des protéines à celle des lettres, des mots, des phrases et des paragraphes du langage naturel. Lorsqu'une erreur de lettre se produit dans une phrase, la phrase perd son sens. De même, une mutation dans les acides aminés d’une protéine peut rendre celle-ci incapable de former une structure stable et ainsi perdre sa fonction.
Comme le montre la figure ci-dessous, dans l'article « Conception de protéines contrôlables avec des modèles de langage », les chercheurs ont fait correspondre le langage naturel aux protéines. Cette approche a été largement reconnue par les chercheurs. Depuis 2020, la recherche sur les protéines connaît une croissance explosive.
Article original :
https://www.nature.com/articles/s42256-022-00499-z
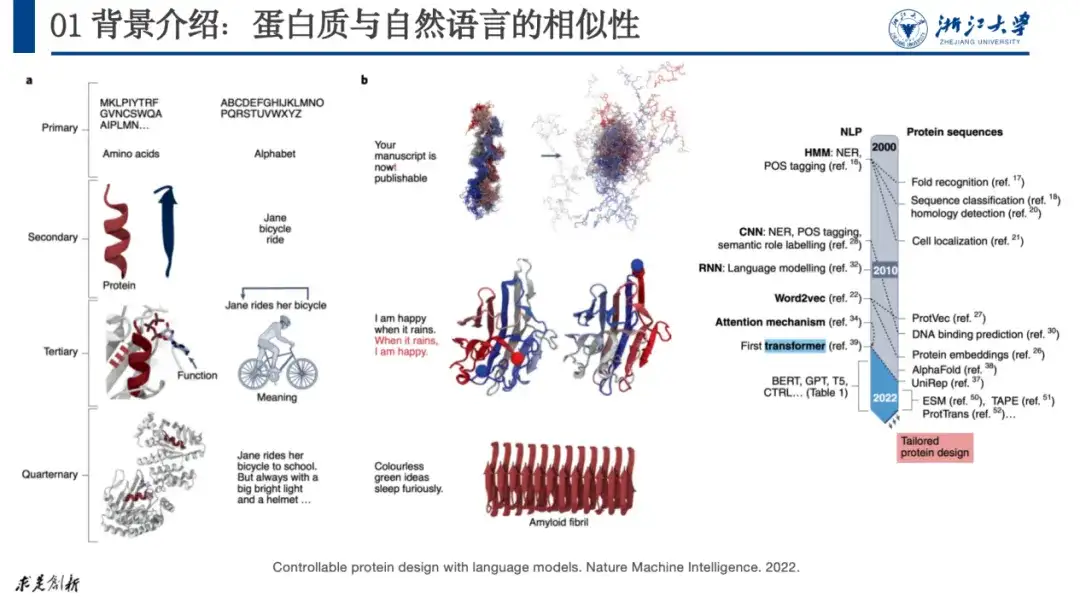
Le problème dont nous discutons cette fois est l'optimisation de l'IA + des protéines, c'est-à-dire, si nous avons une protéine qui ne fonctionne pas comme prévu, comment ajuster sa séquence d'acides aminés pour répondre à la fonction attendue.
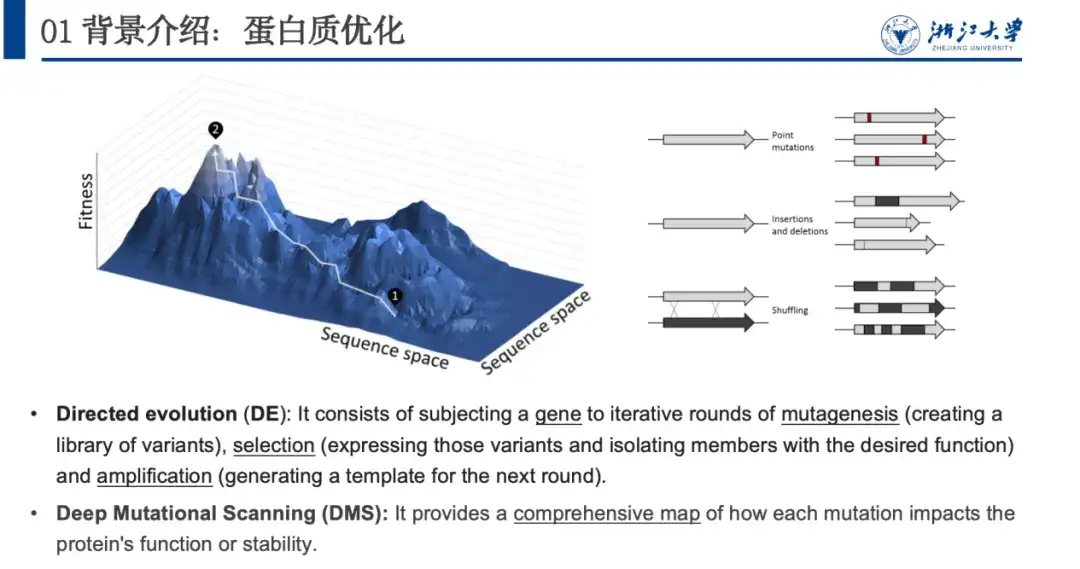
Dans la nature, les protéines s’optimisent continuellement par le biais de changements aléatoires, notamment des insertions ponctuelles, des suppressions ou des mutations ponctuelles. En imitant ce processus, les biologistes ont proposé une évolution dirigée et une analyse mutationnelle approfondie pour optimiser les protéines. Le problème avec ces deux méthodes est qu’elles consomment trop de ressources expérimentales. donc,Nous utilisons des méthodes informatiques pour modéliser la relation entre les protéines et leur aptitude physique, c'est-à-dire prédire le paysage d'aptitude physique, ce qui est crucial pour l'optimisation des protéines.
Afin de modéliser ce problème, nous utilisons généralement des ensembles de données, des mesures d’évaluation et des méthodes de calcul.Comme le montre la figure ci-dessous, un ensemble de données d'optimisation de protéines contient généralement une séquence de type sauvage xwt, plusieurs paires de mutations μi et la valeur de fitness prédite yi après mutation. Les modèles d’évaluation s’appuient principalement sur le coefficient de corrélation de Spearman. Cet indicateur ne se concentre pas sur la valeur prédite spécifique, mais sur le classement des changements de valeur de fitness causés par les mutations. Plus la valeur de classement de la mutation réelle R(Y) est proche du score de fitness prédit, meilleur est l'entraînement du modèle.
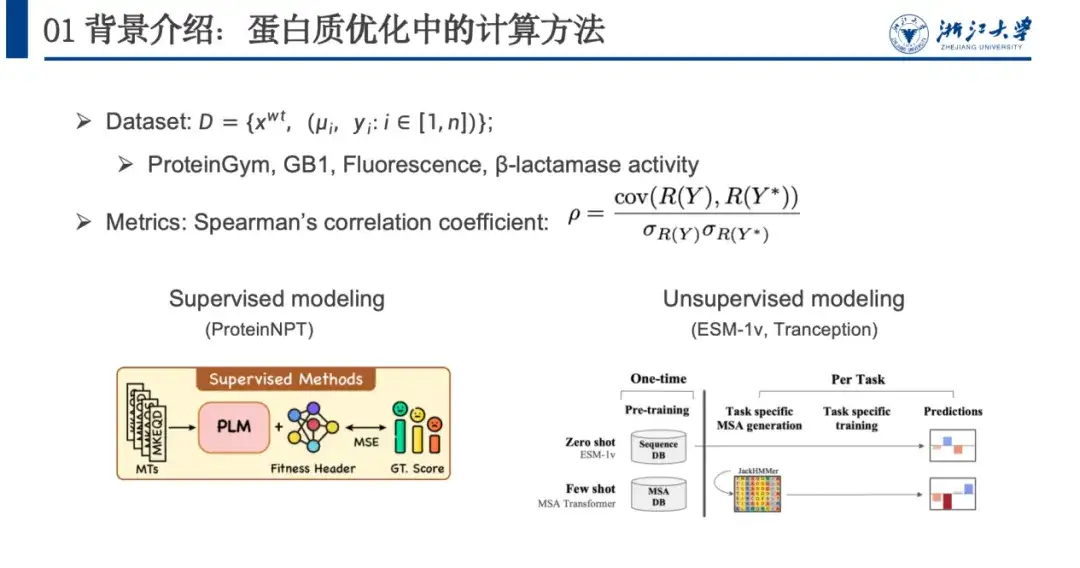
Les méthodes de calcul peuvent être grossièrement divisées en modélisation supervisée et modélisation non supervisée. L'apprentissage supervisé s'appuie sur des données étiquetées et entraîne le modèle en optimisant la fonction de perte pour améliorer la capacité prédictive de la condition physique. L'apprentissage non supervisé ne nécessite pas de données étiquetées, mais effectue plutôt un apprentissage auto-supervisé sur un ensemble de données protéiques à grande échelle qui n'est pas lié à la condition physique. Le modèle n'a besoin d'être formé qu'une seule fois et peut être généralisé à diverses tâches de prédiction de protéines.
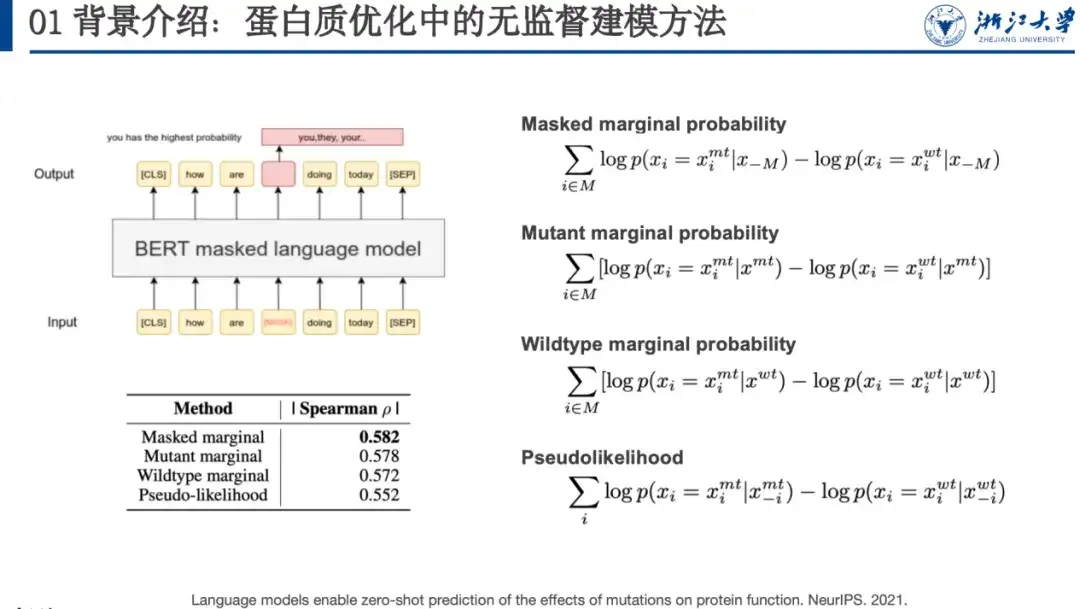
Par exemple, la modélisation du langage masqué est une méthode d’apprentissage non supervisé. Lors de la formation du modèle, nous devons fournir au modèle une séquence contaminée. Nous pouvons masquer un mot (comme le mot dans la case rouge dans la figure ci-dessous) ou le changer aléatoirement en un autre mot et laisser le modèle de langage le restaurer, c'est-à-dire restaurer la séquence d'origine. Dans un article publié à NeurIPS 2021, les chercheurs ont découvert que la probabilité de mutation protéique prédite par ces modèles est corrélée au paysage de fitness. À cette fin, ils ont conçu quatre formules de notation des mutations, comme indiqué sur le côté droit de la figure ci-dessous.
Article original :
https://proceedings.neurips.cc/paper/2021/file/f51338d736f95dd42427296047067694-Paper.pdf
En résumé, les méthodes supervisées fonctionnent bien mais ont des capacités de généralisation limitées, tandis que les méthodes non supervisées fonctionnent légèrement moins bien mais ont de fortes capacités de généralisation.Afin de combiner les avantages des deux, comme le montre la figure ci-dessous, nous avons emprunté la stratégie Pre-train + Fine-tune au domaine du PNL. Après quelques tentatives, nous avons constaté que même si cette méthode fonctionnait bien, elle avait une faible capacité de généralisation, similaire à l’apprentissage supervisé. Nous avons ensuite analysé pourquoi les méthodes non supervisées ont d’excellentes capacités de généralisation et émis l’hypothèse que cette capacité de généralisation provient de l’information évolutive (IE). C’est parce que les organismes peuvent optimiser les protéines grâce à l’évolution naturelle, et ces mutations évolutives seront également conservées. Par conséquent, nous considérons que la corrélation entre la probabilité de mutation et le paysage de fitness est positivement corrélée.

Cependant, lorsque nous essayons d’affiner le modèle, nous utilisons en réalité les informations d’intégration et n’utilisons pas pleinement les informations évolutives. De plus, il existe un biais d’informations non pertinentes dans les données expérimentales humides. Nous pensons que les informations évolutives contiennent des informations complètes dans diverses directions, telles que la stabilité, l'activité, l'expression, la liaison, etc. Lorsque nous optimisons la stabilité des protéines, l'évolution de l'activité, de l'expression et de la liaison n'est pas une information pertinente. Si la valeur de probabilité de cette information inintéressante peut être supprimée, les performances du modèle peuvent être améliorées. Étant donné que l’ensemble du processus est réalisé dans l’espace de vraisemblance, cela n’affectera pas la capacité de généralisation du modèle.Par conséquent, nous devons maximiser l’utilisation des informations évolutives lors du réglage fin tout en minimisant le signal de biais introduit dans l’ensemble de données.
Cadre d'algorithme DePLM : modèle de débruitage basé sur l'espace de tri
Sur cette base, nous avons proposé le modèle DePLM, dont l’idée centrale est de considérer les informations évolutives capturées par le modèle de langage protéique comme une fusion de signaux intéressants et inintéressants. Ce dernier est considéré comme du « bruit » dans la tâche d’optimisation des attributs cibles et doit être éliminé. DePLM débruite les informations évolutives en effectuant un processus de diffusion dans l'espace d'ordination des valeurs d'attribut, améliorant ainsi la capacité de généralisation du modèle et prédisant les effets de mutation.
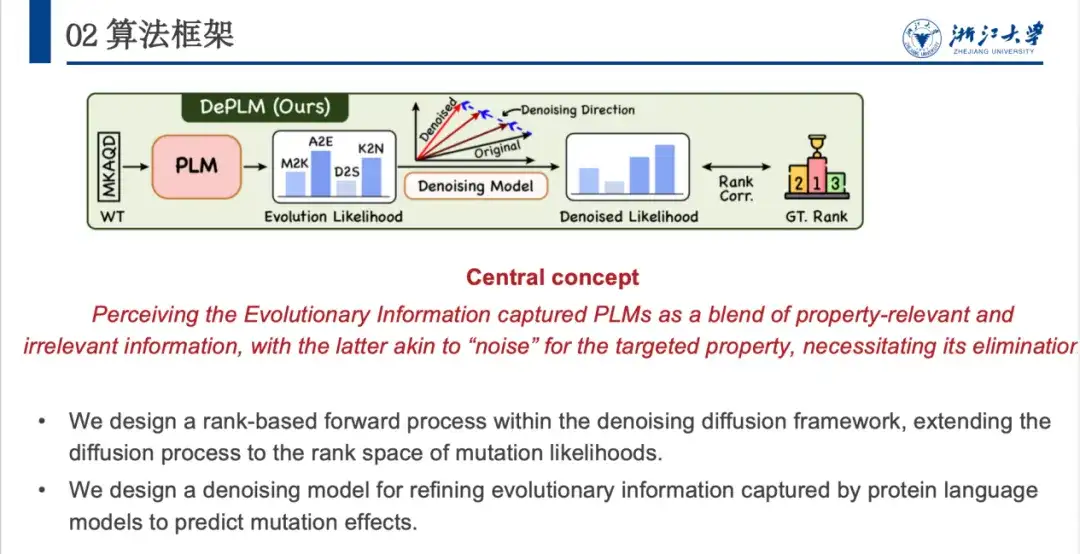
Étant donné une séquence d'acides aminés d'une protéine, le modèle prédit la probabilité que chaque position mute en divers acides aminés, et la probabilité évolutive génère ensuite la probabilité de la propriété d'intérêt via le module de dénosage. Spécifiquement,DePLM se compose principalement de deux parties : le processus de diffusion vers l'avant et le processus de débruitage vers l'arrière appris.Dans le processus direct, une petite quantité de bruit est progressivement ajoutée à la situation réelle, et dans le processus de débruitage inverse, un apprentissage est effectué pour supprimer progressivement le bruit accumulé et restaurer la situation réelle.
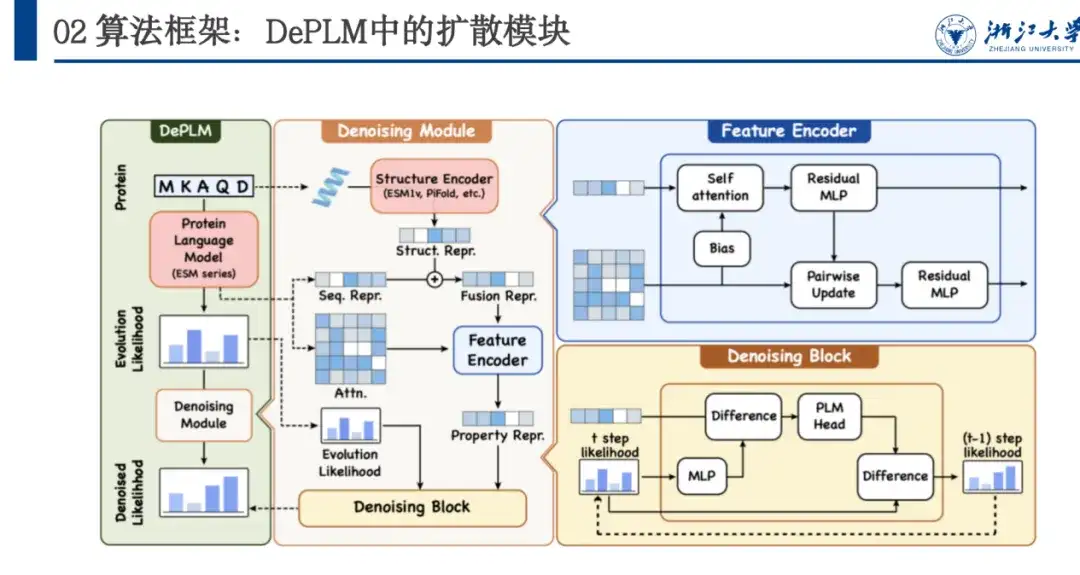
Comme le montre la figure ci-dessous, DePLM est basé sur la série ESM et adopte l’architecture Transformer. Son module de dénosage est basé sur la formation au processus de diffusion et l'architecture réseau comprend Feature Encoder et Denosing Block. Feature Encoder extrait les caractéristiques de séquence du modèle de langage protéique et extrait les caractéristiques structurelles via le modèle ESM 1v. Ces deux fonctionnalités sont utilisées comme points d'ancrage et plusieurs séries d'itérations du bloc de débruitage sont utilisées pour débruiter progressivement et obtenir la vraisemblance débruitée.
Dans le passé, les méthodes de débruitage étaient principalement utilisées dans le domaine de la génération d'images, en particulier dans les modèles de diffusion. Comme le montre la figure ci-dessous, l'image d'origine x0 est convertie en un espace de bruit (xT) proche d'une distribution gaussienne via un processus de débruitage défini, puis le modèle apprend le processus de débruitage inverse.
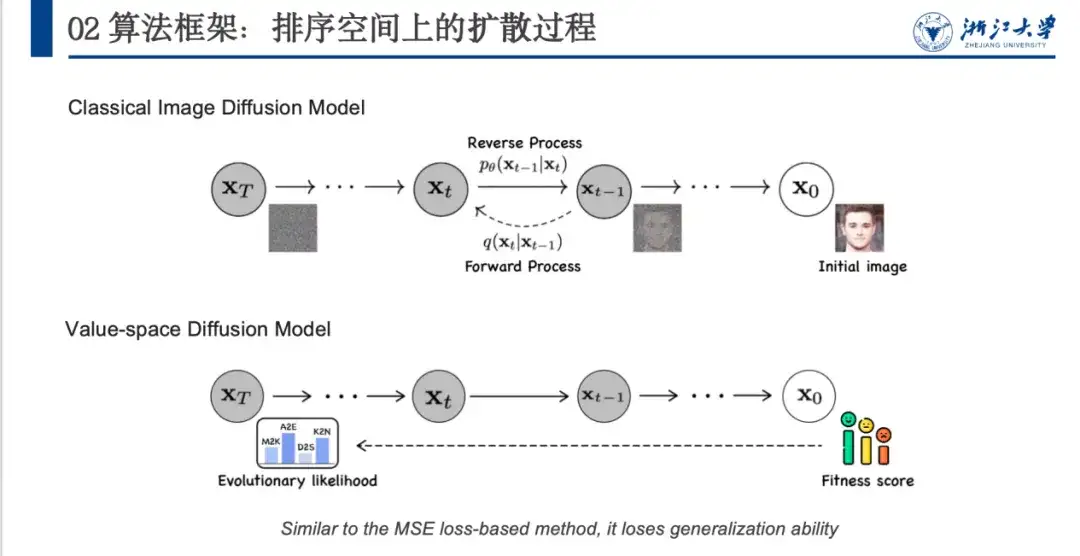
Cependant, l’application directe des modèles de débruitage d’images au domaine des protéines pose certains problèmes. Comme le montre la figure ci-dessus, le modèle de débruitage d'image peut ajouter du bruit aléatoire pour former un espace de bruit inséparable (de x0 à xT). Cependant, les protéines ont un score de fitness et une probabilité évolutive, et les états initial et final sont fixes. Par conséquent, le processus d’ajout de bruit doit être soigneusement conçu. Deuxièmement, le modèle s’alignera sur le score de fitness, ce qui se traduira par de bonnes performances mais une faible capacité de généralisation.
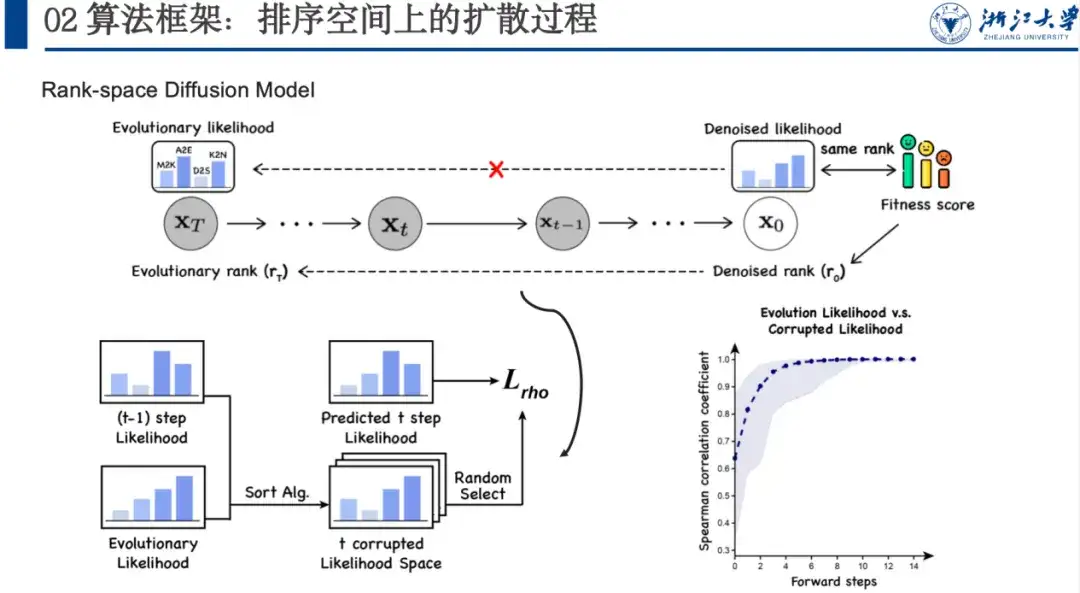
Nous proposons ensuite un modèle de débruitage basé sur l'espace de classement, en mettant l'accent sur la maximisation de la pertinence du classement.C'est parce que nous voulons débruiter la vraisemblance évolutive de l'espace de propriétés d'intérêt. Bien que nous ne connaissions pas la situation spécifique de cet espace, nous savons que son tri est cohérent avec le tri de fitness.
Nous ajoutons du bruit à cet espace et laissons le modèle apprendre un grand nombre d'ensembles de données, apprenant progressivement à quoi devrait ressembler la vraisemblance dénosurée, plutôt que d'aligner directement le score de fitness. Dans ce processus bruyant vers l'avant, nous utilisons un algorithme de tri pour rendre chaque étape du tri plus proche de l'état final et contenir le caractère aléatoire. Le modèle apprendra également l’idée du tri étape par étape inverse. Plus précisément, comme le montre la figure ci-dessous, si nous avons xt-1, nous pouvons alimenter xt-1 et xT vers l'algorithme de tri et le laisser trier plusieurs fois. Après avoir obtenu l'espace de tri de la t-ième étape, nous pouvons échantillonner aléatoirement les variables de tri de la t-ième étape, laisser le modèle prédire la vraisemblance de l'étape t+1 à la t-ième étape et calculer la perte de Spearman. Comme nous n’avons pas besoin d’ajouter de nombreuses étapes comme la débruitisation de l’image, le processus de tri peut généralement être réalisé en 5 à 6 étapes, ce qui améliore également l’efficacité.
Conclusion expérimentale : DePLM présente des performances supérieures et une forte capacité de généralisation
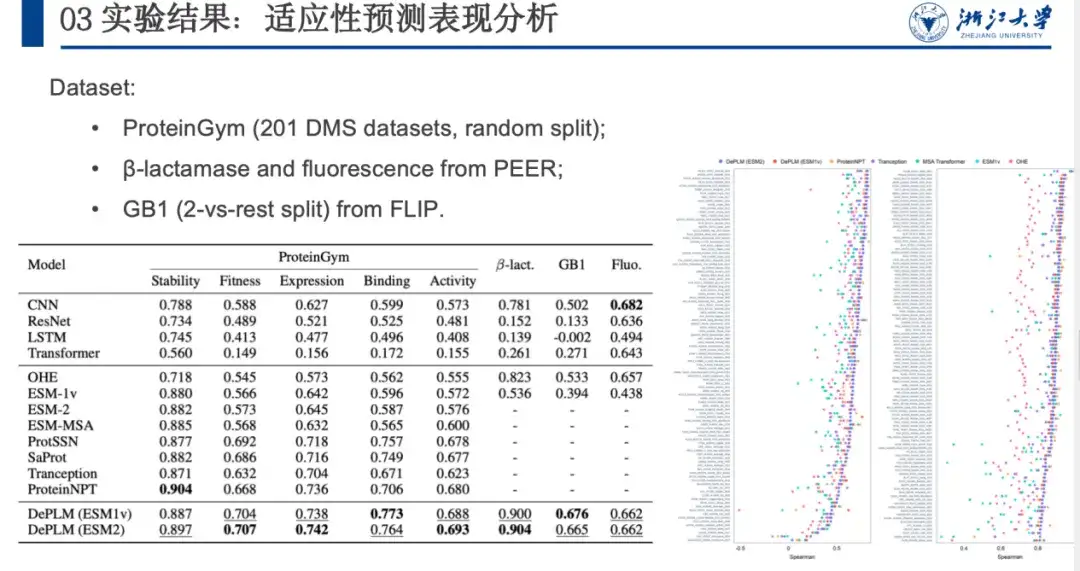
Pour évaluer les performances de DePLM dans les tâches d'ingénierie des protéines, nous le comparons à des encodeurs de séquences protéiques formés à partir de zéro, à des modèles auto-supervisés, etc. sur les ensembles de données ProteinGym, β-lactamase, GB1 et Fluorescence. Les résultats sont présentés dans la figure ci-dessous. DePLM surpasse le modèle de base.Nous constatons que des informations évolutives de haute qualité peuvent améliorer considérablement les résultats de réglage fin, ce qui démontre l'efficacité de notre procédure de formation au débruitage proposée et confirme l'avantage d'intégrer des informations évolutives avec des données expérimentales dans les tâches d'ingénierie des protéines.
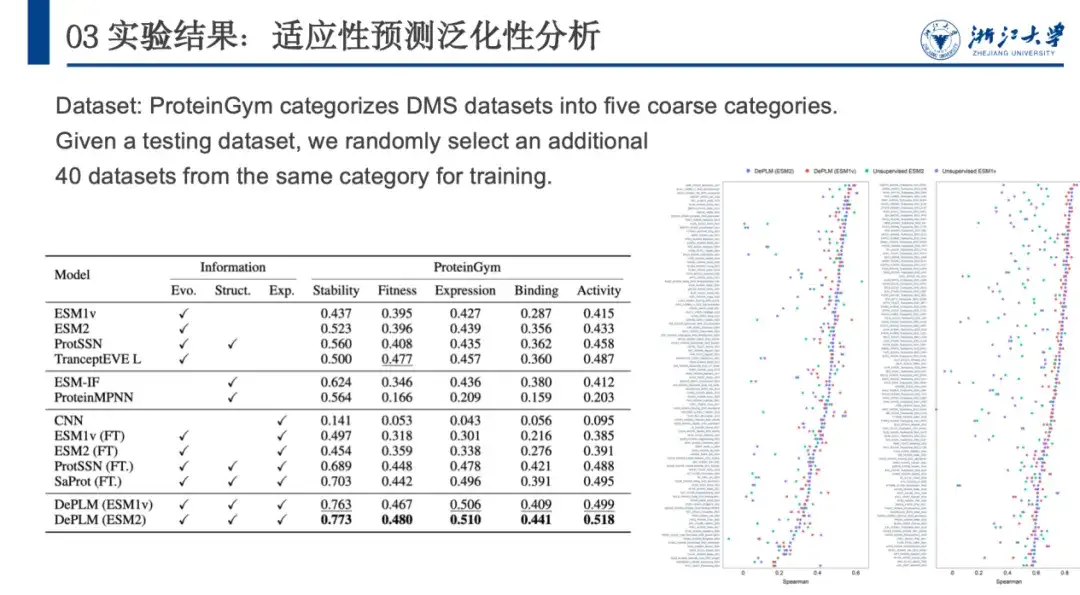
Ensuite, pour évaluer davantage la capacité de généralisation de DePLM, ProteinGym a classé l’ensemble de données DMS en cinq catégories en fonction des propriétés protéiques mesurées, à savoir la stabilité, la forme physique, l’expression, la liaison et l’activité. Nous le comparons à d’autres modèles auto-supervisés, modèles basés sur la structure et modèles de base supervisés. Les résultats sont présentés dans la figure ci-dessous. DePLM surpasse tous les modèles de base.Cela montre que les modèles qui s’appuient uniquement sur des informations évolutives non filtrées sont insuffisants, car ils diluent souvent les attributs cibles en raison de l’optimisation simultanée de plusieurs objectifs. En éliminant l’influence des facteurs non pertinents, DePLM améliore considérablement les performances.
Pour analyser plus en détail les performances de généralisation et déterminer l’importance de filtrer les informations non pertinentes pour les attributs, nous avons effectué une validation croisée de la formation et des tests entre les attributs. Comme le montre la figure ci-dessous, dans la plupart des cas, lorsque le modèle est formé sur l’attribut A et testé sur l’attribut B, ses performances sont inférieures à celles lorsqu’il est formé et testé sur le même attribut (c’est-à-dire A).Cela montre que les directions d’optimisation des différentes propriétés ne sont pas cohérentes et qu’il existe une interférence mutuelle, ce qui confirme notre hypothèse initiale.
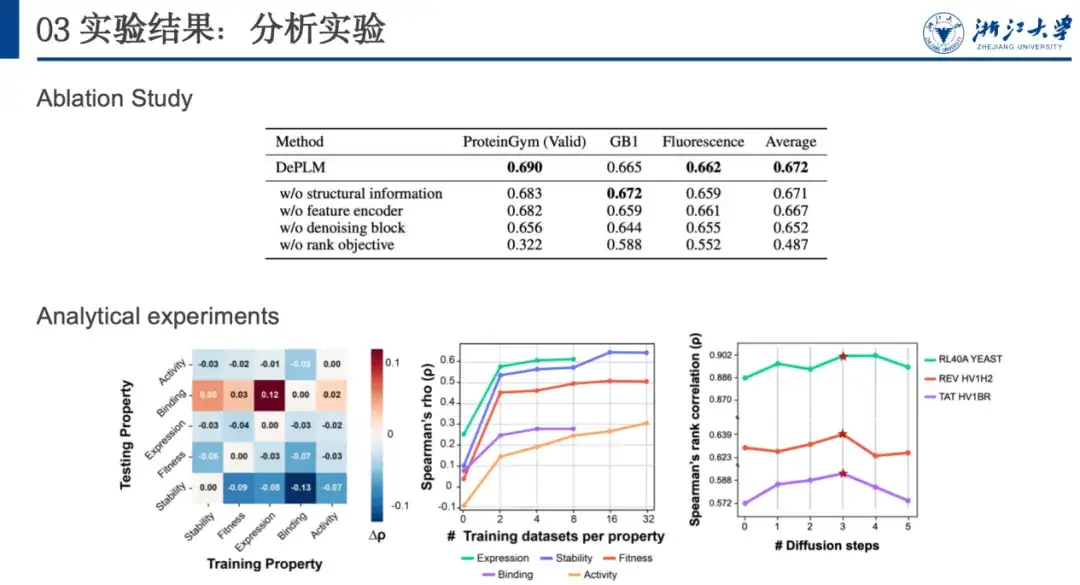
De plus, nous avons constaté que les performances du modèle étaient améliorées par la formation sur des ensembles de données d’autres propriétés et par des tests sur l’ensemble de données de liaison. Cela peut être attribué au volume limité de données et à la faible qualité des données de l'ensemble de données de liaison, ce qui conduit à sa propre capacité de généralisation insuffisante. Cela nous inspire,Lors de l'optimisation des protéines avec de nouvelles propriétés, s'il existe moins d'ensembles de données liés à cette propriété, on peut envisager d'utiliser des données avec des propriétés liées pour la réduction du bruit et la formation afin d'obtenir de meilleures capacités de généralisation.
Continuer à approfondir le domaine des protéines
L'invité de cette émission en direct est Wang Zeyuan, doctorant au Laboratoire de moteur de connaissances de l'Université du Zhejiang. Son équipe, dirigée par le professeur Chen Huajun, le chercheur Zhang Qiang et d'autres, est engagée dans la recherche universitaire dans les domaines des graphes de connaissances, des grands modèles de langage, de l'IA pour la science, etc., et a publié de nombreux articles lors de grandes conférences sur l'IA telles que NeurIPS, ICML, ICLR, AAAI et IJCAI.
Page d'accueil personnelle de Zhang Qiang :
https://person.zju.edu.cn/H124023
Dans le domaine des protéines, l’équipe a non seulement proposé des modèles avancés tels que DePLM pour optimiser les protéines, mais a également travaillé à combler le fossé entre les séquences biologiques et le langage humain.À cette fin, ils ont proposé le modèle InstructProtein.Utiliser les instructions de connaissances pour aligner le langage des protéines et le langage humain, explorer les capacités de génération bidirectionnelle entre le langage des protéines et le langage humain, intégrer des séquences biologiques dans de grands modèles linguistiques et combler efficacement le fossé entre les deux langages. Des expériences sur un grand nombre de tâches de génération de texte protéique bidirectionnel démontrent qu'InstructProtein surpasse les LLM de pointe existants.
Cliquez pour voir plus de détails : Sélectionné pour la conférence principale de l'ACL2024 | InstructProtein : Aligner le langage des protéines sur le langage humain à l'aide d'instructions de connaissances
En outre, l'équipe a également proposé une méthode de conception de séquences protéiques polyvalente PROPEND basée sur le cadre de « pré-formation et d'incitation ».Grâce à des invites vers des squelettes, des plans, des étiquettes de fonction et leurs combinaisons, une variété de propriétés peuvent être directement contrôlées, et cette méthode présente une grande praticité et une grande précision. Parmi les cinq séquences testées dans des expériences in vitro, le taux de récupération fonctionnelle maximal de PROPEND a atteint 105,2%, dépassant significativement les 50,8% du pipeline de conception classique.
Article original :
https://www.biorxiv.org/content/10.1101/2024.11.17.624051v1
À l’heure actuelle, de nombreux résultats publiés par l’équipe sont en open source. Ils recrutent également des chercheurs postdoctoraux exceptionnels, 100 personnes, des ingénieurs R&D et d'autres chercheurs à temps plein sur une base à long terme. Tout le monde est le bienvenu ~
Page d'accueil Github du laboratoire :
http://github.com/zjunlp









