Command Palette
Search for a command to run...
Soyez Le Premier À Découvrir La Démo ! Le Modèle Basé Sur Le Génome Evo Fait La Couverture De Science, Permettant La Prédiction Et La Génération Des Échelles Moléculaires Aux Échelles Génomiques

Récemment, l’IA a fait de grands progrès dans le domaine de la biologie.Une équipe de recherche de l'Université de Stanford et de l'Arc Institute aux États-Unis a proposé un modèle basé sur le génome Evo, qui peut réaliser une prédiction à échantillon zéro et une génération de haute précision dans des tâches multimodales d'ADN, d'ARN et de protéines.
La recherche connexe a été publiée dans Science sous la forme d'un article de couverture intitulé « Modélisation et conception de séquences de l'échelle moléculaire à l'échelle du génome avec Evo ».
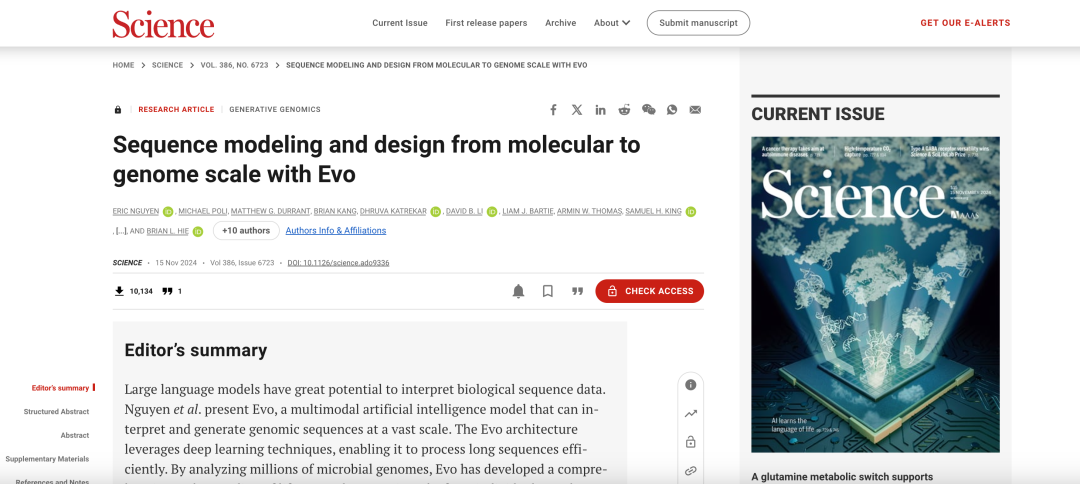
Adresse du document :
https://www.science.org/doi/10.1126/science.ado9336
Suivez le compte officiel et répondez « Evo » pour obtenir le PDF complet
En tant que premier auteur de l'article, Eric Nguyen a publié plusieurs mises à jour pour présenter les réalisations d'Evo, a remercié les membres de l'équipe à plusieurs reprises et a déclaré franchement : « C'est un honneur de travailler avec une équipe aussi incroyable ! »

Selon le journal, Evo utilise l'architecture StripedHyena et est formé sur un grand ensemble de données génomiques contenant plus de 80 000 génomes bactériens et archéens.et des millions de séquences de phages et de plasmides prédites, couvrant 300 milliards de jetons nucléotidiques, capables de générer des séquences d'ADN avec une architecture génomique raisonnable de plus d'une mégabase de longueur.
aussi,Avec une échelle de paramètres de 7 milliards et une longueur de contexte maximale de 131 072 jetons, Evo peut révéler la co-évolution complexe entre les séquences codantes et non codantes et concevoir des systèmes biologiques complexes.Tels que les complexes CRISPR-Cas et les transposons IS200 et IS605.
En résumé, la capacité d'Evo à prédire, générer et concevoir des séquences génomiques entières fournit non seulement un nouveau support théorique pour les sciences de la vie, mais devrait également être appliquée à l'édition de gènes, à la découverte de médicaments, au diagnostic des maladies, à l'agriculture et à d'autres domaines, contribuant à développer des résultats révolutionnaires dans de multiples domaines.
De nombreux internautes ont été choqués par la sortie d'Evo et attendaient avec impatience l'application spécifique de ce modèle.
Afin de permettre à chacun de découvrir pour la première fois les puissantes fonctions du modèle Evo,La section Tutoriel HyperNeural d'HyperAI est désormais en ligne « Evo : Prédiction et génération de l'échelle moléculaire à l'échelle génomique ».Pas besoin de saisir de commandes, juste un clonage en un clic pour une expérience rapide !
Lien du tutoriel :
Essai de démonstration
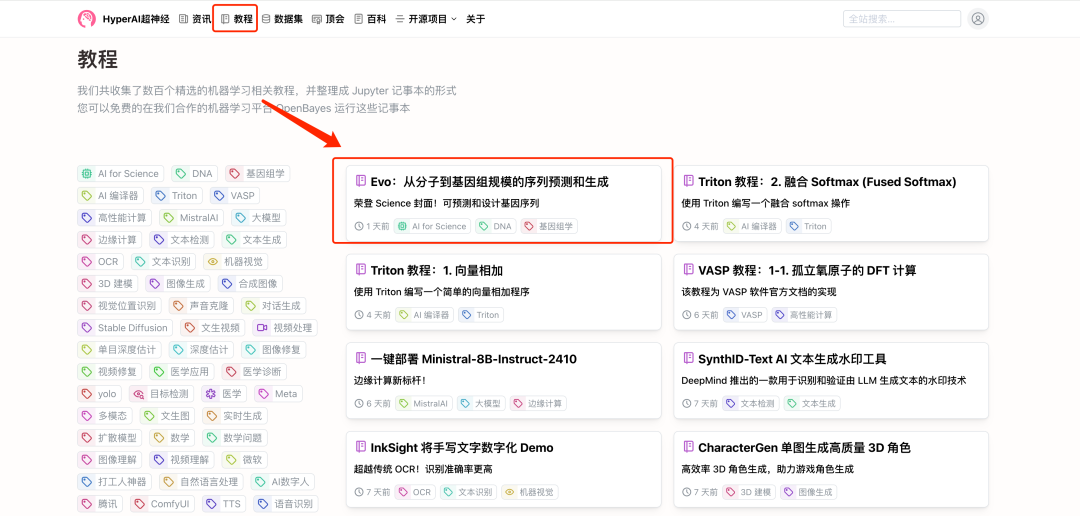
1. Connectez-vous à hyper.ai, sur la page Tutoriels, sélectionnez Evo : Prédiction et génération de l'échelle moléculaire à l'échelle génomique, puis cliquez sur Exécuter ce tutoriel en ligne.
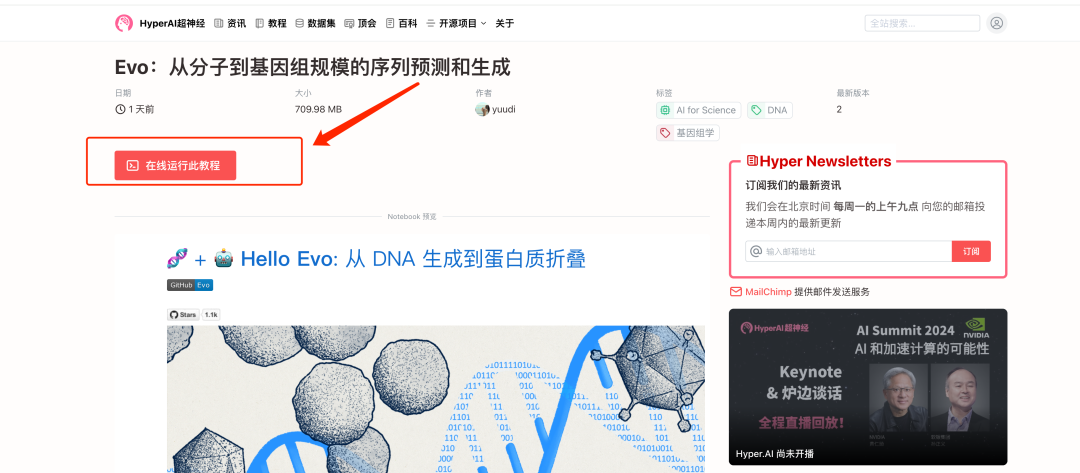
2. Une fois la page affichée, cliquez sur « Cloner » dans le coin supérieur droit pour cloner le didacticiel dans votre propre conteneur.
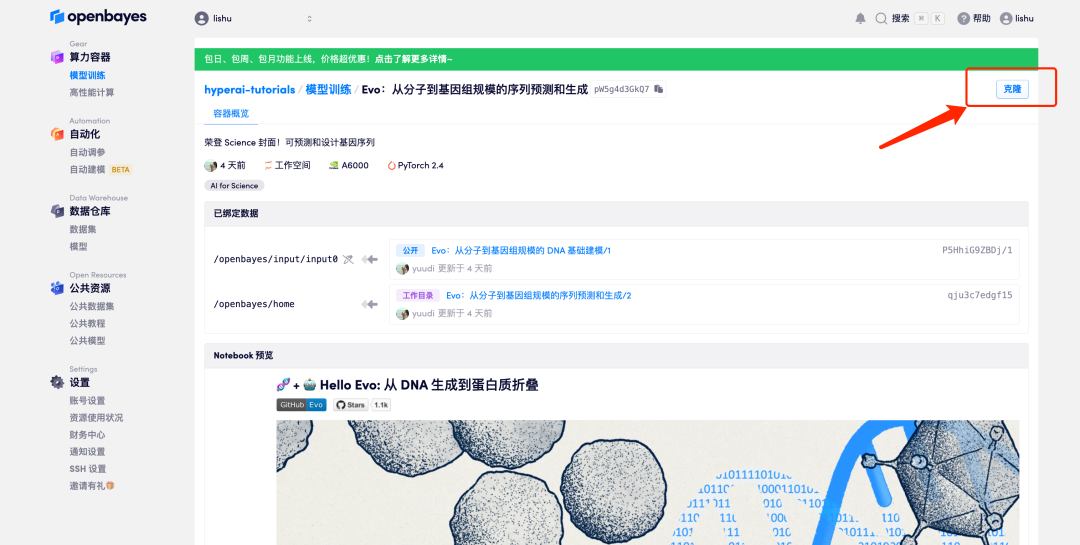
3. Cliquez sur « Suivant : sélectionner le taux de hachage » dans le coin inférieur droit.
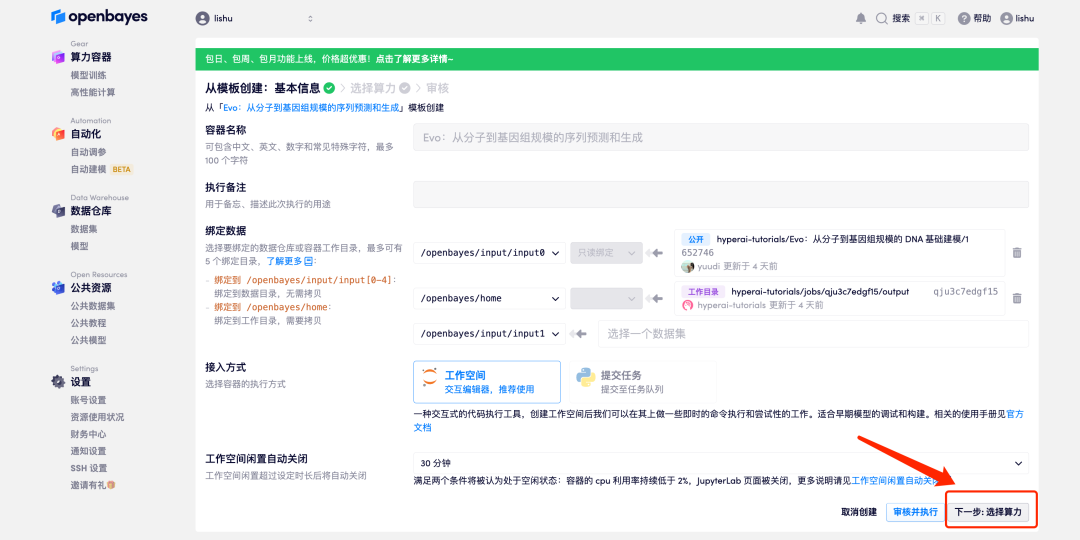
4. Une fois la page affichée, sélectionnez « NVIDIA RTX A6000 », puis « Pay as you go » ou « Daily/Weekly/Monthly Package » selon vos besoins. Après avoir sélectionné l'image « PyTorch », cliquez sur « Suivant : Réviser ». Les nouveaux utilisateurs peuvent s'inscrire en utilisant le lien d'invitation ci-dessous pour obtenir 4 heures de RTX 4090 + 5 heures de temps CPU gratuit !
Lien d'invitation exclusif HyperAI (copier et ouvrir dans le navigateur) :
https://openbayes.com/console/signup?r=Ada0322_QZy7
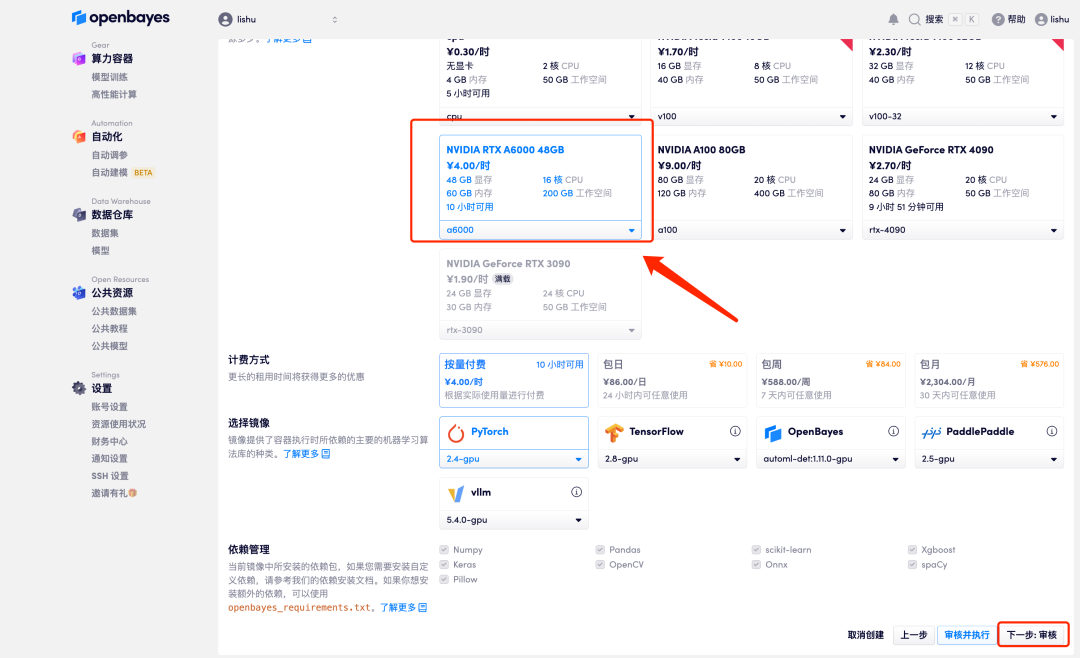
5. Après confirmation, cliquez sur « Continuer » et attendez que les ressources soient allouées. Le premier processus de clonage prendra environ 2 minutes.
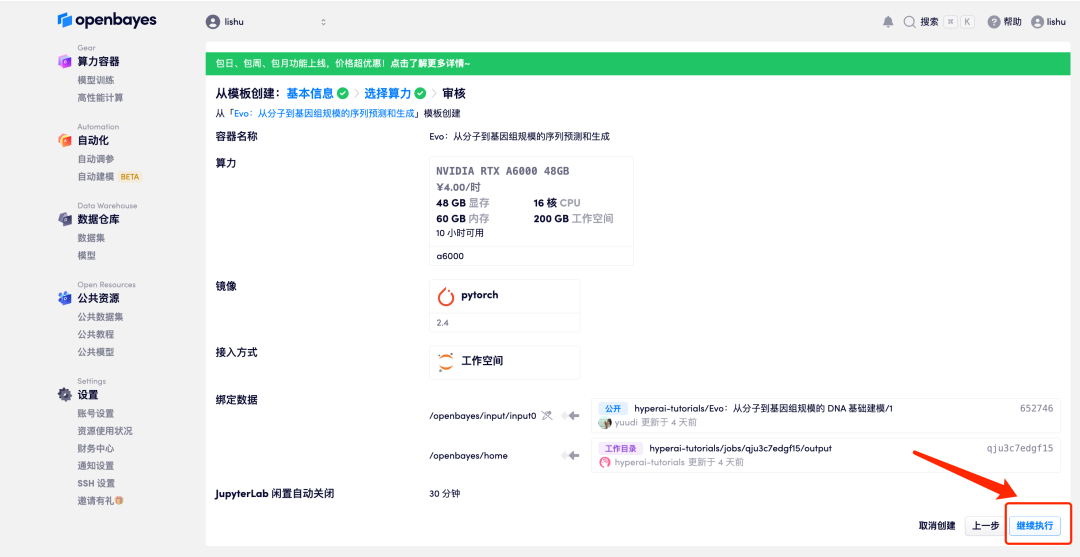
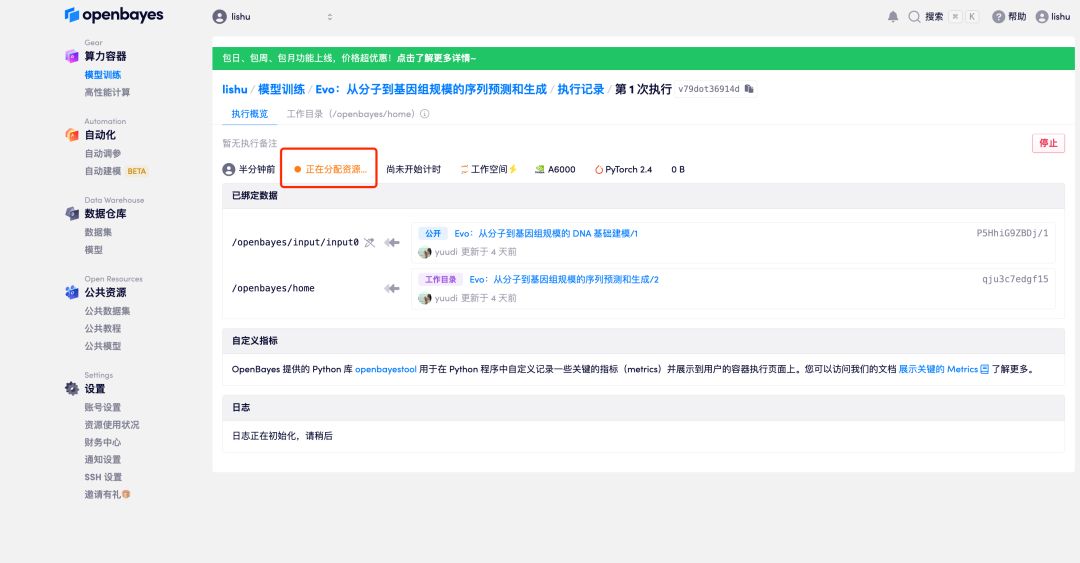
6. Lorsque le statut passe à « En cours d'exécution », cliquez sur l'option « Ouvrir l'espace de travail ».
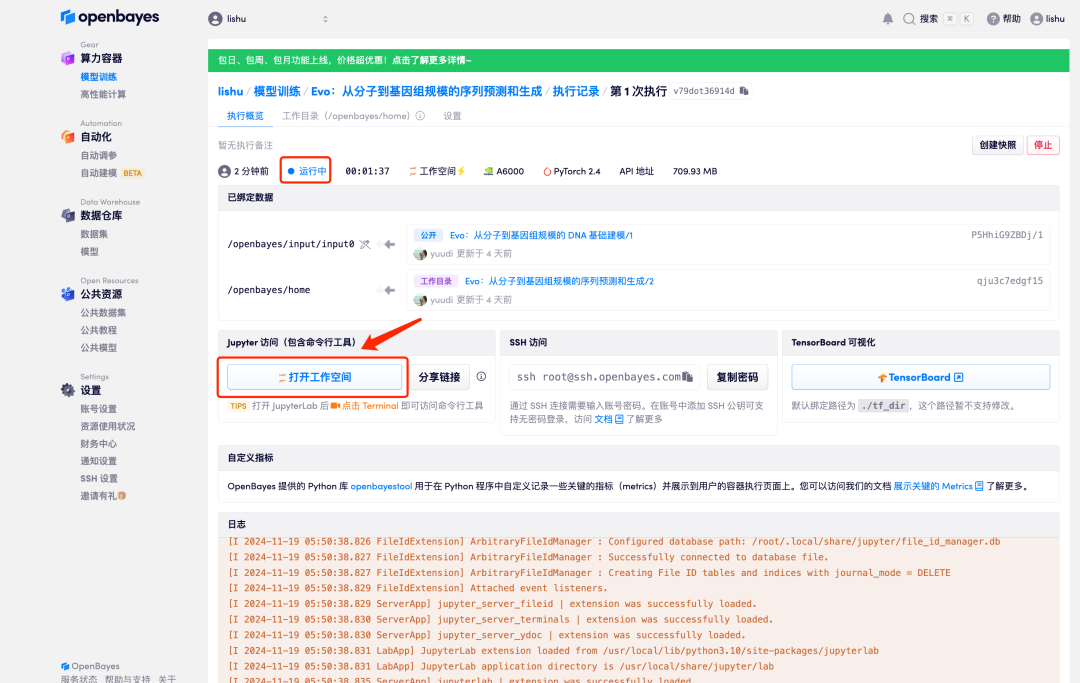
7. Après être entré dans l'espace de travail Jupyter, double-cliquez sur le fichier « README » pour accéder officiellement à la page d'exécution du modèle Evo.
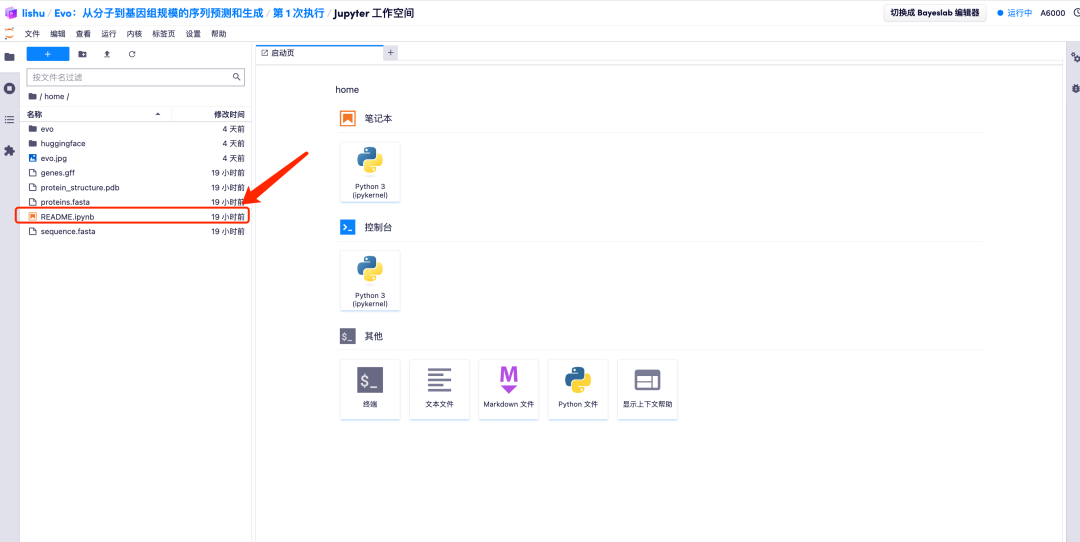
Démonstration d'effet
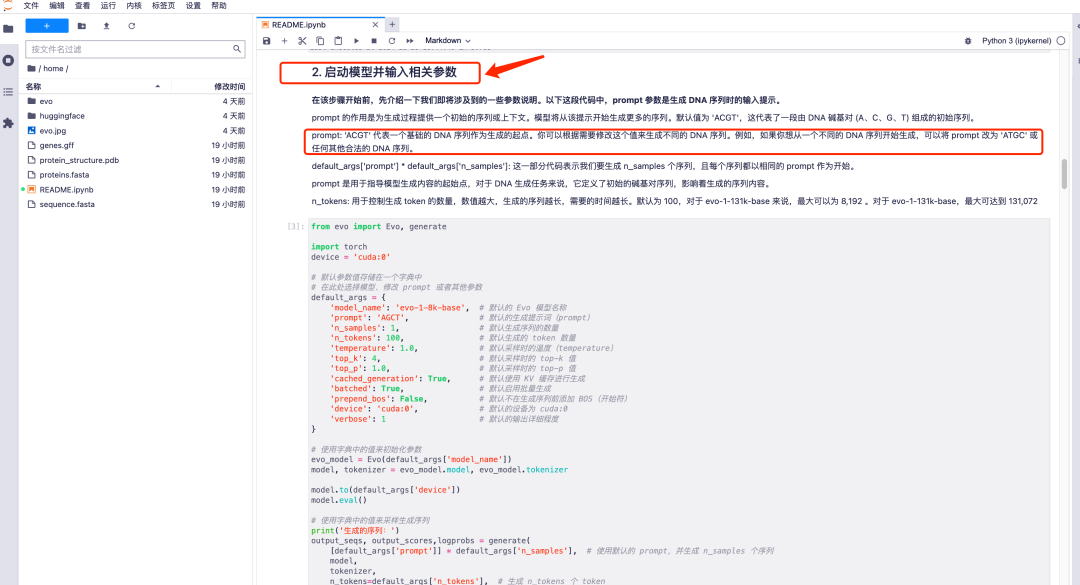
1. Après être entré dans la page d'exécution du modèle Evo, tous les paramètres sont en mode par défaut. Faites défiler jusqu'à « 2. Démarrez le modèle et entrez les paramètres pertinents » et ajustez les valeurs des paramètres d'invite en fonction de vos besoins.Il convient de noter que la valeur par défaut de l'invite est « ACGT », qui représente une séquence initiale composée de paires de bases d'ADN (A, C, G, T).Vous pouvez modifier cette valeur pour générer différentes séquences d'ADN selon vos besoins.
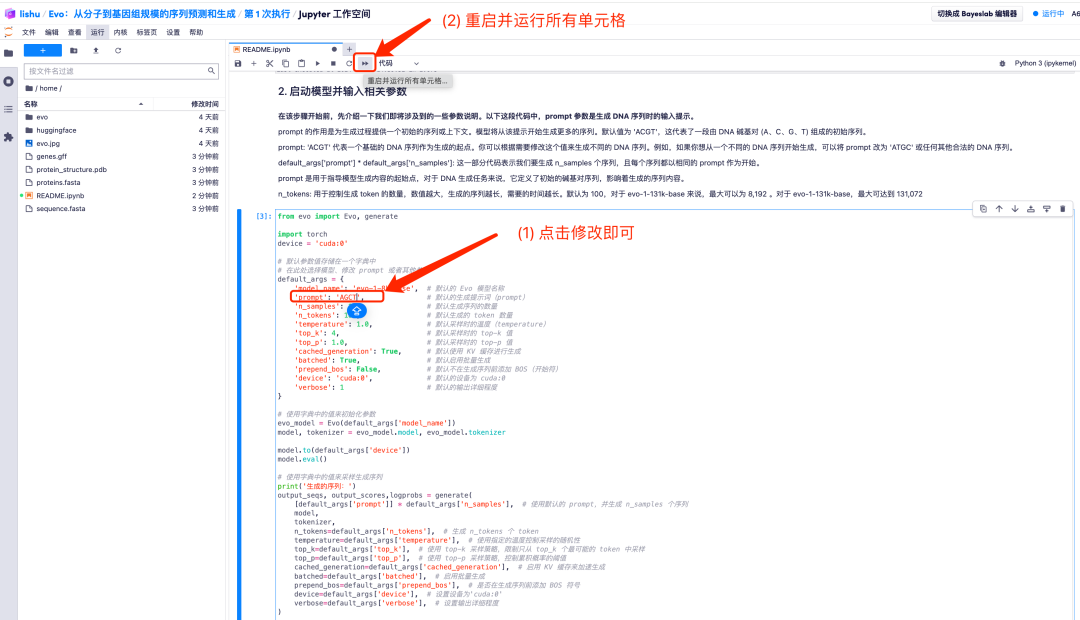
2. Par exemple, modifiez la valeur par défaut de l’invite sur AGCT. Après avoir ajusté les valeurs des paramètres par défaut, cliquez sur l'option « Redémarrer et exécuter toutes les cellules » et sélectionnez « Redémarrer » pour exécuter.
3. Attendez un moment. Lorsque le symbole [*] se transforme en nombre, l’opération est terminée. En bas de « 2. Démarrez le modèle et entrez les paramètres pertinents », vous pouvez voir la séquence générée.
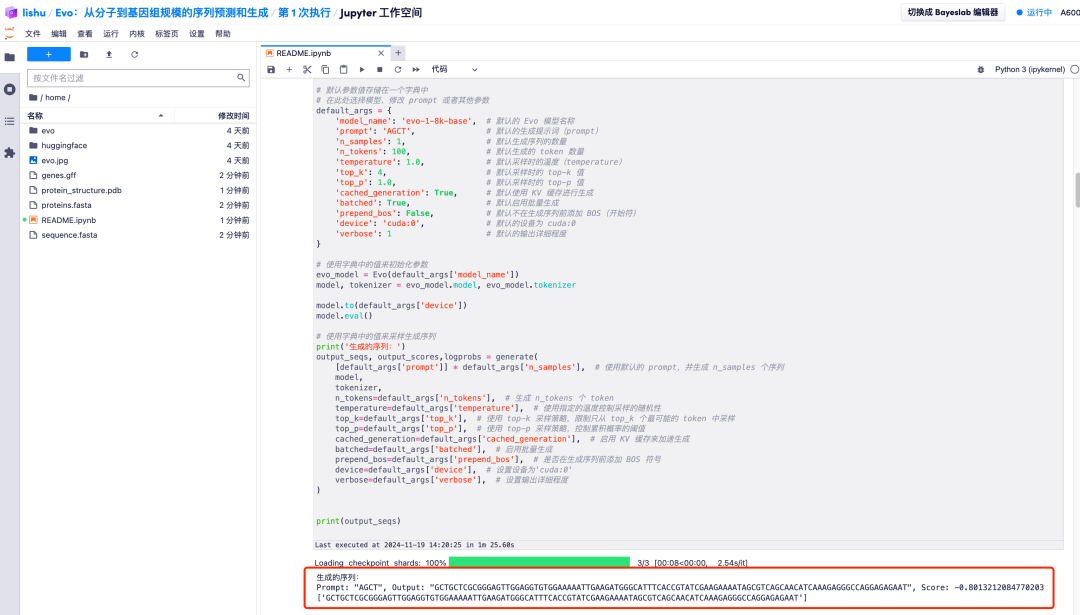
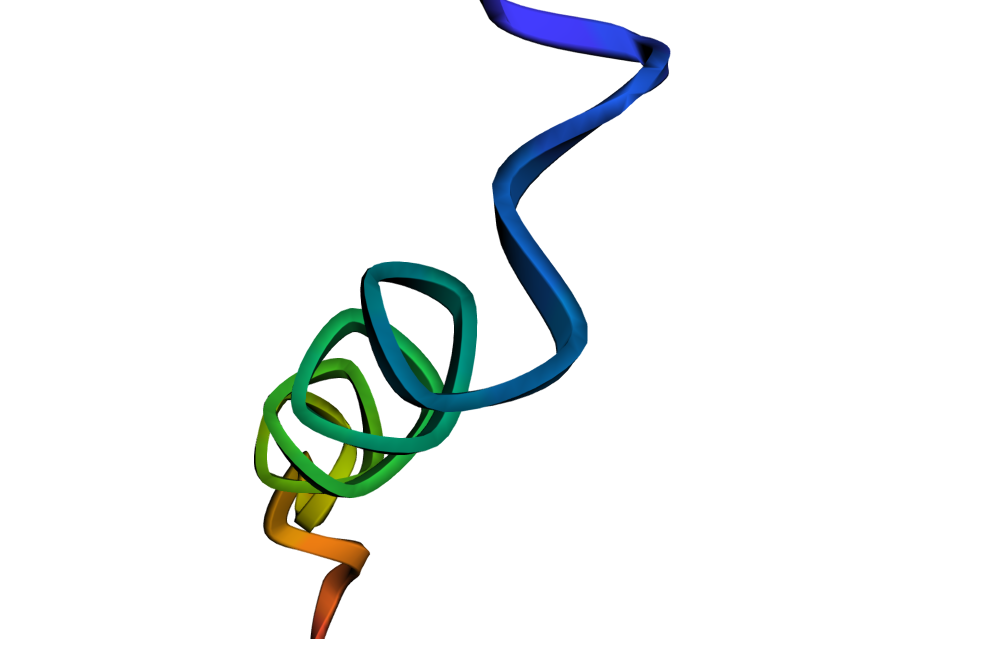
4. De plus, le modèle Evo peut également analyser les séquences d’ADN générées et apprendre la relation co-évolutive entre les séquences codantes et les séquences non codantes. Il peut également prédire les gènes codant pour les protéines à partir de séquences d'ADN, coder et concevoir des systèmes d'ARN, et ainsi prédire la structure de repliement des protéines résultante, qui peut finalement être présentée sous la forme d'une image.
Nous avons créé un « Groupe d'échange de tutoriels de diffusion stable ». Bienvenue aux amis pour rejoindre le groupe pour discuter de divers problèmes techniques et partager les résultats des applications~
Scannez le code QR ci-dessous pour ajouter HyperaiXingXing sur WeChat (ID WeChat : Hyperai01) et notez « SD Tutorial Exchange Group » pour rejoindre le chat de groupe.