Command Palette
Search for a command to run...
Les Nouveaux Résultats De DeepMind Critiqués Car Ils Ressemblent À Une Publicité ? AlphaProteo Peut Concevoir Efficacement Des Liants Protéiques Cibles Avec Une Affinité 300 Fois Supérieure
AlphaFold est très en avance dans le domaine de la prédiction des protéines, et AlphaFold 3 a dépassé les limites et a réussi à prédire toutes les molécules de la vie. Lors de sa sortie, il a été dit que la prédiction et la génération structurées d'AlphaFold 3 accéléreraient le développement de l'AIDD (conception de médicaments basée sur l'intelligence artificielle).
DeepMind a désormais tenté une nouvelle fois de concevoir des médicaments à base d'IA en lançant AlphaProteo pour la conception de nouvelles protéines.
Ce nouveau membre de la famille Alpha a également démontré une amélioration révolutionnaire des capacités. Parmi les sept protéines cibles testées dans l'expérience, AlphaProteo a non seulement obtenu un taux de réussite expérimental plus élevé,L'affinité de liaison est également 3 à 300 fois supérieure à celle des meilleures méthodes existantes.Dans le même temps, les chercheurs l’ont également appliqué à la protéine cible VEGF-A liée au cancer et aux complications diabétiques.Une avancée a été réalisée dans l’utilisation d’outils d’IA dans la conception de conjugués de protéines VEGF-A.
Le criblage à débit moyen en un seul tour génère des liants protéiques « prêts à l'emploi »
Le 5 septembre, heure locale, les chercheurs de DeepMind ont proposé AlphaProteo, un modèle d'apprentissage automatique pour la conception de protéines, qui peut concevoir des liants protéiques cibles avec un taux de réussite élevé, une affinité élevée et sans nécessiter d'intervention humaine complexe. AlphaProteo ne nécessite qu'un seul cycle de criblage à débit moyen et ne nécessite pas d'optimisation supplémentaire pour générer des liants protéiques « prêts à l'emploi » pour de nombreuses applications de recherche.
Structure du modèle : Combinaison de modèles génératifs avec des filtres pour générer efficacement des liants protéiques cibles
AlphaProteo se compose de 2 composants, comme le montre la figure ci-dessous.
Un modèle génératif (Générateur),Formé sur les données de structure et de séquence de la Protein Data Bank (PDB) et l'ensemble de distillation des structures prédites d'AlphaFold pour apprendre comment les molécules se lient les unes aux autres ;Un filtre,Utilisé pour noter les conceptions générées afin de prédire si le liant peut se lier avec succès à la protéine cible dans l'expérience.
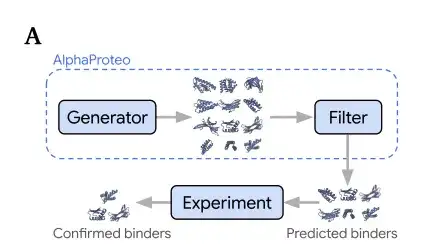
Plus précisément, afin de concevoir des liants de protéines cibles, les chercheurs saisissent d'abord la structure de la protéine cible et sélectionnent les résidus « points chauds » de l'épitope de la protéine cible pour déduire la position de liaison préférée. Le modèle génératif produira la structure et la séquence du liant candidat pour la protéine cible. Le filtre filtrera ces liants prédits dans un ensemble plus petit avant les tests expérimentaux. Lors des tests de référence de simulation par ordinateur, le modèle génératif s'est montré plus performant que les meilleures méthodes existantes.
Conclusion expérimentale : Surpasser les meilleures solutions existantes, avec un taux de réussite élevé et une forte affinité
Les chercheurs ont utilisé AlphaProteo pour concevoir des liants pour huit protéines cibles avec des structures différentes et les ont testés expérimentalement. Parmi elles, on retrouve deux protéines virales liées à l'infection, BHRF1 et le domaine de liaison au récepteur de la protéine de pointe du SARS-CoV-2 SC2RBD, ainsi que cinq protéines liées au cancer, à l'inflammation et aux maladies auto-immunes, IL-7Rɑ, PD-L1, TrkA, IL-17A et VEGF-A, et une protéine TNF liée aux maladies auto-immunes telles que la polyarthrite rhumatoïde.
Comme le montre la figure ci-dessous,Les sept liants protéiques cibles générés par AlphaProteo ont pu se lier étroitement à la protéine cible lorsqu'ils ont été testés expérimentalement.Cependant, il n’a pas été possible de concevoir des liants efficaces pour la huitième cible, le TNF.
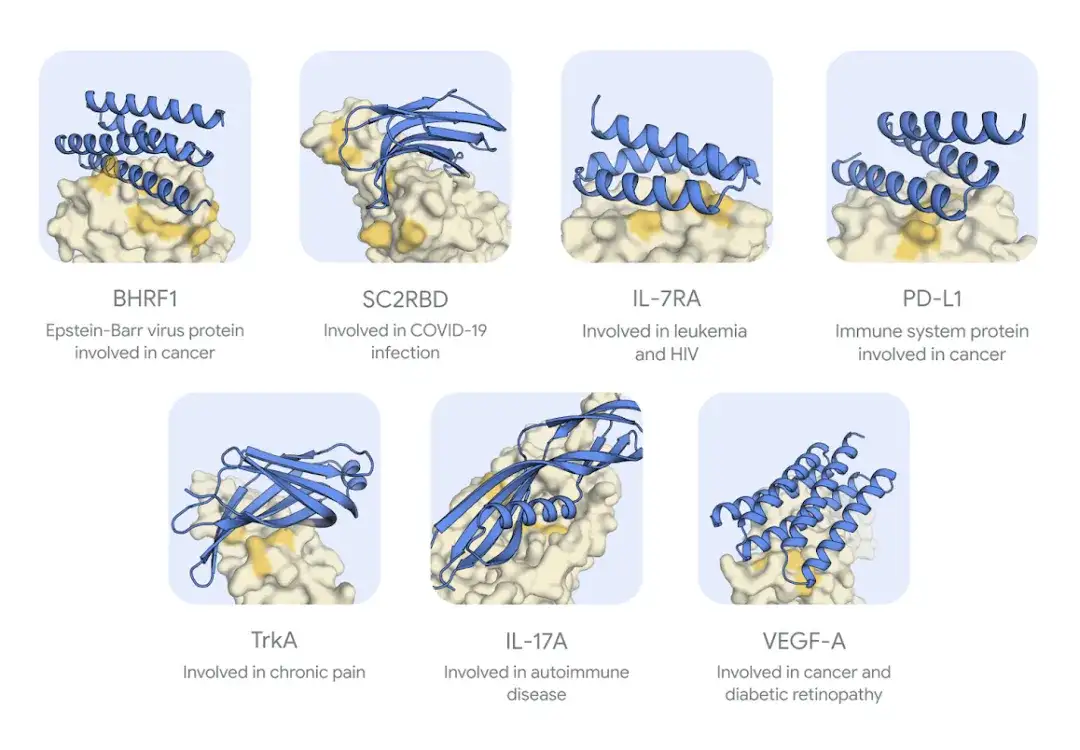
* Le bleu indique le liant, le jaune indique la cible protéique, le jaune foncé est la région de liaison attendue
De plus, comme le montre la figure ci-dessous, les chercheurs ont comparé AlphaProteo avec d’autres méthodes de conception pour comparer le taux de réussite in vitro. s'avérer,Lors de tests en laboratoire humide, les candidats liants 9%-88% ont pu se lier avec succès à la protéine cible à des niveaux 5 à 100 fois plus élevés que les autres méthodes.
Parmi elles, pour la protéine virale BHRF1, les molécules candidates générées par AlphaProteo ont un taux de réussite de liaison de 88% avec la protéine cible, et la force de liaison est 10 fois supérieure à celle de la meilleure méthode de conception existante ; pour la protéine TrkA, le nouveau liant est plus fort que le liant le mieux conçu qui a été optimisé pour cette protéine après plusieurs séries d'expériences.
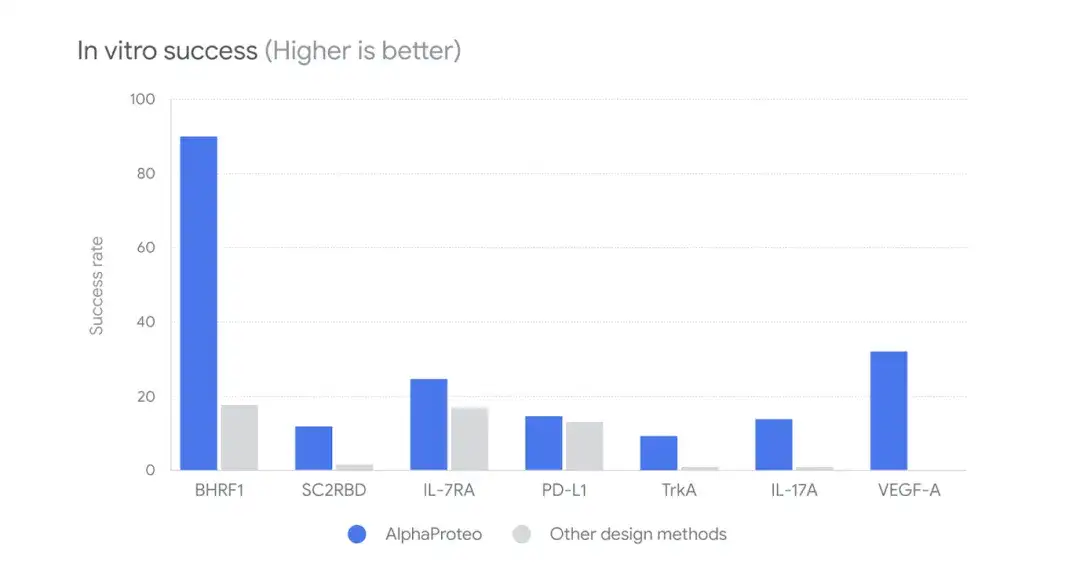
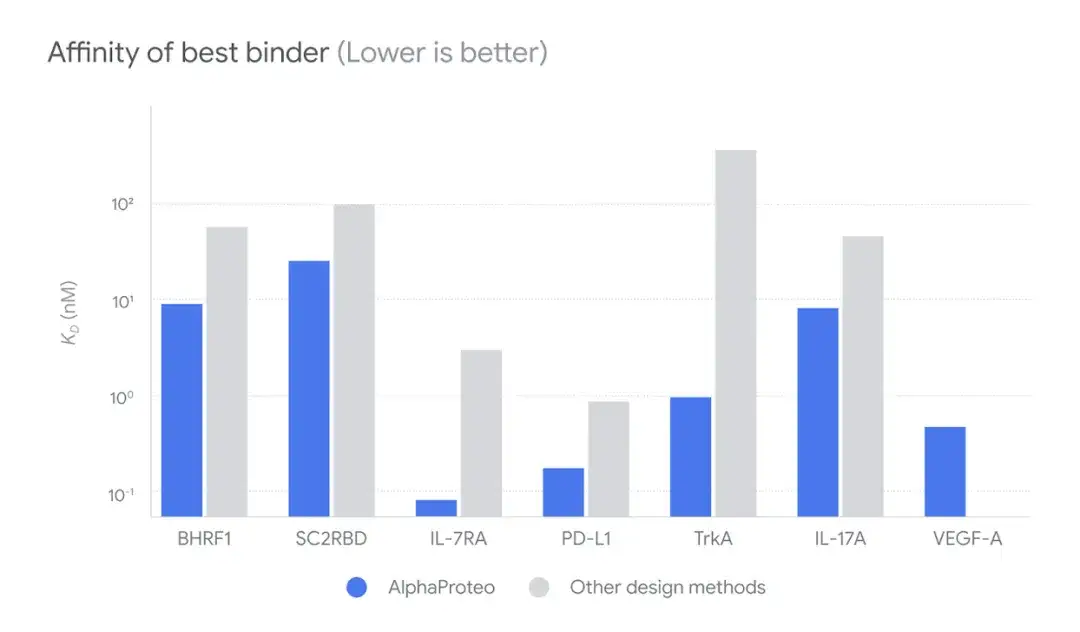
De plus, les chercheurs ont exploré la meilleure affinité des liants conçus par AlphaProteo pour cibler les protéines par rapport à d’autres méthodes de conception sans optimisation expérimentale. s'avérer,Les liants générés par AlphaProteo ont des affinités de liaison environ 3 à 300 fois supérieures à celles des liants conçus à l'aide des meilleures méthodes disponibles.
* Plus l'affinité est grande, plus la liaison est étroite
Les chercheurs ont exploré plus en détail les similitudes entre les liants plus forts de la protéine SC2RBD et VEGF-A et ceux conçus par AlphaProteo et ont découvert queLes interactions de liaison de ces liants plus forts étaient similaires à celles prédites par AlphaProteo, ce qui indique la fiabilité d'AlphaProteo.
De plus, les chercheurs ont également confirmé que ces liants ont de réelles fonctions biologiques. Par exemple, il a été démontré que certains liants SC2RBD empêchent le SARS-CoV-2 et ses variantes d’infecter les cellules. Plus important encore, AlphaProteo a une large applicabilité et ne nécessite pas d’expériences de criblage à haut débit ou d’optimisation d’affinité, ce qui peut réduire considérablement le temps expérimental initial pour la conception de liants protéiques.
Collaborer avec le plus grand laboratoire de recherche biomédicale d'Europe
Parmi les premières expériences visant à tester le nouveau système de conception de protéines AlphaProteo figurent trois équipes du Francis Crick Institute.
Le Francis Crick Institute est situé à Londres, en Angleterre. Il s’agit actuellement du plus grand laboratoire de recherche biomédicale d’Europe et d’un centre de recherche au Royaume-Uni. Il a été lancé conjointement par l’Institut national de recherche médicale (NIMR) et Cancer Research UK pour un coût d’environ 700 millions de livres sterling.
À ce jour, l’Institut Crick compte plus de 2 000 chercheurs et plus de 100 groupes de recherche. En outre, l’Institut est un partenariat unique entre six des plus grandes organisations de recherche biomédicale au monde, dont le Medical Research Council, Cancer Research UK, le Wellcome Trust, l’University College London, l’Imperial College London et le King’s College London.
Adresse du site officiel du Francis Crick Institute :
* https://www.crick.ac.uk/
Les derniers résultats de Deepmind suscitent des critiques mitigées
Importance de la recherche par rapport à l'open source
Bien que la grande valeur d’AlphaProteo dans la recherche pratique soit évidente pour tous, de nombreux internautes se demandent davantage si cet outil est open source. Un internaute a demandé : « Est-ce open source ? Sinon, ce serait trop ennuyeux. »
D'autres internautes ont déclaré :AlphaProteo est plus important pour sauver des vies que son caractère open source.Et a exprimé son optimisme quant à son potentiel de développement.
Les détails méthodologiques sont trop peu nombreux, cela ressemble plus à une publicité qu'à un article
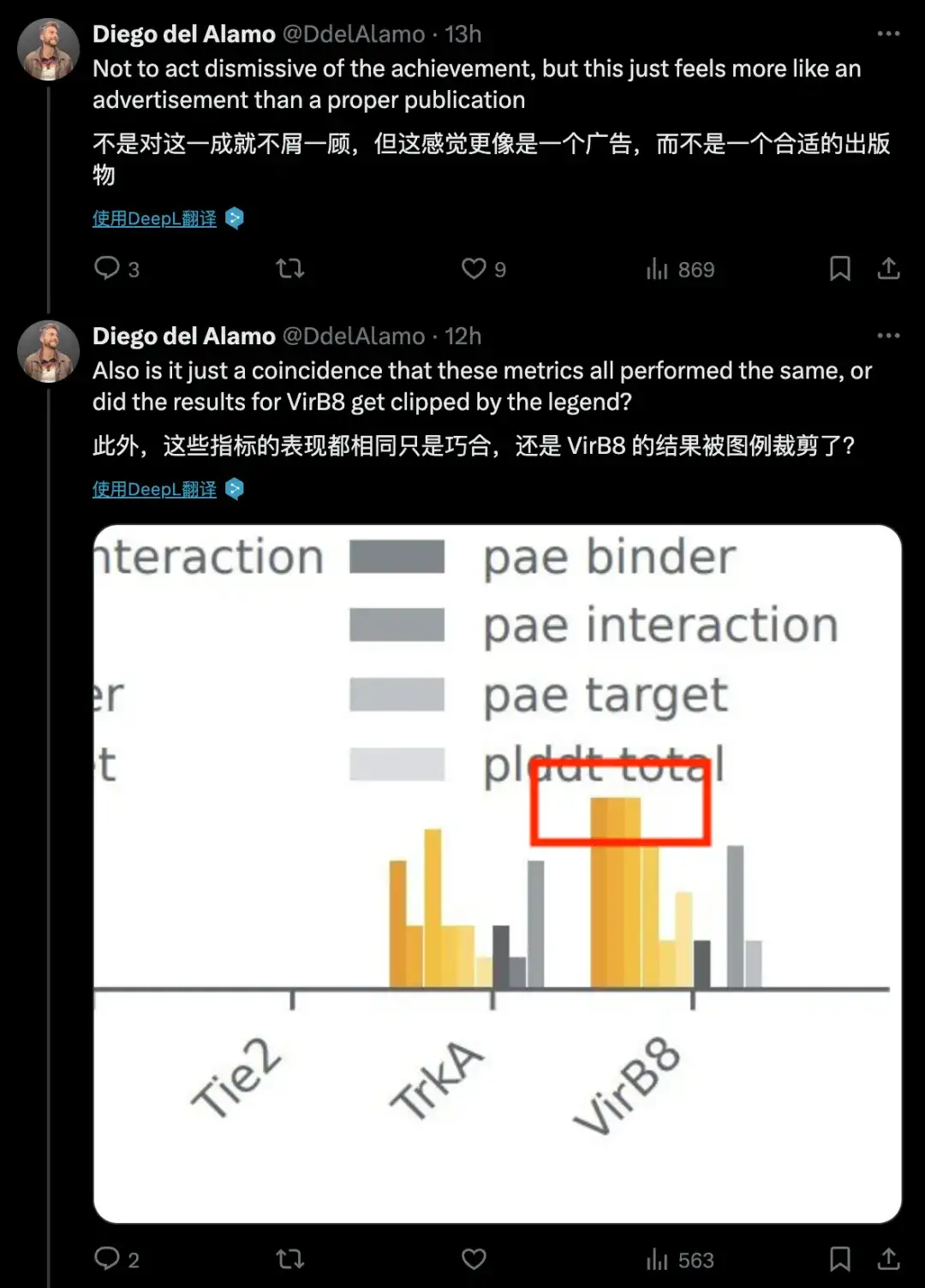
L'équipe DeepMind a rédigé un article sur le modèle, mais il contient très peu de détails sur les méthodes du modèle, voire presque aucun détail. Un scientifique d'une importante société pharmaceutique et de santé mondiale a publié trois mises à jour consécutives en déclarant : « Il y a trop peu de détails méthodologiques dans ce livre blanc » et « Cela ressemble plus à une publicité qu'à une véritable publication », et a remis en question les mêmes indicateurs de légende dans le document.
Un autre docteur en biologie du MIT a non seulement réitéré l'importance de la science open source, mais a également critiqué la démarche de DeepMind : « DeepMind est allé trop loin, et je veux vraiment savoir comment cela fonctionne. »
DeepMind ouvre un nouveau champ de bataille et complète AlphaFold
La « mise à niveau et la lutte contre les monstres » de DeepMind pour l’industrie biopharmaceutique ont ouvert un nouveau champ de bataille. Cela peut-il devenir un coup de pouce pour accélérer la mise en œuvre de la conception de médicaments par l’IA ?
Comme nous le savons tous, AlphaFold 3 est déjà capable de prédire les structures et les interactions de toutes les molécules vivantes (protéines, ADN, ARN, ligands, etc.) avec une précision ultra élevée. En ce qui concerne l’orientation future de son itération, les initiés du secteur prédisent qu’il pourrait y avoir des améliorations dans la prédiction des informations cinétiques.L'AlphaProteo sorti cette fois-ci peut, dans une certaine mesure, se compléter et se promouvoir mutuellement avec AlphaFold.
d'une part,La conception des protéines doit partir de la fonction et concevoir des structures protéiques nouvelles ou améliorées pour atteindre des fonctions biologiques ou chimiques spécifiques. Elle dépend donc fortement de la compréhension de la relation entre la structure et la fonction des protéines.d'autre part,La prédiction des protéines peut fournir des informations structurelles clés et guider la conception. Dans le même temps, les protéines conçues peuvent également aider à améliorer les modèles de prédiction et à accroître la précision des prédictions dans des aspects tels que le repliement et la dynamique des protéines.
DeepMind a mentionné dans son introduction à AlphaProteo que cette innovation pourrait accélérer la compréhension des processus biologiques et aider à la découverte de nouveaux médicaments, au développement de biocapteurs, et plus encore. À l’avenir, l’entreprise travaillera avec la communauté scientifique pour utiliser AlphaProteo afin de répondre à des questions biologiques de grande envergure et de comprendre ses limites. Parallèlement, DeepMind explore également ses applications de conception de médicaments avec Isomorphic Labs.
Isomorphic Labs est né de DeepMind et se concentre sur le domaine de la médecine de l'IA. Sa mission est d’utiliser l’intelligence artificielle et les méthodes d’apprentissage automatique pour accélérer et améliorer le processus de découverte de médicaments afin de trouver des traitements pour certaines des maladies les plus dévastatrices de l’humanité.
Dans une certaine mesure, Isomorphic Labs est également le bras droit de DeepMind dans le domaine de la médecine de l'IA, qui peut explorer l'application de ses derniers résultats en matière de conception de médicaments de manière ciblée pour la première fois. L'équipe est également apparue dans la recherche d'AlphaFold 3. Nous espérons qu'avec le soutien de DeepMind, cela apportera de bonnes nouvelles pour la mise en œuvre de la conception de médicaments par IA.
Enfin, je vous recommande une activité de partage académique !
La troisième diffusion en direct de Meet AI4S a invité Zhou Ziyi, chercheur postdoctoral à l'Institut des sciences naturelles de l'Université Jiao Tong de Shanghai et au Centre national de mathématiques appliquées de Shanghai. Le 25 septembre, le Dr Zhou Ziyi partagera avec tout le monde la méthode d'apprentissage par petits échantillons du modèle de langage protéique sous forme de diffusion en direct en ligne et explorera de nouvelles idées pour l'évolution dirigée assistée par l'IA. Cliquez pour prendre rendez-vous pour regarder la diffusion en direct !








