Command Palette
Search for a command to run...
La Dernière Application De SAM 2 Est Désormais Disponible ! L'équipe De l'Université d'Oxford Publie Medical SAM 2, Actualisant La Liste SOTA De Segmentation d'images Médicales
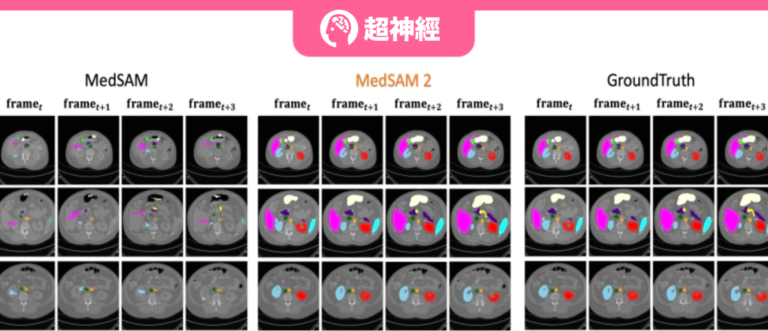
En avril 2023, Meta a publié le Segment Anything Model (SAM), prétendant pouvoir « tout segmenter ». Ce fut comme une bombe qui a secoué tout le domaine de la vision par ordinateur et a même été considéré par beaucoup comme une recherche qui a subverti les tâches traditionnelles du CV.
Après plus d'un an,Meta a publié une autre mise à jour importante - SAM 2, qui peut fournir une segmentation d'objets suggestible en temps réel pour les images statiques et le contenu vidéo dynamique, intégrant les fonctions de segmentation d'image et de vidéo dans le même système.Comme vous pouvez l’imaginer, cette forte force a permis à l’industrie d’accélérer l’exploration des applications de SAM dans différents domaines, notamment dans le domaine de la segmentation d’images médicales. De nombreux laboratoires et équipes de recherche universitaires le considèrent déjà comme le seul choix pour les modèles de segmentation d’images médicales.
La segmentation d'images médicales consiste à segmenter les parties ayant des significations particulières dans les images médicales et à extraire les caractéristiques pertinentes, fournissant ainsi une base fiable pour le diagnostic clinique, la recherche pathologique, etc.
Ces dernières années, avec les progrès continus de la technologie d'apprentissage en profondeur, la segmentation basée sur des modèles de réseaux neuronaux est progressivement devenue la méthode dominante de segmentation d'images médicales, et les méthodes de segmentation automatisées ont considérablement amélioré l'efficacité et la précision. Cependant,Compte tenu de la particularité du domaine de la segmentation des images médicales, certains défis doivent encore être relevés.
La première est la généralisation du modèle.Les modèles entraînés pour des cibles spécifiques (telles que des organes ou des tissus) sont difficiles à adapter à d’autres cibles, il est donc souvent nécessaire de redévelopper des modèles correspondants pour différentes cibles de segmentation ;Le deuxième problème concerne les différences de données.De nombreux cadres d’apprentissage profond standard développés pour la vision par ordinateur sont conçus pour les images 2D, mais dans l’imagerie médicale, les données sont généralement au format 3D, comme les images CT, IRM et échographiques. Cette différence pose sans aucun doute de gros problèmes pour la formation des modèles.
Afin de résoudre les problèmes ci-dessus,L’équipe de l’Université d’Oxford a développé un modèle de segmentation d’images médicales appelé Medical SAM 2 (MedSAM-2).Le modèle est conçu sur la base du framework SAM 2 et traite les images médicales comme des vidéos. Il fonctionne non seulement bien dans les tâches de segmentation d'images médicales 3D, mais débloque également une nouvelle capacité de segmentation à invite unique. L'utilisateur n'a qu'à fournir un indice pour un nouvel objet spécifique, et la segmentation des objets similaires dans les images suivantes peut être automatiquement complétée par le modèle sans autre saisie.
Les articles et résultats pertinents ont été publiés sur la plateforme de pré-impression arXiv sous le titre « Medical SAM 2 : Segmenter les images médicales en vidéo via Segment Anything Model 2 ».
Points saillants de la recherche :
* L'équipe a été la pionnière du modèle de segmentation d'images médicales MedSAM-2 basé sur SAM 2
* L'équipe a adopté un nouveau concept « d'images médicales sous forme de vidéos », libérant ainsi la « fonction de segmentation à invite unique »

Adresse du document :
https://arxiv.org/pdf/2408.00874
Téléchargement direct de l'ensemble de données de segmentation vidéo SA-V :
Ensemble de données de segmentation médicale d'échantillon SAM 2 :
Le projet open source « awesome-ai4s » rassemble plus de 100 interprétations d'articles AI4S et fournit des ensembles de données et des outils massifs :
https://github.com/hyperai/awesome-ai4s
Ensemble de données : conception de la classification, évaluation complète
L'équipe a mené des expériences sur cinq ensembles de données de segmentation d'images médicales différents en utilisant des indices de masque générés automatiquement, qui ont été divisés en deux catégories :
La première catégorie vise à évaluer la performance générale de la segmentation,L’équipe a choisi la tâche de segmentation multi-organes abdominale et a sélectionné l’ensemble de données BTCV, qui contient 12 structures anatomiques.
La deuxième catégorie vise à évaluer la capacité de généralisation du modèle à travers différentes modalités d’imagerie.Les chercheurs ont utilisé l’ensemble de données REFUGE2 pour la segmentation des images du disque optique et de la cupule optique ; l'ensemble de données BraTs 2021 pour la segmentation des tumeurs cérébrales dans les examens IRM ; le benchmark TNMIX a été utilisé pour segmenter les nodules thyroïdiens dans les images échographiques, qui se composent de 4 554 images de TNSCUI et de 637 images de DDTI ; et l'ensemble de données ISIC 2019 a été utilisé pour segmenter les lésions cutanées en mélanome ou naevus.
De plus, l'équipe a mis en place 10 tâches supplémentaires de segmentation d'images 2D pour évaluer davantage la capacité de segmentation à indice unique du modèle en utilisant différents types d'indices. Plus précisément, les ensembles de données KiTS23, ATLAS23, TDSC et WBC utilisent la technologie d’indication de points ; les ensembles de données SegRap, CrossM23 et REFUGE utilisent l'indication BBox (boîte englobante) ; les ensembles de données CadVidSet, STAR et ToothFairy utilisent des indications de masque.
Architecture du modèle : traitement de segmentation efficace pour les images médicales de différentes dimensions
L'architecture de MedSAM-2 est fondamentalement similaire à celle de SAM 2, mais l'équipe de recherche a également construit un module de traitement et un pipeline uniques et efficaces, combinés à une banque de mémoire de confiance et à une stratégie de ramassage pondérée pour garantir techniquement les capacités du modèle.
Spécifiquement,L'architecture de MedSAM-2 est illustrée dans la figure ci-dessous.Inclure:
* Image Encoder, résume l'entrée dans l'intégration
* Attention mémoire, qui utilise les mémoires stockées dans la banque de mémoire pour ajuster l'intégration d'entrée
* Décodeur de mémoire, qui fait abstraction de l'intégration de trame prédite
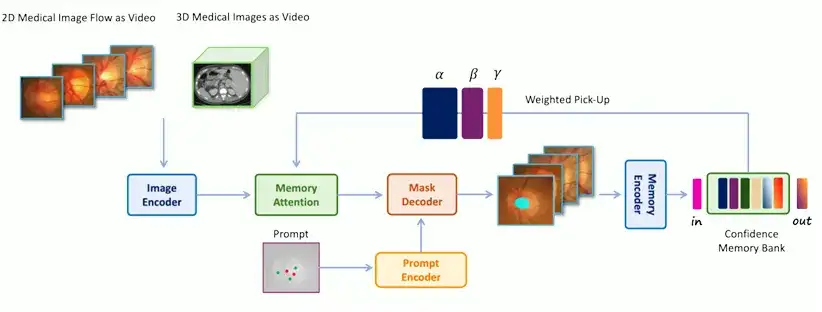
L'encodeur et le décodeur du réseau sont similaires à ceux de SAM. L'encodeur se compose d'un transformateur visuel hiérarchique et le décodeur comprend un transformateur bidirectionnel léger qui intègre l'intégration d'invites et l'intégration d'images, où l'intégration d'invites est générée par l'encodeur d'invites ; Le composant d'attention de la mémoire se compose d'une série de blocs d'attention empilés, chacun contenant un bloc d'auto-attention et un mécanisme d'attention croisée.
Il convient de noter queUne innovation importante de MedSAM-2 est de traiter le traitement des images médicales comme une segmentation vidéo.C'est la clé pour améliorer les performances de la segmentation d'images médicales 3D et débloquer la « fonction de segmentation à invite unique ». À cette fin, l’équipe a également développé deux processus d’exploitation différents pour les images médicales 2D et 3D respectivement afin d’effectuer un traitement de segmentation efficace pour les images médicales de différentes dimensions.
Pour le traitement d'images médicales 3D,Étant donné qu’il existe une forte corrélation temporelle entre les tranches adjacentes dans les images médicales 3D, la méthode de traitement est similaire à celle des données vidéo. Le système de stockage d'origine de SAM 2 est utilisé pour récupérer les tranches précédentes et leurs prédictions correspondantes pour la segmentation continue des tranches. L'intégration de l'image d'entrée est ensuite améliorée grâce au mécanisme d'attention de la mémoire, et les résultats de segmentation sont ajoutés à la zone de stockage pour aider à la segmentation des tranches suivantes.
Pour le traitement d'images médicales 2D,La méthode de traitement est différente de la file d'attente temporelle premier entré, premier sorti utilisée dans SAM 2. Au lieu de cela, un groupe d'images médicales contenant le même organe ou tissu est regroupé dans un « flux d'images médicales » et une zone de stockage « confiance d'abord » est utilisée pour stocker les modèles du modèle. La confiance est calculée en fonction de la probabilité prédite par le modèle et des contraintes de diversité d'images sont mises en œuvre. Une stratégie de sélection pondérée est adoptée lors de la fusion de l'intégration de l'image d'entrée et des informations de la région mémoire. Pendant la phase de formation, une tête d'étalonnage est utilisée pour garantir que les prédictions du modèle sont plus précises. En fin de compte, nous pouvons réaliser une segmentation automatique des cibles avec un seul échantillon sans aucune association temporelle.
Résultats expérimentaux : MedSAM-2 est en tête en termes de performances et de capacité de généralisation
L'équipe de recherche a utilisé IoU (Intersection over Union) et Dice Score pour évaluer les performances du modèle dans la segmentation d'images médicales et a introduit la métrique Hausdorff Distance (HD95) pour garantir l'exactitude de l'évaluation des performances.
*LoU, également connu sous le nom d'indice de Jaccard, est une mesure utilisée pour évaluer la précision d'un détecteur d'objets sur un ensemble de données spécifique.
* Dice Score, également connu sous le nom de Dice Coefficient, est un outil statistique permettant de comparer les similitudes entre deux échantillons.
* La mesure de la distance de Hausdorff (HD95) est une mesure utilisée pour déterminer le degré de différence entre deux ensembles de points. Il est souvent utilisé pour évaluer la précision des limites d'objets dans les tâches de segmentation d'images et est particulièrement efficace pour quantifier la distance du pire des cas entre la segmentation prédite et la limite réelle.
Tout d’abord, l’équipe a comparé MedSAM-2 à une gamme de méthodes de segmentation d’images médicales SOTA, y compris des tâches de segmentation pour les images médicales 2D et 3D. Pour les images médicales 3D, l’indice est fourni de manière aléatoire avec une probabilité de 0,25 ; pour les images médicales 2D, la probabilité est de 0,3.
Afin d'évaluer les performances générales du modèle proposé sur des images médicales 3D,L'équipe a comparé MedSAM-2 avec des méthodes de segmentation avancées établies sur l'ensemble de données de segmentation multi-organes BTCV, y compris les modèles bien connus nnUNET, TransUNet, UNetr, Swin-UNetr et les modèles basés sur la diffusion (tels que EnsDiff, SegDiff et MedSegDiff). En outre, l’équipe a également mené des évaluations comparatives sur des modèles de segmentation interactifs tels que le SAM original, le MedSAM entièrement affiné, le SAMed, le SAM-Med2D, le SAM-U, le VMN et le FCFI. La performance est quantifiée à l'aide du Dice Score, et les résultats sont présentés dans la figure suivante :
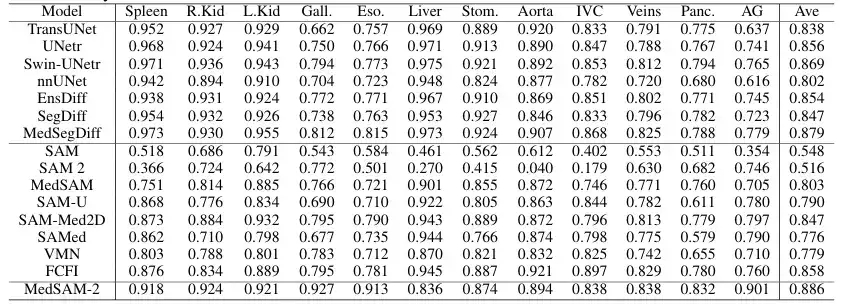
Les résultats ont montré que MedSAM-2 représentait une amélioration significative par rapport aux précédents SAM et MedSAM. Sur l'ensemble de données BTCV, MedSAM-2 obtient d'excellentes performances sur la tâche de segmentation multi-organes, atteignant un score Dice final de 88,57%. Parmi les modèles interactifs, MedSAM-2 maintient sa position de leader, surpassant le précédent modèle interactif leader Med-SA de 2,78%. Tous ces modèles interactifs nécessitent des invites pour chaque image, tandis que MedSAM-2 obtient de meilleurs résultats avec moins d'invites.
Dans la tâche de segmentation d'images médicales 2D,L’équipe a comparé MedSAM-2 avec des méthodes adaptées à des tâches spécifiques sur différentes modalités d’image. Plus précisément, pour la segmentation de la cupule optique, elle a été comparée à ResUnet et BEAL ; pour la segmentation des tumeurs cérébrales, il a été comparé à TransBTS et SwinBTS ; pour la segmentation des nodules thyroïdiens, elle a été comparée à MTSeg et UltraUNet ; pour la segmentation des lésions cutanées, il a été comparé à FAT-Net et BAT. En outre, l’équipe a également évalué le modèle interactif, et les résultats sont présentés dans la figure ci-dessous :
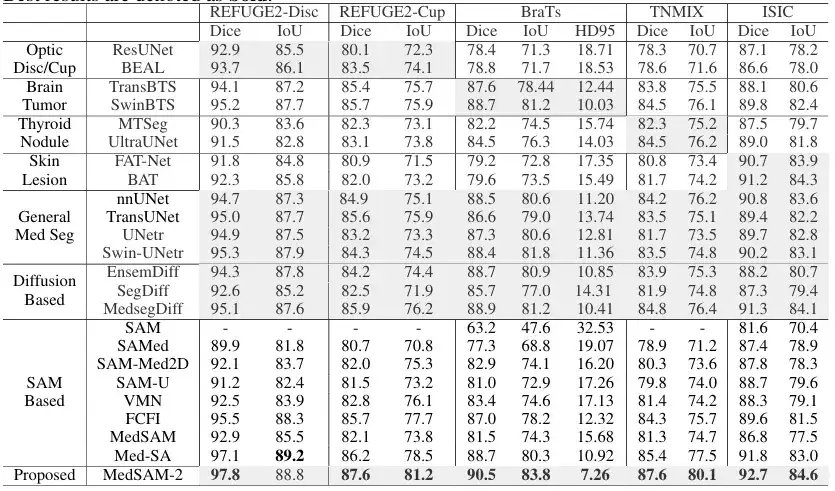
Les résultats montrent que MedSAM-2 surpasse toutes les autres méthodes dans 5 tâches différentes, démontrant son excellente capacité de généralisation dans différentes tâches de segmentation d'images médicales. Plus précisément, MedSAM-2 a obtenu une amélioration de 2,0% sur les cupules optiques, de 1,6% sur les tumeurs cérébrales et de 2,8% sur les nodules thyroïdiens. Dans la comparaison des modèles interactifs, MedSAM-2 conserve toujours ses performances de pointe.
enfin,L’équipe a également évalué les performances de MedSAM-2 lorsqu’un seul signal lui était donné.Et il n’y a pas de lien clair entre les images séquentielles, ce qui vérifie davantage la capacité de MedSAM-2 à segmenter les signaux en un seul coup. Plus précisément, l’équipe a comparé MedSAM-2 avec PANet, ALPNeu, SENet et UniverSeg, qui ont tous été testés avec une seule invite. De plus, l’équipe a comparé MedSAM-2 avec des modèles à lentille unique tels que DAT, ProbONE, HyperSegNas et One-prompt.
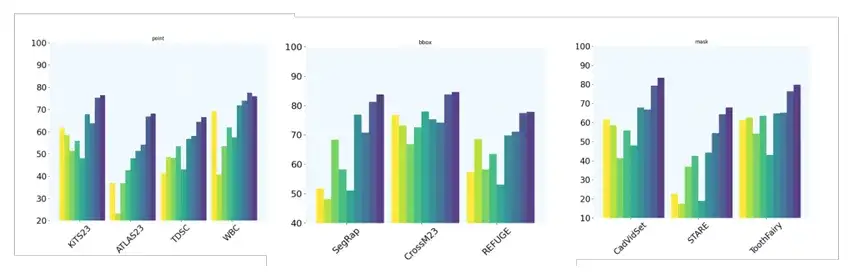
Les résultats montrent que MedSAM-2 démontre de solides capacités de généralisation dans une variété de tâches, et obtient même de bons résultats par rapport au One-prompt intensivement formé, en prenant du retard dans seulement 1 des 10 tâches. De plus, dans les scénarios où toutes les méthodes fournissent des masques, MedSAM-2 montre un avantage plus évident, dépassant souvent la deuxième place de 3,1% en moyenne, ce qui constitue le plus grand écart parmi tous les paramètres d'invite.
SAM contribue à la recherche sur la segmentation des images médicales en plein essor
La publication de cet article peut être considérée comme une autre exploration approfondie du potentiel du SAM et du SAM 2 dans le domaine médical. Il fournit une nouvelle idée et une nouvelle méthode pour le domaine de la segmentation d'images médicales, montrant notamment un grand potentiel et une grande valeur dans les applications cliniques. Il peut réduire considérablement la charge de travail de la segmentation des images médicales et améliorer l'efficacité et la précision de la segmentation des images médicales.
Ce qui mérite d’être mentionné davantage, c’est que, comme mentionné au début de l’article,De nombreux laboratoires et équipes académiques explorent le potentiel du SAM.Dans le domaine de la segmentation des images médicales, il y a bien plus que l’équipe de l’Université d’Oxford mentionnée dans cet article.
Par coïncidence, peu de temps après la sortie de SAM, l’équipe du professeur Ni Dong de l’École d’ingénierie biomédicale de l’École de médecine de l’Université de Shenzhen, en collaboration avec l’Université d’Oxford, l’ETH Zurich, l’Université du Zhejiang, l’Hôpital populaire de Shenzhen et Duying Medical, a lancé des expériences et des évaluations complètes et multi-angles sur l’application de SAM dans les tâches d’imagerie médicale. Les articles et résultats pertinents ont été publiés dans la revue internationale de référence dans le domaine de l'analyse d'images médicales, « Medical Image Analysis », sous le titre « Segment Anything Model for Medical Images ? »

Dans le cadre de la recherche de cet article, l'équipe concernée a finalement construit un ensemble de données de segmentation d'images médicales à très grande échelle COSMOS 1050K, qui comprend 18 modalités d'imagerie, 84 cibles de segmentation biomédicale, 1050K images 2D et 6033K masques de segmentation. Sur la base de cet ensemble de données, les chercheurs ont mené une évaluation complète du SAM et ont exploré les moyens d’améliorer les capacités du SAM en matière de perception des cibles médicales.
Téléchargement direct de l'ensemble de données de segmentation d'images médicales COSMOS 1050K :
En outre, des équipes de l’École de Big Data de l’Université Fudan et de l’École d’ingénierie biomédicale de l’Université Jiao Tong de Shanghai ont également mené une série d’études sur le SAM dans le domaine de la segmentation des images médicales. L'article pertinent s'intitule « Modèle de segmentation pour la segmentation d'images médicales : applications actuelles et directions futures » et est inclus dans des sites Web et des revues universitaires bien connus tels qu'arXiv et Computer in Biology and Medicine.

Cet article se concentre sur l'application possible de SAM, qui a réalisé des progrès remarquables dans la segmentation d'images naturelles, dans le domaine de la segmentation d'images médicales, et explore le réglage fin du module SAM et le recyclage d'architectures similaires pour s'adapter à la segmentation d'images médicales.
Adresse du document :
https://www.sciencedirect.com/science/article/abs/pii/S0010482524003226
En résumé, comme indiqué dans les articles ci-dessus, en explorant le potentiel du SAM, les scientifiques ont rendu le traitement et l’analyse des images médicales plus simples et plus efficaces, ce qui sera un résultat attendu avec impatience par le monde universitaire, la communauté médicale et même les patients. Dans le même temps, la sortie de modèles généraux de segmentation d’images tels que SAM a également ouvert une porte magique vers divers domaines.Je crois que non seulement le domaine de l’imagerie médicale, mais aussi la conduite autonome, les nouveaux médias, la RA/RV… pourraient en bénéficier grandement à l’avenir.
Tirage au sort du livre

HyperAI et la maison d'édition de l'industrie électronique vous ont conjointement proposé des livres gratuits ! Nous avons préparé 5 livres de vulgarisation scientifique super utiles « AI pour la science : l'intelligence artificielle stimule l'innovation scientifique », venez participer au tirage au sort~
Comment participer
Suivez le compte officiel HyperAI WeChat, répondez « Livre gratuit AI4S » en arrière-plan et cliquez sur la page du tirage au sort pour participer au tirage au sort. Nous avons préparé pour vous 5 livres qui vous seront livrés par livraison express. Venez participer !
Présentation du livre
De la prédiction de la structure des protéines à la déduction de la pathogénicité des mutations génétiques, le nouveau paradigme porté par l’IA nous a permis de voir de nouvelles opportunités dans divers domaines scientifiques, y compris les sciences de la vie.
Le livre « AI for Science: Artificial Intelligence Drives Scientific Innovation » se concentre sur l'intégration croisée de l'intelligence artificielle avec cinq domaines majeurs : la science des matériaux, les sciences de la vie, la science électronique, la science de l'énergie et la science de l'environnement. Il utilise un langage facile à comprendre pour présenter de manière exhaustive les concepts de base, les principes techniques et les scénarios d'application, permettant aux lecteurs de maîtriser rapidement les connaissances de base de l'IA pour la science. En outre, pour chaque domaine transversal, le livre fournit une introduction détaillée à travers des cas, trie la carte du secteur et donne des informations politiques pertinentes.








