Command Palette
Search for a command to run...
Aperçu En Direct : Li Yuzhe, Chercheur Postdoctoral De l'Université Tsinghua, Explique En Détail Les Articles De La sous-revue Cell/Nature, Explorant Les Applications De l'IA En Génomique

Le deuxième épisode de la série en direct « Meet AI4S » débutera à 19h00 le 21 août ! HyperAI a l'honneur d'inviter Li Yuzhe, chercheur postdoctoral au laboratoire de Zhang Qiangfeng à l'Université Tsinghua, à partager avec nous des méthodes d'IA dans la recherche en transcriptomique spatiale et en omique unicellulaire.
Les résultats de recherche du Dr Li Yuzhe en tant que premier auteur, « Découverte de modules tissulaires dans les données transcriptomiques spatiales à résolution unicellulaire via l'intégration cellulaire consciente de l'interaction cellule-cellule », ont été publiés dans Cell Systems.Il présentera également plus en détail la méthode innovante proposée dans cette étude : l'algorithme d'intelligence artificielle SPACE basé sur le framework d'apprentissage profond de l'autoencodeur de graphes.
aussi,Le Dr Li Yuzhe a participé à la recherche d'algorithmes d'intelligence artificielle basés sur le cadre d'apprentissage profond de l'autoencodeur variationnel SCALEX,Il a également été publié dans Nature Communications sous le titre « Intégration de données monocellulaires en ligne via la projection d'ensembles de données hétérogènes dans un espace commun d'intégration de cellules ». Il partagera également avec tout le monde les idées de recherche à l'origine de cette réalisation lors de la diffusion en direct.
Détails de l'événement

Partager le sujet
Explorer les applications de l'IA en génomique : prendre l'algorithme de représentation spatiale des données du transcriptome SPACE comme exemple
Introduction
Avec le développement rapide de la recherche en sciences de la vie et de la technologie de l'intelligence artificielle (IA), l'IA joue un rôle de plus en plus important dans la recherche biomédicale, donnant naissance à un nouveau paradigme de recherche interdisciplinaire « l'IA pour la science ».
Cette diffusion en direct partagera la recherche en génomique, principalement les méthodes d'intelligence artificielle en transcriptomique spatiale et la recherche en omique unicellulaire.
Avantages pour le public
1. Acquérir une compréhension préliminaire du processus de développement et des progrès de pointe des méthodes d’intelligence artificielle dans la recherche en génomique.
2. Comprendre SCALEX, un algorithme d'intégration de données omiques à cellule unique basé sur des modèles génératifs.
3. Comprendre l'algorithme de représentation des données transcriptomiques spatiales SPACE basé sur un réseau neuronal graphique et le concept de communauté cellulaire.
Revue de presse
HyperAI a déjà interprété et partagé un article de recherche intitulé « Découverte de modules tissulaires dans les données de transcriptomique spatiale à résolution unicellulaire via l'intégration cellulaire consciente de l'interaction cellule-cellule » avec le Dr Li Yuzhe comme premier auteur.
Points saillants de la recherche
* Développement de SPACE, un outil d'analyse d'intelligence artificielle pour les données de transcriptome spatial, qui peut identifier les types de cellules spatiales et découvrir des modules tissulaires à partir de données de transcriptome spatial avec une résolution unicellulaire.
* SPACE surpasse considérablement les autres outils d’identification des types de cellules et de découverte de modules tissulaires, en particulier dans les tissus complexes contenant plusieurs types de cellules.
* SPACE définit et découvre des communautés cellulaires, c'est-à-dire des modules tissulaires composés de types cellulaires spatialement homogènes avec des limites reconnaissables.
* Les communautés cellulaires sont définies par des réseaux d'interaction similaires entre les cellules qu'elles composent, qui peuvent être utilisés pour affiner les inférences basées sur le ligand-récepteur concernant la communication cellulaire.
* SPACE peut être utilisé pour des études de transcriptome spatial à grande échelle afin de comprendre comment les interactions entre cellules spatialement voisines affectent les fonctions biologiques des types de cellules et des modules tissulaires.
Acquisition de jeux de données
Afin de vérifier les capacités de SPACE, plusieurs ensembles de données ont été utilisés dans l’étude. Adresse de téléchargement :
Architecture du modèle : un modèle intégré aux cellules prenant en compte les interactions cellule-cellule
SPACE utilise un cadre d'autoencodeur graphique pour apprendre les intégrations cellulaires de faible dimension qui décrivent les informations d'expression génétique de chaque cellule dans les données du transcriptome spatial ainsi que ses informations d'interaction avec les cellules voisines spatiales (d'où le nom d'intégration cellulaire intégration cellulaire consciente de l'interaction cellule-cellule). Sur la base de cette intégration cellulaire, SPACE utilise ensuite des algorithmes de clustering pour identifier les sous-types de cellules spatiales et découvrir les modules tissulaires.
Du point de vue de l'architecture, le modèle SPACE se compose de trois parties : un encodeur (réseau d'attention graphique à trois couches), un décodeur de graphe voisin et un décodeur d'expression génétique. La figure suivante montre le cadre général du modèle :
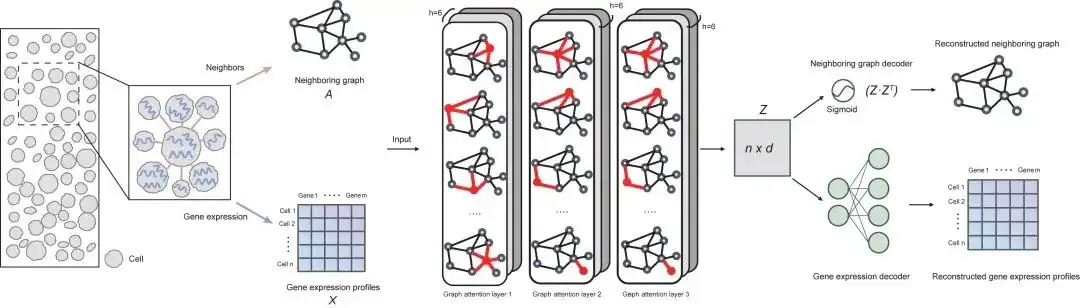
Évaluation des performances
* SPACE peut identifier des types de cellules biologiquement distincts sur la base d'informations spatiales dans des ensembles de données ST.
* SPACE surpasse les outils actuellement disponibles pour distinguer les types de cellules spatialement informatives des données ST.
* SPACE surpasse les outils de pointe en matière de découverte de modules tissulaires.
Laboratoire Zhang Qiangfeng de l'Université Tsinghua

Le laboratoire de Zhang Qiangfeng est affilié à l'École des sciences de la vie de l'Université Tsinghua. Il s'agit également d'une partie importante du Centre conjoint des sciences de la vie de l'Université Tsinghua-Pékin et du Centre d'innovation avancée de Pékin pour la biologie structurelle.
Les recherches du laboratoire se concentrent sur des domaines interdisciplinaires tels que la biologie structurale, la génomique, l'apprentissage automatique et l'analyse de big data. L'orientation principale de la recherche est de combiner la biologie structurale et la biologie des systèmes, de développer et d'utiliser une combinaison de méthodes informatiques et expérimentales pour interpréter la relation structure-fonction des macromolécules biologiques (telles que les protéines, l'ARN, l'ADN), de reconstruire leurs réseaux d'interaction et de découvrir la pathogenèse et les traitements possibles de maladies complexes (y compris le cancer et les maladies infectieuses) liées aux changements dans la structure des protéines et de l'ARN et aux interactions macromoléculaires anormales.
Le laboratoire dispose d'une modélisation unique de la structure des protéines et de l'ARN, d'une mesure de la structure de l'ARN basée sur le séquençage de nouvelle génération, d'une technologie de détection d'interaction ARN-protéine à haut débit et de puissantes plateformes informatiques et expérimentales pour faire progresser la recherche de pointe des chercheurs.
Découvrez la série AI4S Live
HyperAI (hyper.ai) est le plus grand moteur de recherche de Chine dans le domaine de la science des données. Il se concentre sur les derniers résultats de recherche scientifique de l'IA pour la science et suit en temps réel les articles universitaires dans les meilleures revues telles que Nature et Science. Jusqu’à présent, il a achevé l’interprétation de plus de 100 articles sur l’IA pour la science.
De plus, nous exploitons également le seul projet open source d'IA pour la science en Chine, awesome-ai4s.
* Adresse du projet :
https://github.com/hyperai/awesome-ai4s
Afin de promouvoir davantage la popularisation de l'IA4S, de réduire davantage les barrières de diffusion des résultats de la recherche scientifique des institutions universitaires et de les partager avec un plus large éventail de chercheurs de l'industrie, de passionnés de technologie et d'unités industrielles, HyperAI a planifié la colonne vidéo « Meet AI4S », invitant les chercheurs ou les unités connexes qui sont profondément engagés dans le domaine de l'IA pour la science à partager leurs résultats de recherche et leurs méthodes sous forme de vidéos, et à discuter conjointement des opportunités et des défis auxquels est confrontée l'IA pour la science dans le processus de progrès, de promotion et de mise en œuvre de la recherche scientifique, afin de promouvoir la vulgarisation et la diffusion de l'IA pour la science.








