Command Palette
Search for a command to run...
Le Niveau Est Presque Aussi Bon Que Celui Des Pathologistes Seniors ! L'équipe De Tsinghua Propose Un Modèle ROAM Basé Sur l'IA Pour Obtenir Un Diagnostic Précis Du Gliome
Le gliome est une tumeur qui provient des cellules gliales du cerveau, représentant 40 à 60 % de toutes les tumeurs primaires du système nerveux central et est connu comme la tumeur intracrânienne primaire la plus courante chez les adultes. La classification histopathologique des gliomes est très complexe et est généralement divisée en trois sous-types : les astrocytomes, les oligodendrogliomes et les épendymomes, chacun pouvant être divisé en plusieurs grades. donc,Une classification et un classement précis sont essentiels pour le pronostic et le traitement des gliomes.
Le diagnostic du gliome est généralement réalisé par des pathologistes expérimentés grâce à l'observation de coupes de tissus. Cependant, cette méthode présente des défis tels que la pénurie de pathologistes, le diagnostic subjectif et le processus de diagnostic long.Il est difficile de répondre aux besoins diagnostiques actuels des gliomes.
Les progrès récents en pathologie numérique et en apprentissage automatique ont permis la numérisation de sections histologiques, qui sont converties en images de diapositives entières (WSI) de plusieurs gigapixels contenant des données contextuelles riches avec un grand potentiel pour le diagnostic, le pronostic et la caractérisation moléculaire. Cependant, ces méthodes analysent uniquement les régions d’intérêt (ROI) dans les images de pathologie sélectionnées par les pathologistes.L'analyse automatisée de la diapositive entière n'est pas possible.
Dans ce contexte,Le chercheur associé Lu Hairong, le professeur Jiang Rui, le professeur Zhang Xuegong du laboratoire de modèles de base de la vie du département d'automatisation de l'université Tsinghua et le professeur Hu Zhongliang de l'hôpital Xiangya de l'université du centre-sud,Un modèle de base d'IA de diagnostic pathologique précis ROAM basé sur de grandes régions d'intérêt et un transformateur pyramidal est proposé pour le diagnostic au niveau clinique et la découverte de marqueurs moléculaires du gliome, et peut être étendu au diagnostic pathologique d'autres types de tumeurs.
Les résultats de recherche connexes ont été publiés dans Nature Machine Intelligence sous le titre « Une méthode de pathologie computationnelle faiblement supervisée basée sur un transformateur pour le diagnostic de qualité clinique et la découverte de marqueurs moléculaires des gliomes ».
ROAM peut extraire efficacement des informations multi-échelles riches à partir d'images pathologiques et réaliser un diagnostic précis pour diverses tâches de classification telles que la détection de tumeurs de gliome, la classification/le classement des sous-types et la prédiction des caractéristiques moléculaires.Sur la base de données internes, ROAM a démontré d’excellentes performances diagnostiques, en capturant automatiquement les principales caractéristiques morphologiques conformes à l’expérience des pathologistes et en fournissant un diagnostic clinique précis, fiable et adaptable pour les gliomes.
De plus, ROAM peut être étendu à des données externes indépendantes et possède une excellente capacité de généralisation. En visualisant et en expliquant les diagnostics, ROAM peut aider les pathologistes à vérifier la fiabilité de la base diagnostique du modèle, à extraire des informations précieuses et à promouvoir le diagnostic auxiliaire pour améliorer les soins médicaux. Plus important encore, ROAM facilite la découverte de biomarqueurs moléculaires et morphologiques, offrant de nouvelles perspectives sur le diagnostic et le traitement des gliomes.
Points saillants de la recherche :
* ROAM permet une extraction efficace des représentations visuelles des images de pathologie tissulaire en coupes entières grâce à des patchs d'images de grande taille et à des modules d'apprentissage de fonctionnalités multi-échelles
* ROAM peut être étendu à des données externes indépendantes et possède une excellente capacité de généralisation
* ROAM facilite la découverte de biomarqueurs moléculaires et morphologiques, offrant de nouvelles perspectives sur le diagnostic et le traitement des gliomes

Adresse du document :
https://www.nature.com/articles/s42256-024-00868-w
Téléchargement du jeu de données :
Le projet open source « awesome-ai4s » rassemble plus de 100 interprétations d'articles AI4S et fournit des ensembles de données et des outils massifs :
https://github.com/hyperai/awesome-ai4s
Ensemble de données : Deux principaux ensembles de données de lames de pathologie tissulaire numérique
Dans cette étude, deux ensembles de données de lames de pathologie tissulaire numérique (WSI) à grande échelle ont été collectés pour la recherche sur le gliome.
1. Ensemble de données WSI sur le gliome de Xiangya
Cet ensemble de données provient de sections de gliome de l'hôpital Xiangya de l'Université du Centre-Sud.Comme le montre la figure ci-dessous, il contient 1 109 WSI traités à un grossissement × 40 dans le même nombre de cas différents, couvrant des tâches de diagnostic telles que la détection de gliomes, la classification des sous-types, le classement et la prédiction des caractéristiques moléculaires, et le même nombre de tranches a été collecté pour chaque cas.

L'ensemble de données contient uniquement des annotations au niveau des tranches qui indiquent le sous-type et le grade de 530 astrogliomes, 224 oligodendrogliomes et 213 épendymomes. De plus, des tests moléculaires ont été réalisés dans 634 cas présentant une mutation IDH et 641 cas présentant une méthylation du promoteur MGMT.
L'ensemble de données est divisé aléatoirement en deux parties :L'un est un ensemble de données de formation interne contenant 736 WSI, qui est utilisé pour la formation du modèle ; l'autre est un ensemble de données de test interne contenant 373 WSI, qui est utilisé pour l'évaluation du modèle et les expériences liées aux médecins. La proportion de classes dans les deux ensembles de données est la même que dans l’ensemble de données.
2. Ensemble de données WSI sur le gliome TCGA
Un autre ensemble de données WSI d'histopathologie des gliomes provient du projet Brain Low-Grade Gliomas and Glioblastoma.Il y a un total de 860 sections de glioblastome (sur 389 cas) et 844 sections de gliome de bas grade (sur 491 cas). Les critères de diagnostic sont différents de ceux utilisés dans l'ensemble de données Xiangya, de sorte que seules les annotations au niveau des tranches dans les données sont conservées. Deux pathologistes qui ont participé à l’annotation de l’ensemble de données Xiangya ont été invités à examiner et à réviser les résultats diagnostiques de ces sections conformément à la version 2016 des directives diagnostiques.
Enfin, l'ensemble de données examiné comprend 618 WSI avec des grossissements de ×40 et ×20, couvrant 4 tâches cohérentes avec l'ensemble de données Xiangya. Cet ensemble de données sert d'ensemble de données de test externe pour la classification des sous-types de gliome, le grade et la prédiction des caractéristiques moléculaires.
Architecture du modèle : basée sur un grand intérêt régional et un transformateur pyramidal
ROAM est une méthode de pathologie computationnelle faiblement superviséeCette méthode utilise l'apprentissage multi-instances comme cadre de base, des blocs d'images de tissus de grande taille comme unité de recherche de base et utilise Pyramid Transformer pour apprendre systématiquement les caractéristiques de corrélation intra-échelle et inter-échelle de chaque bloc de tissu, permettant ainsi une extraction efficace des représentations visuelles d'images de tranches de tissus entières.
d'abord,ROAM effectue une segmentation tissulaire sur chaque image de tranche entière et en extrait des patchs d'images tissulaires de grande taille (2048 × 2048) comme unité de base pour l'analyse ultérieure, à savoir ROI, comme illustré dans la figure suivante Patching WSI :
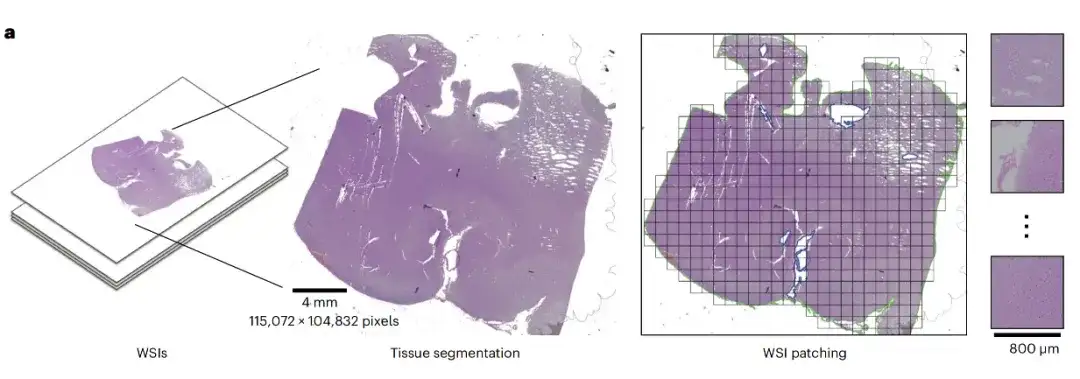
Deuxièmement,Chaque ROI a été sous-échantillonnée deux fois en continu pour générer trois images avec des grossissements différents. Chaque image est ensuite segmentée en petits morceaux d'image, qui sont codés via un réseau neuronal convolutif pré-entraîné pour extraire leurs représentations visuelles, qui sont utilisées comme entrée du modèle MIL, comme indiqué dans l'extraction de caractéristiques d'instance sur le côté gauche de la figure b ci-dessous ; un module d'auto-attention multi-échelle (SA) et un réseau d'attention sont utilisés pour générer des représentations au niveau de l'instance, et ces informations sont agrégées dans des représentations au niveau de la tranche, comme indiqué dans l'extraction de caractéristiques multi-échelles sur le côté droit de la figure b ci-dessous.
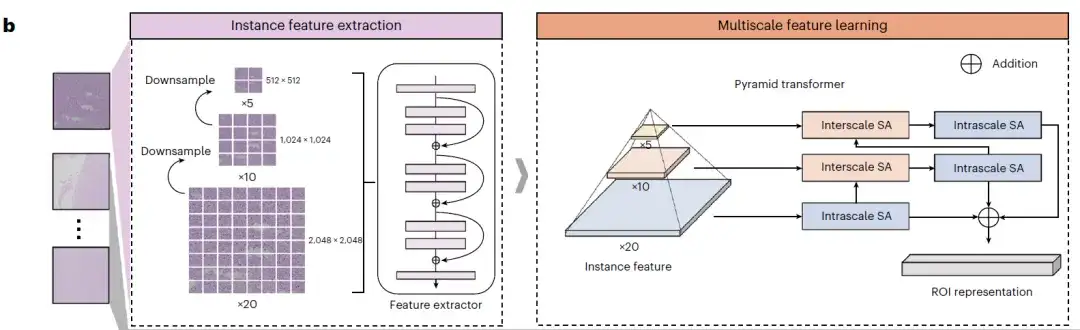
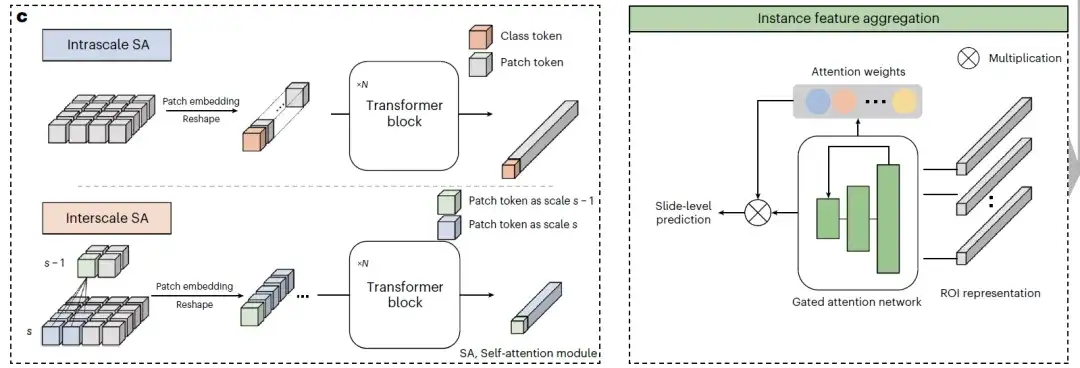
enfin,Comme le montre la figure c (agrégation de caractéristiques d'instance), deux types différents de modules SA utilisent l'architecture du transformateur pyramidal pour fusionner progressivement des caractéristiques multi-échelles d'un grossissement élevé à un faible grossissement afin d'obtenir une représentation visuelle multi-échelle du bloc de tissu. Le module SA intra-échelle et le module SA inter-échelle apprennent respectivement les caractéristiques intra-échelle et inter-échelle du ROI. Les deux modules contiennent plusieurs couches SA multi-têtes et couches de rétroaction.
Résultats de recherche : ROAM permet un diagnostic précis du gliome
ROAM permet un diagnostic précis du gliome
Les chercheurs ont évalué les performances de classification de ROAM sur des ensembles de données internes et des ensembles de données externes TCGA.
Comme le montre la figure a ci-dessous, ROAM surpasse cinq méthodes, dont CLAM, TransMIL, GTP, TEA-graph et H(2)MIL, et surpasse d'autres méthodes de base dans les tâches liées au diagnostic du gliome sur des ensembles de données internes. Lors de la détection de 3 types de gliomes, à savoir normaux, glioses et tumoraux, la surface de la courbe ROC macro-moyenne (AUC) de ROAM était de 0,990 ± 0,002.
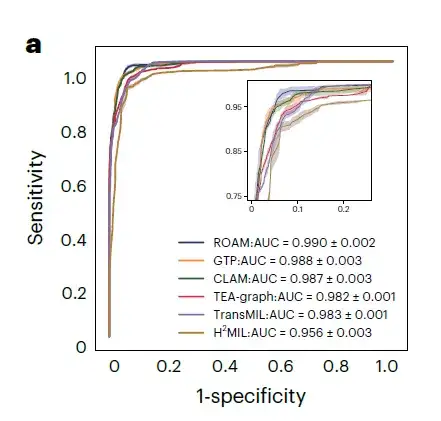
Pour la classification de trois sous-types de gliome, à savoir l'astrocytome, l'oligodendrogliome et l'épendymome, l'ASC de ROAM était de 0,950 ± 0,003, comme indiqué dans la figure b ci-dessous.
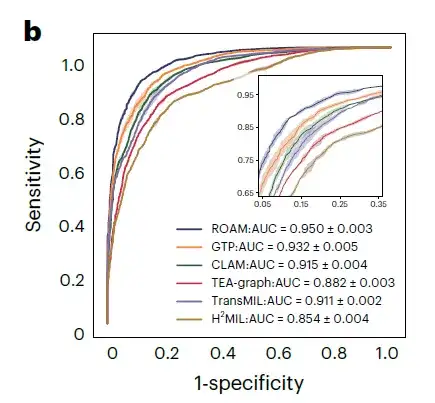
Dans ces tâches de diagnostic du gliome, ROAM a obtenu l’ASC la plus élevée parmi toutes les méthodes de base.L’efficacité et la haute performance du modèle ROAM dans le diagnostic du gliome ont été démontrées.
en même temps,ROAM possède également de bonnes propriétés de généralisation.Formé uniquement avec des ensembles de données internes et testé sur des ensembles de données TCGA externes, ROAM surpasse toujours les autres méthodes de base dans l'ensemble. aussi,Les résultats de visualisation des prédictions ROAM montrent également que la base diagnostique résumée par cette méthode est très cohérente avec les critères de diagnostic clinique.
ROAM fait progresser considérablement le diagnostic clinique auxiliaire du gliome
Les chercheurs ont mené une évaluation clinique complète du ROAM.La performance du ROAM dans le diagnostic auxiliaire du gliome a été étudiée : 3 groupes de 5 pathologistes ont été invités à participer à l'étude, un groupe était composé de pathologistes juniors avec moins de 5 ans d'expérience clinique, deux groupes étaient des pathologistes intermédiaires avec 5 à 15 ans d'expérience clinique, et les deux autres groupes étaient des pathologistes seniors avec plus de 15 ans d'expérience clinique.
Comme le montre la figure ci-dessous, le système proposé a obtenu de bons résultats sur les cinq tâches du diagnostic de la cascade de gliomes, surpassant quatre pathologistes sur cinq et comparable au pathologiste senior le plus performant (Senior 1). Plus précisément, dans la détection du gliome,Le nouveau système a largement surpassé tous les pathologistes.Y compris les pathologistes les plus performants, dépassant 21.30%, comme le montre la figure f ci-dessous.
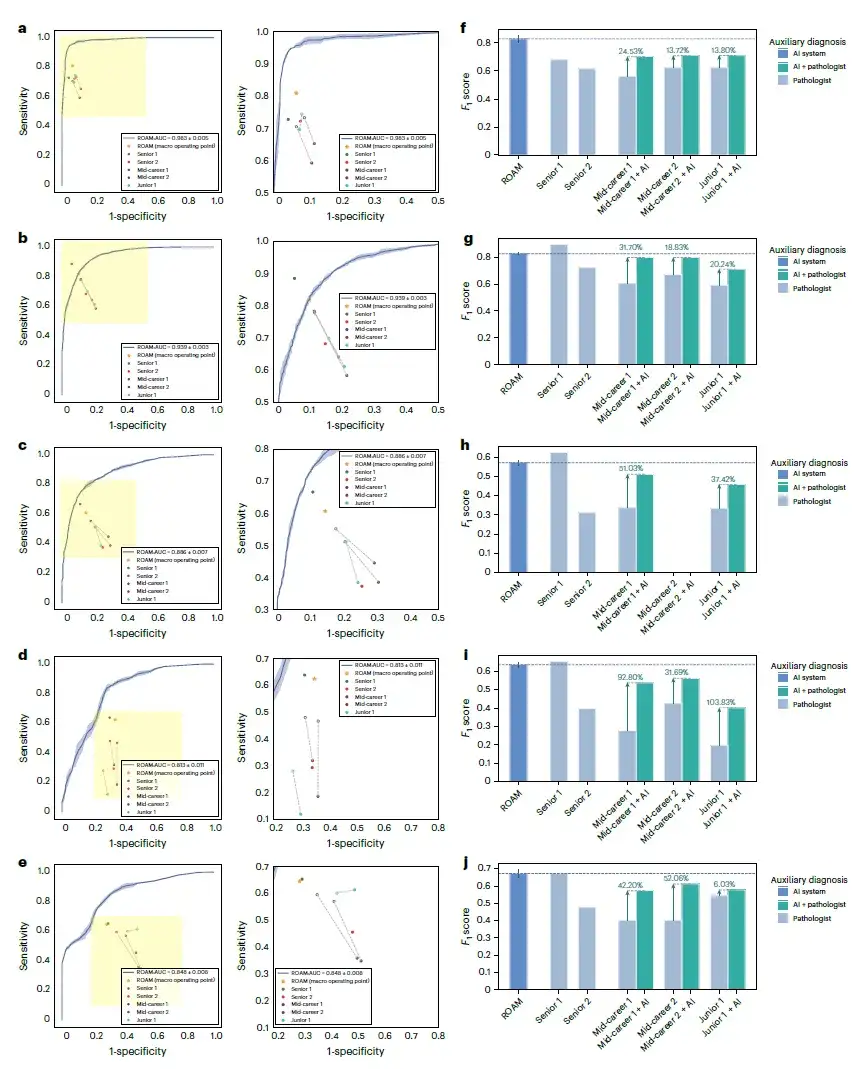
Les chercheurs ont ensuite demandé à trois pathologistes juniors et intermédiaires d’effectuer des diagnostics à l’aide de ROAM pour déterminer si leurs performances diagnostiques s’amélioraient. Les résultats montrent qu'avec l'aide de ROAM, la précision diagnostique des trois pathologistes s'est améliorée en moyenne de 7,27% (primaire 1), 12,87% (intermédiaire 1) et 9,96% (intermédiaire 2) dans toutes les tâches.Cela reflète la grande valeur d’application clinique de ROAM.
ROAM favorise la découverte de marqueurs morphologiques moléculaires du gliome
Avec l'aide de ROAM, les chercheurs ont exploré les manifestations morphologiques des principales caractéristiques moléculaires liées au diagnostic du gliome et ont remarqué que ROAM fonctionnait bien dans la tâche de prédiction des mutations de l'isocitrate déshydrogénase (IDH). Ils ont effectué une analyse visuelle complète des résultats de prédiction de ROAM dans cette tâche, et ont analysé et résumé les caractéristiques morphologiques des tissus des zones clés à forte attention sur lesquelles ROAM s'est concentré. Ils ont découvert que l’éosinophilie, le cytoplasme uniforme et la coloration sombre du noyau étaient courants dans les images pathologiques des mutations IDH.
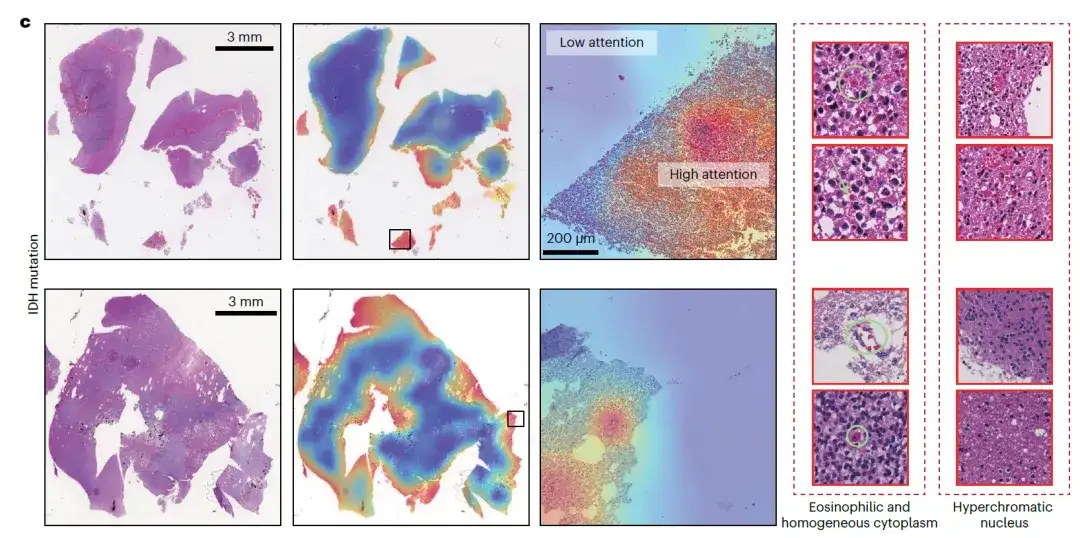
Cette découverte importante aide les médecins à faire des prédictions préliminaires du statut IDH sans l’aide de tests moléculaires et favorisera considérablement l’optimisation et l’amélioration des normes de diagnostic clinique pour les gliomes.
Le laboratoire de modèles de base de la vie du département d'automatisation de l'université Tsinghua continue de promouvoir l'IA pour renforcer la recherche en sciences de la vie
Dans ce document,Le chercheur associé Lü Hairong, le professeur Zhang Xuegong et le professeur Hu Zhongliang de l'hôpital Xiangya de l'Université du Centre-Sud sont les auteurs correspondants.Le professeur Jiang Rui et l'étudiant en master Yin Xiaoxu de l'Université Tsinghua, Yang Pengshuai de l'Institut de recherche mobile de Chine et Cheng Lingchao de l'hôpital Xiangya sont les co-premiers auteurs de l'article. Hu Jun, Yang Jiao, Wang Ying, Fu Xiaodan, Shang Li, Li Liling, Lin Wei et Zhou Huan ont participé à la collecte et à l'annotation des données pour cette étude. Chen Fuxun et l'Institut de recherche en technologie des données de Fuzhou ont fourni un soutien en R&D pour la plate-forme logicielle en ligne.
En tant que l’un des principaux contributeurs à cette étude,Le laboratoire de modèles de base de la vie du département d'automatisation de l'université Tsinghua s'engage constamment à explorer comment renforcer la recherche en sciences de la vie grâce à des technologies avancées d'intelligence artificielle.
En juin de cette année, le professeur Zhang Xuegong, directeur du laboratoire des modèles de base de la vie du département d'automatisation de l'université Tsinghua, le professeur Ma Jianzhu du département d'électronique/AIR et le Dr Song Le de biotechnologie,Un modèle de grande cellule appelé scFoundation a été établi.Le modèle a été formé sur des données d’expression génétique provenant de 50 millions de cellules, comporte 100 millions de paramètres et peut traiter environ 20 000 gènes simultanément. En tant que modèle de base, il a montré d'excellentes améliorations de performances dans une variété de tâches biomédicales en aval telles que les « essais virtuels de médicaments ».Fournit un nouveau paradigme pour l’intelligence artificielle dans la recherche sur les cellules uniques.
Les résultats de la recherche ont été publiés dans Nature Methods sous le titre « Modèle de fondation à grande échelle sur la transcriptomique à cellule unique ».Cliquez pour consulter le rapport complet : « Un modèle cellulaire avec 100 millions de paramètres est disponible ! Publié dans la sous-revue Nature, l'équipe de l'Université Tsinghua publie scFoundation : Modélisation simultanée de 20 000 gènes. »
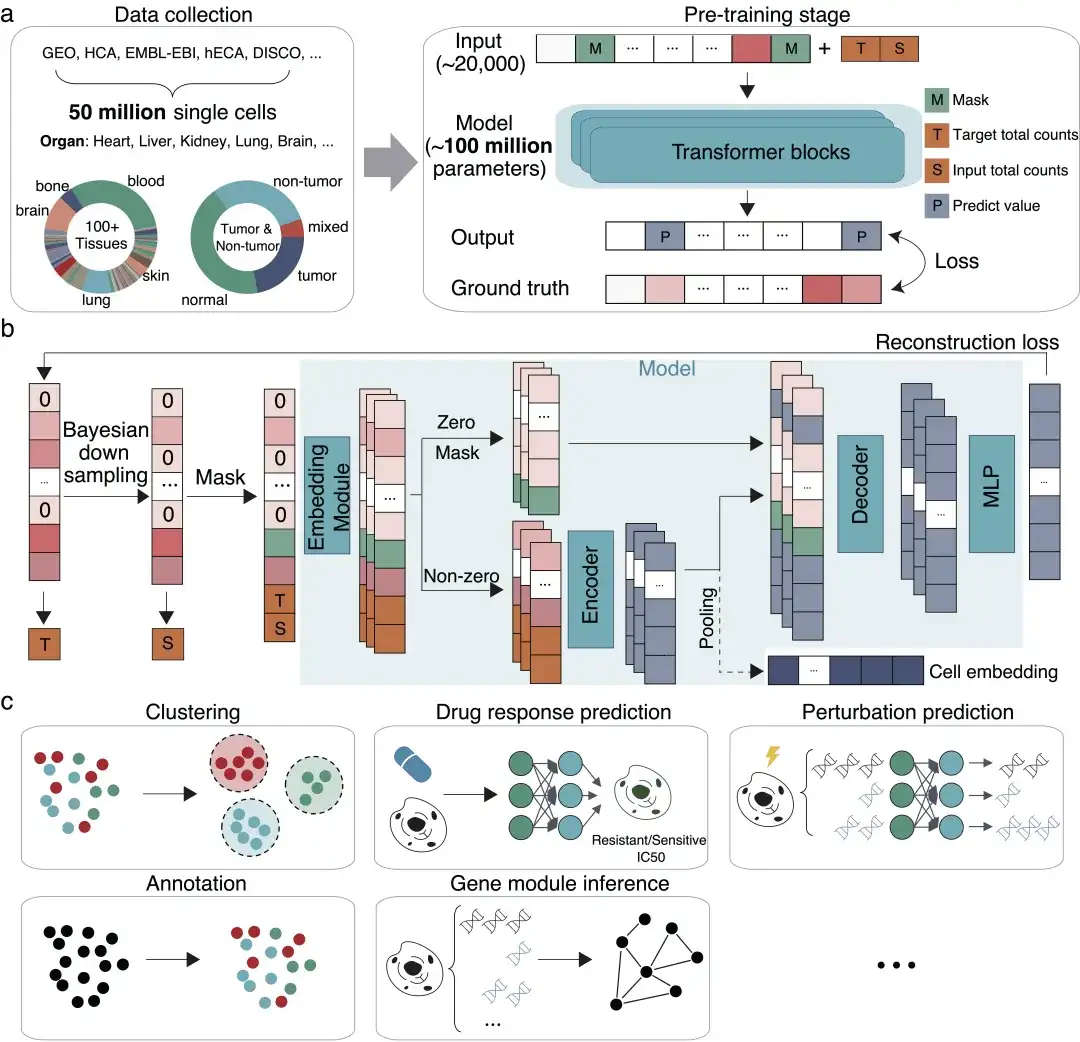
Le modèle scFoundation fournit des méthodes et des outils innovants pour la recherche fondamentale en sciences de la vie, la prédiction des réponses aux perturbations cellulaires, la découverte de cibles médicamenteuses et d'autres domaines. Il fournit également de nouvelles idées et méthodes pour la recherche sur les modèles cellulaires à grande échelle en termes d'architecture de modèle, de cadre de formation et de système d'application de démonstration en aval. Il a réussi à repousser les limites des modèles de base dans le domaine des cellules uniques et à jeter les bases de recherches futures telles que les expériences de médicaments virtuels dans l'espace numérique.
En regardant vers l’avenir, le laboratoire de modèles de base de la vie du département d’automatisation de l’université Tsinghua continuera de mener des recherches dans le domaine interdisciplinaire de l’intelligence artificielle et des sciences de la vie. Avec le développement et l'amélioration continus de la technologie de l'IA, l'application de l'intelligence artificielle dans le domaine des sciences de la vie favorisera grandement l'avancement de la technologie médicale, améliorera la précision du diagnostic et du traitement, réduira les coûts médicaux et, en fin de compte, améliorera la santé humaine et la qualité de vie.
Références :
1.https://www.nature.com/articles/s42256-024-00868-w
2.https://mp.weixin.qq.com/s/oB3kTgcgObawPKU-75KsHQ
3.https://mp.weixin.qq.com/s/nflI4PVTJB3xVPXuA5zbZQ
Appel à l'action
HyperAI (hyper.ai) est le plus grand moteur de recherche de Chine dans le domaine de la science des données. Il se concentre depuis longtemps sur les derniers résultats de recherche en matière d’IA pour la science et a interprété plus de 100 articles universitaires dans des revues de premier plan.
Les groupes et équipes de recherche qui mènent des recherches et des explorations autour de l'IA pour la science sont invités à nous contacter pour partager leurs derniers résultats de recherche, contribuer à des articles d'interprétation approfondis et participer à la chronique de diffusion en direct Meet AI4S. D’autres moyens de promouvoir l’IA4S nous attendent pour être explorés ensemble !
Ajouter WeChat : PH (ID WeChat : G18539589505)








