Command Palette
Search for a command to run...
L'Université De Floride Utilise Des Réseaux Neuronaux Pour Déchiffrer La Sélectivité Du Couplage Des Protéines GPCR-G
Sommaire en un coup d'œilLes récepteurs couplés aux protéines G (GPCR) sont des protéines transmembranaires qui transmettent des stimuli à l'extérieur de la membrane cellulaire et sont largement impliqués dans les activités physiologiques humaines. Récemment, des chercheurs de l’Université de Floride ont déterminé la sélectivité de liaison des GPCR et des protéines G, ont développé un algorithme pour prédire la sélectivité des deux et ont étudié la base structurelle de cette sélectivité.
Mots-clésDéveloppement de médicaments à base de réseaux neuronaux GPCR
Auteur | Xuecai
Rédacteur | Sanyang
Les récepteurs couplés aux protéines G (GPCR) sont des protéines transmembranaires qui transmettent des stimuli de l'extérieur de la membrane cellulaire vers l'intérieur de la membrane cellulaire. En activant les protéines G dans la membrane cellulaire et leurs voies de signalisation en aval, les GPCR peuvent être largement impliqués dans des activités physiologiques importantes telles que le développement, l'immunité, la régulation hormonale et l'activité neuronale.
Les protéines G sont composées de sous-unités Gα, Gβ et Gγ, et leur diversité détermine la diversité des réponses au signal des GPCR. Le génome humain code 16 sous-unités Gα, qui sont divisées en quatre sous-familles :E/S , Gαq , Gαs et Gα12/13 . Les protéines G déterminent les voies de signalisation en aval, qui à leur tour déterminent les réponses cellulaires. Par conséquent, la liaison sélective des GPCR et des protéines G est la clé pour comprendre le système de signalisation des organismes.
La communauté universitaire pensait autrefois que les récepteurs couplés aux protéines G (GPCR) ne pouvaient se coupler qu’à une seule protéine G et a donc divisé les récepteurs couplés aux protéines G (GPCR) en quatre types en fonction de leurs fonctions. Cependant, les chercheurs ont récemment découvert que la plupart des GPCR se couplent à plusieurs protéines G pour activer des réponses cellulaires complexes. Le modèle de classification un à un n'est plus suffisant pour décrire la relation de couplage entre les GPCR et les protéines G, mais le mécanisme de sélectivité des GPCR-protéines G n'est pas encore clair.
à cette fin,Des chercheurs de l'Université de Floride ont utilisé des mesures cinétiques et la technologie de transfert d'énergie par résonance de bioluminescence (BRET) pour mesurer les facteurs d'échange de nucléotides guanine des GPCR pour les protéines G afin d'analyser la liaison sélective des deux. Sur cette base, les chercheurs ont classé les GPCR en fonction de leur préférence pour les protéines G et ont établi un modèle à gros grains de 124 GPCR provenant de différents mammifères. Par la suite, un algorithme permettant de prédire la sélectivité des protéines GPCR-G a été développé et la base structurelle de cette sélectivité a été étudiée.Des résultats connexes ont été publiés dansRapports cellulaires".

Ce résultat a été publié dans « Cell Reports »
Lien vers l'article :
https://doi.org/10.1016/j.celrep.2023.113173
01 BRET : Quantification sélective des protéines GPCR-G
Pour quantifier la sélectivité des protéines GPCR-G, les chercheurs ont utilisé la technique BRET pour mesurer l’activité des protéines G dans les cellules vivantes.

Technologie BRET pour la détection en temps réel de l'activité des protéines G
Les chercheurs ont ensuite vérifié cette découverte sur le récepteur de la cholécystokinine de type II (CCKBR). Les résultats de l'amplitude de réponse ont montré que CCKBR peut activer GαE/S , Gαq , Gα15 et Gα12/13 Les protéines G de la famille ont des niveaux d'activation similaires, mais ne peuvent pas activer Gαs Famille de protéines.
Les résultats basés sur le taux d'activation montrent clairement que CCKBR a un effet important sur Gαq L'effet d'activation de la famille est le meilleur, suivi de GαE/S , Gα15 et Gα12/13, ce qui montre que la technologie BRET basée sur le taux d'activation peut capturer des différences subtiles entre les activités de différentes protéines G.

Résultats BRET basés sur l'amplitude (C) et résultats BRET basés sur le taux d'activation (D)
Par conséquent,Les chercheurs ont mesuré la sélectivité de 124 GPCR et protéines G comme ensemble de données pour cette étude.

Mesures de sélectivité des GPCR de classe B et des protéines G
02 Construction de modèles : réseau neuronal de classification binaire
Les résultats ci-dessus indiquent que la technique BRET basée sur le taux d'activation peut distinguer la sélectivité entre des centaines de GPCR et de protéines G. Sur la base de ce résultat,Les chercheurs ont développé un algorithme basé sur l’apprentissage automatique pour prédire la sélectivité des protéines GPCR-G de classe A.
L'algorithme a deux tâches :
1. Concernant le couplage des GPCR, déterminer si un certain GPCR peut se coupler à la protéine G, c'est-à-dire l'amplitude > 0% ;
2. Concernant la sélectivité des GPCR, il est déterminé qu'un certain couple de protéines GPCR-G peut être rapidement activé, c'est-à-dire que le taux d'activation est >30%.
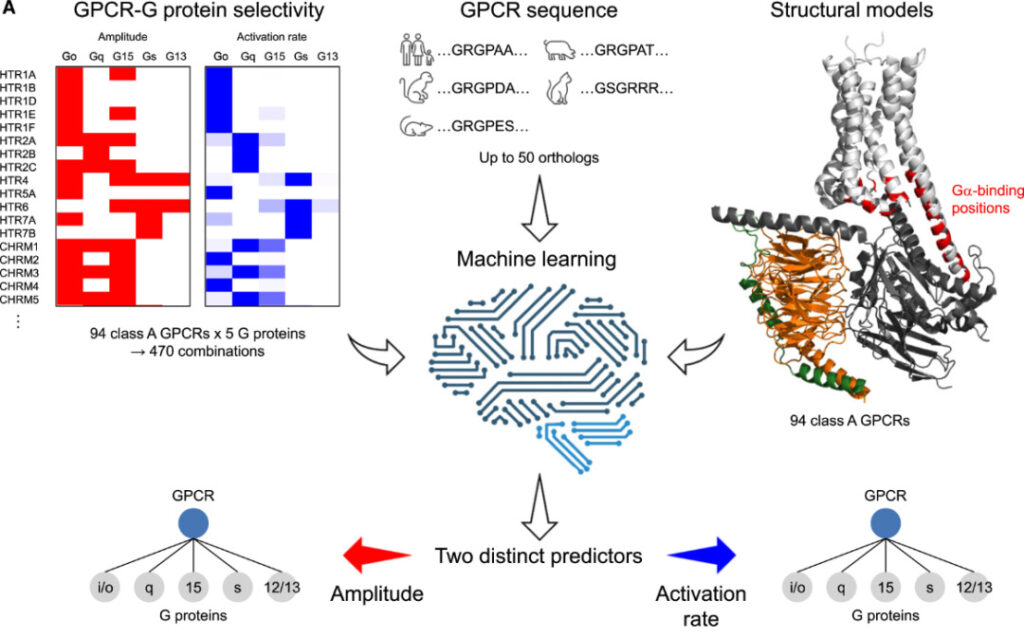
Diagramme conceptuel des algorithmes d'apprentissage automatique
Le couplage entre chaque GPCR et une protéine G d'une famille différente est un problème de classification, donc chaque tâche peut être conçue comme 5 classifications binaires. Sur cette base, les chercheurs ont conçu 10 classificateurs de réseaux neuronaux pour gérer ces tâches. Le réseau neuronal se compose de deux couches entièrement connectées (avec respectivement 128 et 16 neurones), d'une couche d'aplatissement, de trois couches entièrement connectées (avec respectivement 128, 32 et 4 neurones) et d'une couche de sortie (1 neurone). Les couches internes sont activées par des unités linéaires rectifiées (ReLU) et enfin une normalisation par lots est effectuée. La couche de sortie est activée par la fonction sigmoïde.
En raison de la quantité limitée de données, 50 séquences homologues ont été ajoutées à chaque GPCR pour l'expansion des données, en supposant que les séquences qui déterminent la sélectivité des protéines G sont relativement conservées au cours de l'évolution. Un protocole d'intégration de séquences est déployé dans le modèle pour décrire les propriétés des résidus de protéines dans un environnement spécifique à l'aide d'un modèle d'apprentissage profond non supervisé.
L'entrée du réseau neuronal est un tenseur de taille B*30*1024. La première dimension est la taille du lot (B=32), la deuxième dimension est le nombre de résidus (30) et la troisième dimension est pour chaquerésidu d'acide aminéLa taille des séquences intégrées pré-entraînées (1024).
Moyenne des prédictions du modèle pour l'amplitude et le taux d'activation AUROC Les deux sont à 0,85, ce qui indique que le modèle a de bonnes performances dans la prédiction des deux indicateurs.Parmi eux, pour Gs Les protéines familiales ont été les mieux prédites, avec des valeurs AUROC de 0,89 et 0,95, respectivement. Cependant, pour Gα15 et Gα12/13 famille de protéines, le modèle n'a montré aucune capacité d'apprentissage évidente.

Courbes ROC pour la prédiction de l'amplitude (C) et du taux d'activation (D)
03 Décoder le mécanisme de sélectivité des protéines GPCR-Gα
Les expériences BRET et l'apprentissage automatique fournissent une solution pour déchiffrer la base structurelle de la sélectivité des protéines GPCR-G. Sur cette base, les chercheurs ont étudié les complexes GPCR-protéines G disponibles et analysé 33 récepteurs de classe A pour trouver la structure qui détermine la sélectivité de la protéine GPCR-Gα de classe A.
Les chercheurs ont étudié le réseau de résidus des protéines GPCR-Gα et ont découvert que toutes les structures des GPCR faisant face au cytoplasme étaient impliquées dans la liaison aux protéines Gα à des degrés divers. De même, il existe 13 structures dans la protéine Gα qui sont liées à la liaison des récepteurs GPCR à la protéine Gα, parmi lesquelles l'hélice α C-terminale (H5) est la plus impliquée.

Interactions entre différents éléments structurels des GPCR et des protéines Gα
Pour le couplage universel GPCR-Gα, les GPCR utilisent ICL2, H8 et la plupart des résidus TM pour se lier aux protéines Gα. Parmi elles, la grande majorité des structures étaient principalement connectées à H5, tandis que ICL2 était plus largement connectée.
GPCR et GαE/S et Gαq Les modèles de couplage protéique des familles sont similaires, la seule différence étant que la liaison des GPCR aux premières dépend fortement de TM6, alors que la liaison aux secondes ne l'est pas. GPCR et Gαs Parmi les connexions, la part d'ICL2 et d'ICL3 a été considérablement réduite, et une plus grande dépendance est accordée à TM3 et TM5. Les résultats ci-dessus indiquent que pour différentes familles de protéines Gα, leur connexion avec les GPCR dépend de différentes structures.
De plus, les effets de structures spécifiques sur les protéines Gα de différentes familles ont été étudiés en combinant les séquences sélectives des protéines GPCR-G. Premièrement, jugez et GαE/S Les GPCR liés peuvent-ils se lier à Gα15 Combinez et comparez les différences entre les deux réseaux de résidus. Par rapport au premier, la ligature des GPCR au second a rompu l'association entre ICL3 et H4, affaibli l'interaction de ICL2-H5 et renforcé la connexion entre TM4-HN et ICL2-s2s3. Cela suggère que la connexion entre ICL2 et d’autres résidus peut être liée à GαE/S ou Gα15 Différences majeures dans les GPCR liés.

Les GPCR interagissent uniquement avec GαE/S Réseau de résidus connecté aux GPCR et Gα15/GαE/S Réseau de résidus connectés (K)
De même, les GPCR et Gαs et GαE/S Après avoir combiné les réseaux de résidus, les résultats ont montré que la connexion entre ICL1 et TM5 était la principale différence entre les deux.
Les résultats ci-dessus montrent queLe BRET et l'apprentissage automatique peuvent analyser le réseau de résidus protéiques de la liaison GPCR-protéine G et trouver la base structurelle de leur sélectivité, offrant ainsi une nouvelle méthode pour l'étude des GPCR.
04 AI-GPCR : régions inexplorées de 96.4%
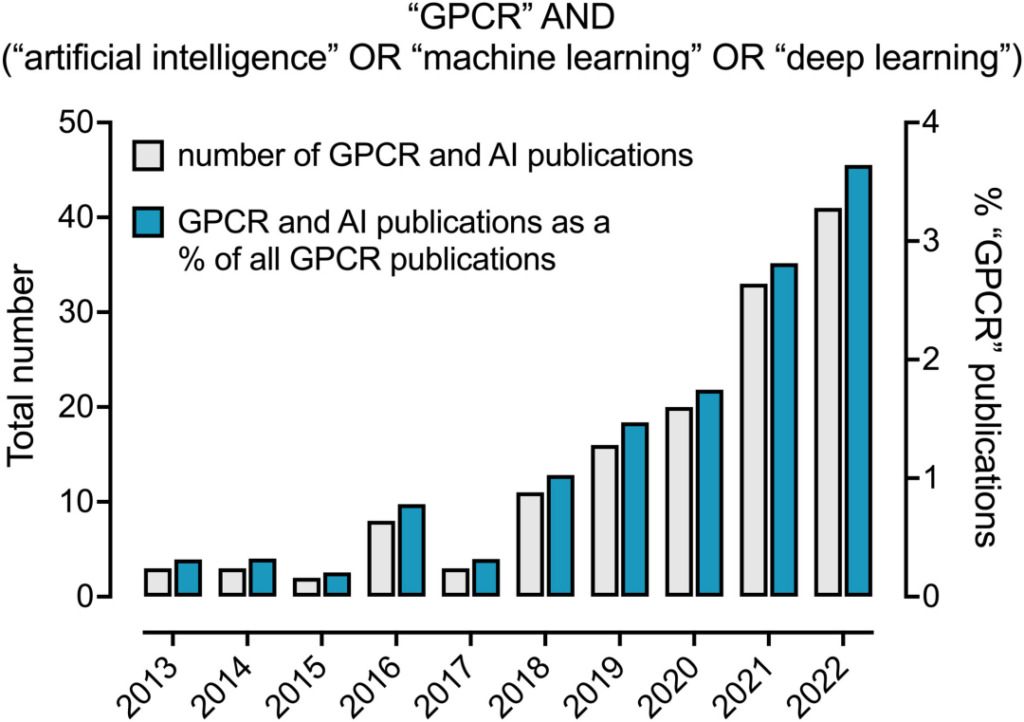
Au cours de la dernière décennie, la proportion d’applications d’IA et d’apprentissage automatique dans le domaine des GPCR a régulièrement augmenté. En 2022, 3,6% d'articles liés aux GPCR mentionnaient des méthodes liées à l'IA.
La proportion d'IA mentionnée dans les articles liés aux GPCR
Compte tenu de l’application croissante de l’IA dans la recherche sur les médicaments GPCR, des algorithmes correspondants sont également en cours de développement. Pour les problèmes de classification, les algorithmes les plus couramment utilisés sont ceux de l'apprentissage automatique traditionnel, tels que ceux de la bibliothèque scikit-learn, notamment les machines à vecteurs de support (SVM), les arbres de décision, les machines de boosting de gradient et les algorithmes des k plus proches voisins.
Pour les résultats numériques, tels que l'affinité de la liaison protéine-ligand, des algorithmes de régression sont souvent utilisés pour les résoudre, tels que la régression linéaire multivariée, les machines à vecteurs de support et les réseaux d'apprentissage profond.
Les résultats récents utilisent principalement des algorithmes d’apprentissage profond tels que le perceptron multicouche et le réseau neuronal convolutif (CNN) pour la prédiction. Avec le développement d’algorithmes génératifs d’apprentissage profond, la conception des ligands et des structures des protéines est devenue plus efficace et plus précise. Des algorithmes tels que le réseau antagoniste génératif, le réseau neuronal récurrent et l’apprentissage par renforcement peuvent utiliser la construction automatique et la mesure adaptative de l’espace vectoriel pour explorer des espaces de génération plus vastes.

Le rôle de l'IA à chaque étape du développement des médicaments GPCR
Par conséquent, ces algorithmes peuvent générer davantage de ligands avec les fonctions souhaitées ou prédire plus précisément les structures de protéines inconnues, par exemple. AlphaFold2 . Bien que des modèles tels qu'AlphaFold2 ne soient pas spécifiquement conçus pour prédire la structure des GPCR, ils peuvent néanmoins prédire la structure des GPCR de manière efficace et précise. De plus, l’apprentissage profond non supervisé ou auto-supervisé fait également son apparition dans la découverte de médicaments.
On peut voir que l’AI-GPCR peut être une nouvelle direction pour le développement futur de médicaments, mais il nous laisse également avec une zone inconnue de 96.4%. Grâce à une classification efficace et à des algorithmes de prédiction précis, les gens peuvent avoir une compréhension plus claire du mécanisme de couplage des GPCR, insufflant ainsi un nouvel élan au développement de la biomédecine.
