Command Palette
Search for a command to run...
Le Laboratoire National d'Argonne Lance FAST, Un Kit De Numérisation Automatisé Rapide Qui Permet Une « Lecture Rapide » De La Microscopie

« Je suis heureux de voir le drapeau national rouge hissé sur la place Tiananmen à Pékin »
Si vous lisez rapidement cette phrase, vous constaterez peut-être que les quelques mots « J’ai regardé la cérémonie de lever du drapeau sur la place Tiananmen » peuvent résumer l’information dont nous avons besoin. En d’autres termes, il n’est pas nécessaire de le lire mot par mot, et vous pouvez déchiffrer toutes les informations en saisissant les points clés. Alors, peut-on en dire autant de la recherche scientifique ?
Inspirés par cela, les chercheurs ont combiné l’intelligence artificielle (IA) avec la technologie de microscopie pour former l’IA à identifier activement les caractéristiques clés des échantillons que les chercheurs doivent analyser. Contrairement à la numérisation complète des échantillons dans la technologie de microscopie traditionnelle, la méthode AI + microscopie a complètement changé la façon dont les chercheurs obtiennent des données d'échantillons, a considérablement accéléré le processus expérimental et a obtenu une « lecture rapide » au niveau microscopique.
Auteur | Ajouter zéro
Rédacteur | Xuecai, Sanyang
Le principe de la microscopie est de générer des signaux résolus spatialement en balayant des échantillons, de collecter les signaux pour analyse et ainsi de former une image de l'échantillon. Avec l'avancement continu de la technologie de l'instrumentation,La vitesse de balayage et la résolution des microscopes ont été considérablement améliorées.
Cependant, les expériences de microscopie à balayage à haute résolution présentent certains inconvénients importants :La quantité de données générées est énorme et la sonde cause de gros dommages à l’échantillon.Par exemple, une imagerie aux rayons X d'un volume de 1 mm^3 à une résolution d'environ 10 nm est réalisée.Les méthodes de numérisation traditionnelles produisent 10^15 voxels de données et nécessitent une dose de sonde assez importante.
Dans le même temps, la plupart des zones de l’échantillon ont une faible densité d’information et peuvent être directement ignorées.Au contraire, un petit nombre de domaines « limites, défauts, éléments spéciaux » contiennent des informations riches et doivent être étudiés en profondeur..
Pour localiser avec précision les zones clés à forte densité d'informations, les méthodes traditionnelles s'appuient principalement sur des opérateurs expérimentés pour analyser les données et guider le balayage des sondes, ce qui augmente considérablement la charge de travail et ralentit la progression de l'expérience.
Alors, est-il possible d’introduire l’IA pour aider les chercheurs à identifier les priorités de recherche dans les échantillons et à accélérer la collecte et l’analyse des données ?

Impression d'artiste des expériences de microscopie à balayage en champ sombre automatisées à l'APS.
À cette fin, les chercheurs du Laboratoire national d'Argonne aux États-Unis ont développé FAST (Fast Autonomous Scanning Toolkit), un kit de numérisation automatique rapide différent de la numérisation complète des échantillons de recherche au microscope traditionnel.FAST permet à l'IA d'identifier automatiquement l'emplacement numérisé, acquérant ainsi des informations sur l'échantillon de manière efficace et précise.Les résultats pertinents ont été publiés dans « Nature Communications ».

Les résultats connexes ont été publiés dans « Advanced Science »
Dans des simulations et des expériences de microscopie à rayons X en champ sombre, les chercheurs de FAST ont effectué des analyses FAST sur des films WSe2. Les résultats ont montré queUne analyse RAPIDE de <25% est suffisante pour imager et analyser avec précision l'échantillon.
Lien vers l'article :
https://www.nature.com/articles/s41467-023-40339-1
Procédures expérimentales
Données de formation:Utilisation d'un algorithme général d'entraînement d'image
L'algorithme utilisé par FAST n'a pas besoin d'être formé à l'aide de grands ensembles de données, et l'IA peut utiliser des images génériques pour identifier les zones d'intérêt.
Les données de formation ont été générées à partir d'images accessibles au public provenant des bibliothèques du MIT, de la base de données d'images USC-SIPI et du progiciel Scikit-image.

Exemples d'images de test utilisées
RAPIDE:Kit d'outils d'analyse autonome rapide
FAST signifie Fast Autonomous Scanning Toolkit.Il combine la méthode SLADS-Net, la technologie d'optimisation de chemin et un contrôle matériel efficace et modulaire.est une suite d'outils d'analyse autonomes rapides pour l'échantillonnage en temps réel et la sélection du chemin d'analyse dans la microscopie à balayage basée sur le synchrotron.

Flux de travail RAPIDE
UN:Un ensemble de mesures initiales quasi-aléatoires est transmis au dispositif périphérique, qui à son tour génère une estimation d'échantillon initiale, calcule les prochains points candidats à mesurer et calcule le chemin de mesure. Les nouvelles mesures sont combinées avec celles existantes pour calculer une nouvelle estimation, et le processus est répété jusqu'à ce que les critères d'achèvement soient atteints.
B:Au début de l'opération de point candidat, chaque point non mesuré est vérifié. PDans le voisinage local (avec un rayon de r), les points qui ont été mesurés sont mis en évidence, générant ainsi un vecteur de caractéristiques à 6 dimensions. Les vecteurs de caractéristiques ont été convertis en vecteurs à 50 dimensions à l'aide du noyau de fonction de base radiale (RBF) et utilisés comme entrée dans un réseau neuronal multicouche (NN). Ensuite, le réseau neuronal (NN) mesurera le point P Prédire l’amélioration attendue des images (ERD). Un groupe de pixels non mesurés avec l'ERD le plus élevé est sélectionné comme candidats pour la mesure suivante.
La formation FAST ne repose pas sur des données d’échantillon spécifiques.Il est possible de mesurer et de reconstruire dynamiquement un échantillon complexe (non binaire), ce qui est différent du flux de travail existant basé sur SLADS. De plus, son coût de calcul est négligeable par rapport au temps d'acquisition, même lorsqu'il est exécuté sur un dispositif de calcul de pointe à faible consommation (situé sur une ligne de faisceau synchrotron), ce qui constitue un avantage significatif pour les techniques expérimentales autonomes plus générales.
Ces caractéristiques permettent d'appliquer FAST à l'instrument de microscope à rayons X à balayage nanométrique de haute précision existant sur la ligne de faisceau de la nanosonde à rayons X durs APS.
Vérification des performances
RAPIDE:Mieux que les méthodes d'échantillonnage statique
Pour vérifier les performances de FAST, les chercheurs ont mené des expériences comparatives avec trois autres techniques d’échantillonnage statique.
Sujets:Autrement dit, l'ensemble de données de test est une image en champ sombre de 600 × 400 pixels, représentant 240 000 positions de mesure possibles, couvrant une zone physique de 900 × 600 μm et contenant plusieurs tranches WSe2 d'épaisseurs différentes ;
Méthode de comparaison :Les trois techniques d'échantillonnage statique sont l'échantillonnage par grille matricielle (RG), l'échantillonnage aléatoire uniforme (UR) et l'échantillonnage quasi-aléatoire à faible divergence (LDR).
Processus expérimental :À la même couverture de balayage, des images de reconstruction d'échantillonnage FAST, RG, UR et LDR sont générées.
Comparaison 1 :

Comparaison des images de reconstruction d'échantillonnage FAST et statique
UN:Image de vérité terrain, l'échelle de couleurs représente l'intensité normalisée
BD:Images de reconstruction RG, LDR et FAST sous couverture de balayage 10%
Les résultats montrent :L'échantillonnage FAST est capable de reproduire les limites, les bulles et les zones de transition entre différents niveaux d'épaisseur dans les objets expérimentaux avec une grande fidélité.
Comparaison 2 :

Comparaison des performances des méthodes d'échantillonnage FAST et statique à différentes couvertures de balayage
UN:L'erreur moyenne quadratique normalisée (NRMSE) varie en fonction de la couverture de l'analyse, et des valeurs inférieures indiquent de meilleures performances ;
B:La mesure de similarité structurelle (SSIM) varie en fonction de la couverture de l'analyse, et plus la valeur est élevée, meilleures sont les performances.
Les résultats montrent :FAST a une efficacité d'échantillonnage élevée et peut réaliser une reconstruction stable lorsque la couverture de numérisation atteint 27% ; les trois méthodes d’échantillonnage statique nécessitent plus de temps pour obtenir le même effet.
Comparaison trois:
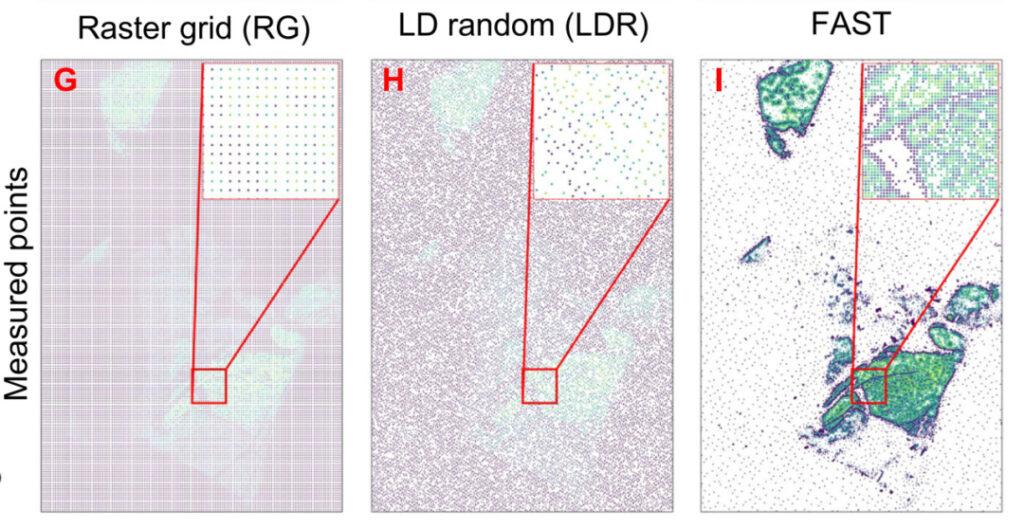
Positions de mesure réelles de FAST et deux méthodes d'échantillonnage statique à un taux de couverture de 10% dans les mêmes conditions d'échantillonnage
Les résultats montrent queLes structures reconstruites FAST présentent une grande similarité et une faible erreur.
À l’avenir, les résultats pourront être encore améliorés en utilisant des techniques de retouche plus sophistiquées dans la méthode FAST.
Les résultats de la comparaison des trois groupes ci-dessus montrent :
FAST surpasse les techniques d’échantillonnage statique. FAST échantillonne préférentiellement les zones présentant une hétérogénéité significative plutôt que les zones homogènes. Cela réduit considérablement le temps d’échantillonnage inefficace dans les zones vides et est particulièrement efficace pour les échantillons clairsemés.
RAPIDE : Capacité de reconstruction d'image en champ sombre efficace et précise
Dans les expériences sur la ligne de faisceau synchrotron, FAST a encore démontré ses excellentes performances.
Le processus expérimental a été entièrement piloté par l'IA, et les chercheurs n'ont interféré avec rien d'autre que le démarrage du script FAST. L'échantillon expérimental est un flocon de WSe2 déformé avec une résolution spatiale de 100 nm.

L'évolution de la numérisation FAST
AS:Images en champ sombre reconstruites FAST avec une couverture de balayage de 5%, 15% et 20% ;
B, D, F :Les points de mesure réels correspondants ;
G:Image obtenue par balayage point par point de la grille complète (couverture 100%) ;
AG:L'échelle de couleurs montre l'intensité normalisée ;
H:Seuls les points d'échantillonnage entre la couverture 15%-20% sont affichés.
La figure ci-dessus montre que dans le cas d'une faible couverture de balayage,La méthode FAST a identifié préférentiellement certaines régions hétérogènes.Comme le bord d’une bulle ; à mesure que la couverture de numérisation augmente progressivement, le résultat de la reconstruction devient de plus en plus clair et l'image reconstruite atteint une stabilité entre la couverture de numérisation de 15%-20%.
Couverture de numérisation 20%,FAST peut reproduire clairement et précisément toutes les caractéristiques principales de l'image numérisée complète.En même temps, cela permet d'économiser environ 80 minutes (≈65%) de temps d'expérience, améliorant considérablement l'efficacité expérimentale.
L'IA + l'avenir de la microscopie
L’avantage du procédé FAST réside non seulement dans l’amélioration de l’efficacité de l’acquisition de données microscopiques, mais également dans sa large gamme d’applications.Tao Zhou, un scientifique du Laboratoire national d'Argonne aux États-Unis, a déclaré : « Des microscopes à rayons X aux microscopes électroniques en passant par les microscopes à sonde atomique, cette technologie peut dynamiser toute recherche en microscopie nécessitant un balayage bidimensionnel. »
À l’avenir, la technologie de l’IA sera également appliquée plus en profondeur dans le domaine de la microscopie. Grâce à la formation, l’IA prend progressivement en charge des tâches telles que l’analyse et la reconnaissance automatisées d’images, l’amélioration et la reconstruction d’images, l’analyse quantitative et le diagnostic des maladies.
La technologie de l’IA et de la microscopie évolue vers un avenir plus clair, plus efficace et plus précis, et les limites de la recherche scientifique continueront de s’élargir.
Liens de référence :
[1]https://www.nature.com/articles/s41467-023-40339-1
[2]https://phys.org/news/2023-10-artificial-intelligence-scientists-self-driving-microscopy.html
[3]Une approche d'apprentissage supervisé pour l'échantillonnage dynamique








