Command Palette
Search for a command to run...
L'IA Combat Les Superbactéries : l'Université McMaster Utilise l'apprentissage Profond Pour Découvrir Un Nouvel Antibiotique, l'abaucine
Contenu en un coup d'œil :Acinetobacter baumannii est un pathogène Gram négatif courant contracté à l’hôpital qui présente souvent une résistance multimédicamenteuse. En utilisant les méthodes traditionnelles, il est difficile de découvrir de nouveaux antibiotiques qui inhibent cette bactérie. Mais l’utilisation de l’apprentissage automatique permet d’explorer rapidement l’espace chimique, augmentant ainsi la probabilité de découvrir de nouvelles molécules antimicrobiennes. Récemment, un résultat de recherche a été publié dans la revue internationale Nature Chemical Biology. Les chercheurs ont découvert que l’abaucine peut inhiber efficacement Acinetobacter baumannii.
Mots-clés:Apprentissage profond de l'abaucine d'Acinetobacter baumannii
Cet article a été publié pour la première fois sur la plateforme publique HyperAI WeChat~
Acinetobacter baumannii (nom scientifique : Acinetobacter baumannii, communément appelé : bactérie AB) est le bacille Gram négatif le plus courant du genre Acinetobacter. Il est largement présent dans l’eau et le sol dans la nature, ainsi que dans la peau humaine normale, les voies respiratoires, le tube digestif et le tractus urogénital. Cette bactérie est fréquemment présente dans les infections nosocomiales et est également un agent pathogène chez les animaux d’aquaculture.Provoque généralement une bactériémie, une pneumonie, une méningite, une péritonite, une endocardite et des infections des voies urinaires et de la peau.
Acinetobacter baumannii présente une forte résistance à l'humidité, à la chaleur, aux rayons ultraviolets, aux désinfectants chimiques et résiste aux basses températures. Il peut survivre à la surface d’objets secs pendant plus de 25 jours. Les désinfectants conventionnels ne peuvent qu’inhiber sa croissance mais ne peuvent pas le tuer. Il est également résistant au savon. Il s’agit du bacille Gram négatif le plus fréquemment isolé sur les mains du personnel médical, sur le matériel médical et sur les surfaces des objets.En raison de l’abus d’antibiotiques, Acinetobacter baumannii a développé une résistance multiple aux médicaments et est identifié par l’Organisation mondiale de la santé comme l’une des bactéries résistantes aux antibiotiques les plus dangereuses au monde.
Récemment, des chercheurs, dont Gary Liu de l’Université McMaster et Denise B. Catacutan du Massachusetts Institute of Technology, ont utilisé l’apprentissage profond pour examiner environ 7 500 molécules afin d’identifier de nouveaux antibiotiques qui inhibent Acinetobacter baumannii.Actuellement, la recherche a été publiée dans la revue Nature Chemical Biology sous le titre « Découverte guidée par l'apprentissage profond d'un antibiotique ciblant Acinetobacter baumannii ».

Les résultats de la recherche ont été publiés dans la revue Nature Chemical Biology
ArgumentAdresse:
https://www.nature.com/articles/s41589-023-01349-8#access-options
Procédures expérimentales
Ensemble de données
Les chercheurs ont examiné 7 684 petites molécules basées sur la croissance d'Acinetobacter baumannii ATCC 17978 dans le milieu LB.Les petites molécules comprenaient 2 341 médicaments génériques et 5 343 produits chimiques synthétiques, tous fournis par la bibliothèque de criblage à haut débit du Broad Institute. En utilisant le seuil de taux de réussite traditionnel (c'est-à-dire les molécules avec un taux d'inhibition de la croissance inférieur à un écart type par rapport à la moyenne de l'ensemble de données), ils ont identifié 480 molécules comme « actives » et 7 204 comme « inactives ».
Formation de modèle
Pour améliorer la structure du modèle, les chercheurs ont connecté la représentation moléculaire à 200 caractéristiques supplémentaires au niveau moléculaire calculées à l'aide de RDCit. De plus, pour améliorer encore les performances du modèle, les chercheurs ont utilisé un ensemble de 10 modèles pour construire et former le modèle sur un ensemble de données de formation de 7 684 molécules (avec ~ 6,2% d'exemples valides) et ont appliqué le modèle au Drug Repurposing Hub mis à jour.Les chercheurs ont divisé aléatoirement l'ensemble de données en 80% de données d'entraînement, 10% de données de validation et 10% de données de test.
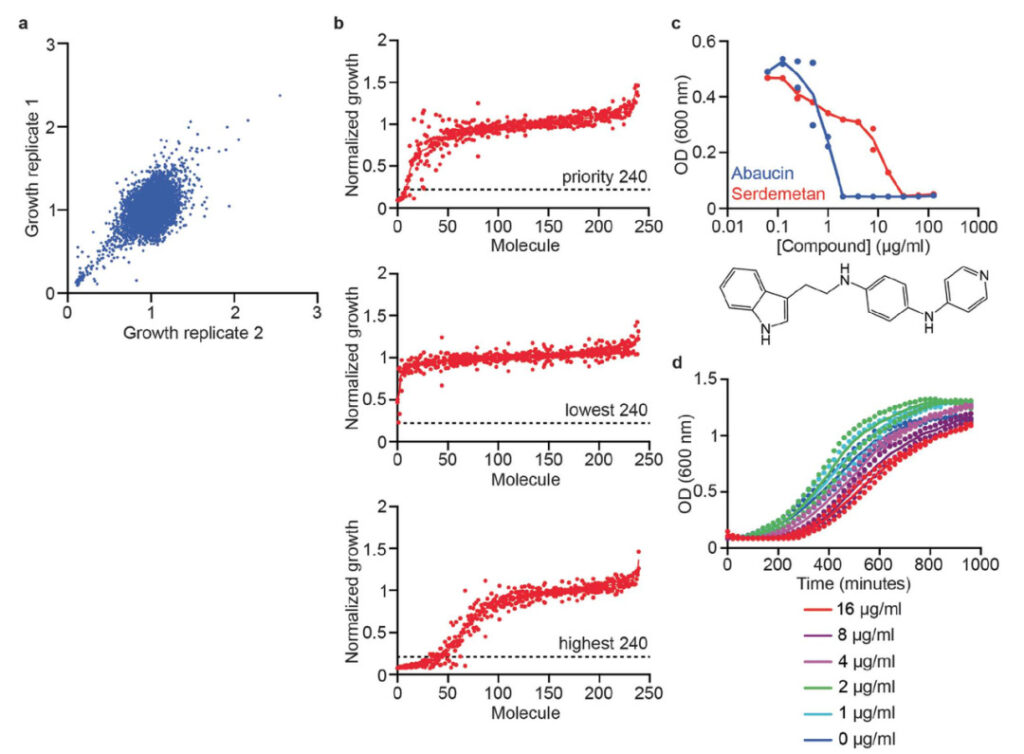
Figure 1 : Données d'entraînement du modèle
un. Données de criblage initiales pour 7 684 petites molécules qui inhibent la croissance d'Acinetobacter baumannii ATCC 17978 dans le milieu LB à 50 μM.
b. Données d'inhibition de la croissance classées de 240 molécules prioritaires sélectionnées pour validation à partir de l'ensemble de prédictions (en haut) ; données d'inhibition de la croissance classées de 240 molécules prédites avec les scores de prédiction les plus bas (milieu) ; données d'inhibition de la croissance classées de 240 molécules prédites avec les scores de prédiction les plus élevés qui n'ont pas été trouvés dans l'ensemble de données de formation (en bas). La ligne horizontale en pointillés représente le seuil de détection strict de l'inhibition de la croissance >80% à 50 μM.
c. La croissance d'Acinetobacter baumannii dans le milieu LB a été inhibée par l'abaucine (bleu) et le serdemetan (rouge).
d. Cinétique de croissance des cellules d'A. baumannii après traitement avec différentes concentrations d'abaucine pendant 6 h.
Acquisition de code :
https://github.com/chemprop/chemprop
Expériences sur des modèles de souris
Les chercheurs ont prétraité des souris femelles C57BL/6N âgées de 6 à 8 semaines avec respectivement 150 mg/kg et 100 mg/kg de cyclophosphamide, en fonction du poids corporel, quatre jours et un jour avant le début de l'expérience pour réduire le nombre de souris. Le jour 0, les souris ont été anesthésiées avec de l’isoflurane et ont reçu une injection intrapéritonéale de buprénorphine à une dose de 0,1 mg/kg comme analgésique. Les chercheurs ont utilisé environ 30 à 35 morceaux de ruban adhésif haute pression pour créer des abrasions de 2 cm2 sur le dos des souris, ont décollé le ruban jusqu'à la couche basale de l'épiderme, puis ont infecté les souris avec environ 6,5 x 106 UFC d'A. baumannii ATCC 17978.Les souris (n = 5/6) ont été traitées avec environ 10 à 20 μl de base Glaxal avec de l'abaucine (groupe expérimental) ou du DMSO (groupe témoin) 1, 2, 3, 4, 6, 10, 21 et 24 h après l'infection.
Résultats expérimentaux
Des chercheurs ont découvert un composé antibactérien appelé abaucine à l’aide de l’apprentissage automatique.Il a un spectre d'activité étroit contre Acinetobacter baumannii et peut interférer avec le processus de transport des lipoprotéines en interférant avec le mécanisme LolE. La figure ci-dessous montre le processus de découverte de l’abaucine guidée par l’apprentissage profond.
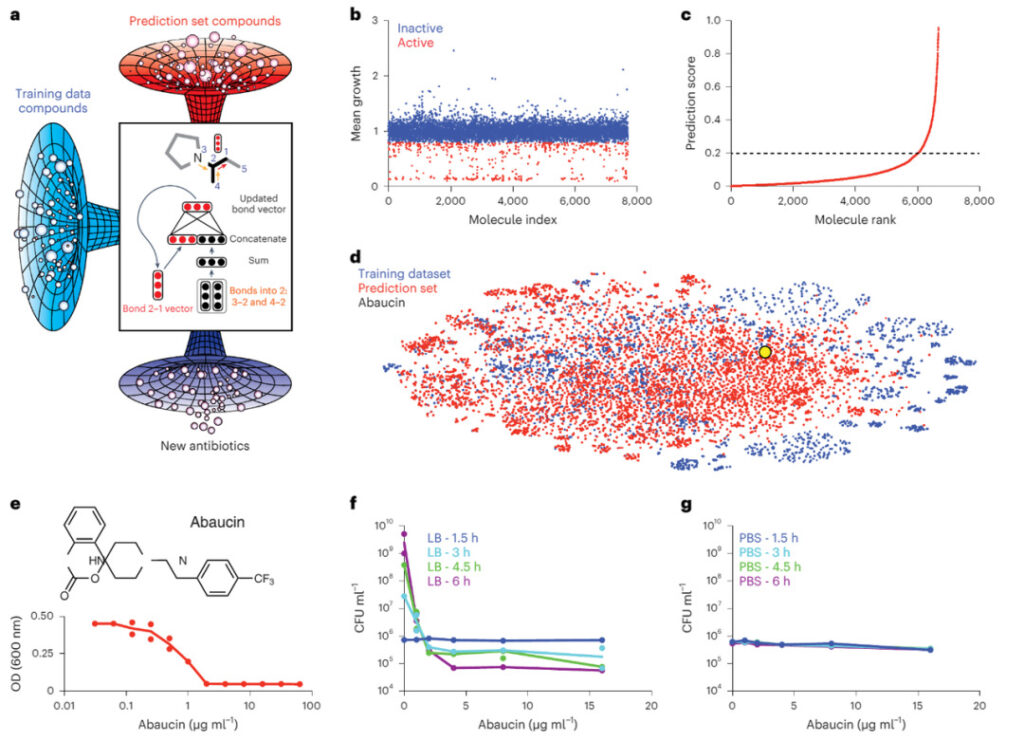
Figure 2 : L’apprentissage automatique a guidé la découverte de l’abaucine
un. ~7 500 molécules criblées (bleu) ; un réseau neuronal profond pour le transfert d'informations dirigé formé avec cet ensemble de données d'inhibition de la croissance et prédit dans le Drug Repurposing Hub (rouge) ; nouvelles molécules capables d'inhiber Acinetobacter baumannii (violet).
b. Inhibition de la croissance d'Acinetobacter baumannii ATCC 17978 à 50 μM par une collection de 7 684 petites molécules.
c. Le modèle formé classe les scores de prédiction des molécules dans le Drug Repurposing Hub. Les molécules avec des scores de prédiction supérieurs à 0,2 ont été considérées comme des molécules candidates préliminaires.
d. Un graphique t-SNE montre les relations chimiques entre l'ensemble de données d'entraînement (bleu), l'ensemble de prédiction (rouge) et l'abaucine (jaune).
f. Inhibition de la croissance d'Acinetobacter baumannii par l'abaucine dans le milieu LB.
f. Effet mortel de l'abaucine sur Acinetobacter baumannii à différentes concentrations après 1,5 h (bleu), 3 h (cyan), 4,5 h (vert) et 6 h (violet) d'incubation dans des conditions riches en nutriments.
g. Effet mortel de l'abaucine sur Acinetobacter baumannii après 1,5 h (bleu), 3 h (cyan), 4,5 h (vert) et 6 h (violet) d'incubation dans des conditions de carence en nutriments.
Comme le montre la figure 2, les chercheurs ont examiné environ 7 500 molécules, formé un réseau neuronal à l’aide de cet ensemble de données d’inhibition de la croissance et prédit de nouvelles molécules susceptibles d’inhiber l’activité d’Acinetobacter baumannii.Collectivement, ces données suggèrent que l’abaucine exerce son pouvoir antimicrobien en inhibant un processus biologique qui est le plus actif pendant la croissance et la division, ce qui est cohérent avec la plupart des antibiotiques connus.
De plus, pour tester l’efficacité in vivo de l’abaucine,Les chercheurs ont constaté des infections de plaies sur le dos des souris.
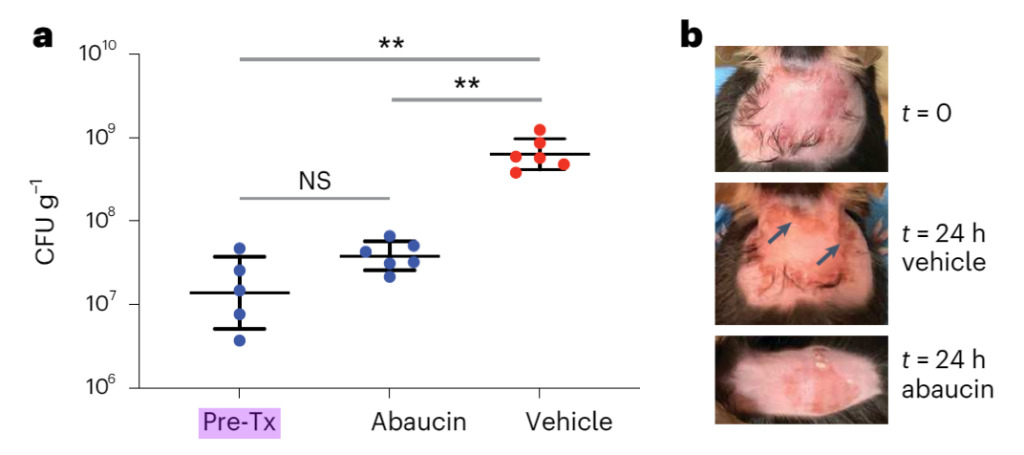
Figure 3 : L'abaucine peut inhiber l'infection à Acinetobacter baumannii dans un modèle de plaie
un. Dans le modèle d'infection de plaie dorsale, les souris ont été infectées par Acinetobacter baumannii ATCC 17978 (~6,5 x 106 UFC). Une heure après l’infection, les souris ont été traitées avec du DMSO (rouge ; n = 6) ou de l’abaucine 4% (bleu ; n = 6) pendant plus de 24 heures. L'axe vertical représente la charge bactérienne dans le tissu de la plaie 25 heures après l'infection. Pre-Tx représente la charge bactérienne au moment du traitement initial (n = 5). Les lignes noires représentent la moyenne ± écart type. NS indique non statistiquement significatif ; ** P < 0,005 en utilisant un test t bilatéral non apparié avec correction de Welch (pré-Tx vs. 4% abaucine, P = 0,0704 ; pré-Tx vs. DMSO, P = 0,0034 ; DMSO vs. 4% abaucine, P = 0,0039).
ps Généralement, un astérisque représente une valeur p inférieure à 0,05, deux astérisques représentent une valeur p inférieure à 0,01 et trois étoiles représentent une valeur p inférieure à 0,001. Plus la valeur p est petite, plus la signification est forte.
b. Images de la surface dorsale des souris avant l'infection (t = 0), après 24 h de traitement au DMSO et après 24 h de traitement à l'abaucine. L’inflammation observée dans le DMSO (flèches) était absente chez les souris traitées à l’abaucine.
La figure 3 montre que la charge bactérienne dans les plaies traitées avec l'abaucine était significativement inférieure à celle du groupe DMSO, et l'inflammation de la plaie qui était encore présente après 24 heures de traitement au DMSO a disparu dans l'abaucine.L'abaucine peut contrôler efficacement les infections causées par Acinetobacter baumannii.
En résumé, les chercheurs ont utilisé l’apprentissage profond pour découvrir que l’abaucine peut contrôler efficacement l’infection à Acinetobacter baumannii.Cette étude met en évidence le rôle de l’apprentissage automatique dans la découverte de nouveaux antibiotiques et dans l’orientation de l’étude des agents pathogènes Gram-négatifs difficiles.
L'IA change la manière traditionnelle de découvrir de nouveaux médicaments
Il convient de noter que cette recherche a été menée par Jon Stokes, professeur adjoint à l’Université McMaster, James J. Collins, professeur de génie médical et de sciences au MIT, et les étudiants diplômés de l’Université McMaster Gary Liu et Denise Catacutan. Jon Stokes, Gary Liu et Denise Catacutan sont tous membres du laboratoire Stokes.

Adresse du laboratoire Stokes :
https://www.thestokeslab.com/projects
Le laboratoire découvre de nouveaux antibiotiques en utilisant des approches de biologie à haut débit de pointe et d'intelligence artificielle contemporaine.Plus précisément, les chercheurs ont formé des modèles d’apprentissage profond pour prédire de nouveaux antibiotiques à partir d’une grande base de données de simulation informatique, et ont également construit des modèles pour prédire le mécanisme d’action et les propriétés in vivo de nouveaux antibiotiques. Ils utilisent l’apprentissage automatique pour accélérer la découverte de nouveaux antibiotiques tout en réduisant les coûts de recherche et développement.
Le professeur Stokes a déclaré un jour dans une interview que « l’intelligence artificielle changera fondamentalement la façon dont les nouveaux médicaments sont découverts ».Les résultats suggèrent que l’intelligence artificielle a des implications importantes pour la découverte de nouveaux antibiotiques contre une variété de pathogènes difficiles, et que cette approche a également du potentiel dans la recherche d’autres thérapies antimicrobiennes. Bien que l’intelligence artificielle ne puisse pas résoudre tous les problèmes, en tant qu’outil puissant, elle peut nous aider à trouver de nouveaux médicaments et apporter de l’espoir aux gens.
Articles de référence :
[1]http://ccm.dxy.cn/article/805579
[2]https://zh.wikipedia.org/wiki/
[3]https://www.ctvnews.ca/health/scientists-use-ai-to-discover-antibiotic-for-very-difficult-to-treat-bacteria-1.6411927
-- sur--








